Interruptores moleculares y métodos para su uso.
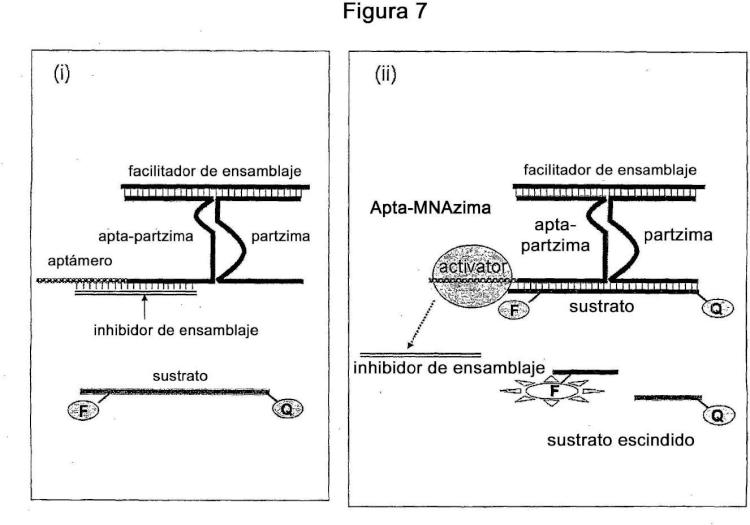
Una proencima inactiva de ácido nucleico multi-componente (MNAi) que comprende dos o más componentes oligonucleótidos y por lo menos una molécula inhibidora de la actividad,
en la que por lo menos un primer componente del oligonucleótido y un segundo componente del oligonucleótido son capaces de autoensamblarse en presencia de por lo menos un inhibidor de la activida de complejo de MNA donde cada uno de dichos componentes primero y segundo de oligonucleótido comprenden una porción de brazo se sustrato, una porción de núcleo catalítico y una porción de brazo sensor;
Donde a continuación del auto-ensamblaje, la porción del brazo sensor de dichos primer y segundo componentes oligonucleótidos actúan como brazos sensores, la porción de brazo de sustrato del primer y segundo componente oligonucleótido actúan como brazos de sustrato y la porción de núcleo catalítico de los componentes primero y segundo oligonucleótido del núcleo catalítico no-funcional; y
Donde a continuación del auto-ensamblaje por lo menos uno de los brazos sensores interactúa con dicho inhibidor de la actividad y dichos primer y segundo componentes oligonucleótidos se mantienen cerca por la asociación de sus respectivas porciones del núcleo catalítico para formar un núcleo catalítico no funcional.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/AU2007/001517.
Solicitante: SpeeDx Pty Ltd.
Nacionalidad solicitante: Australia.
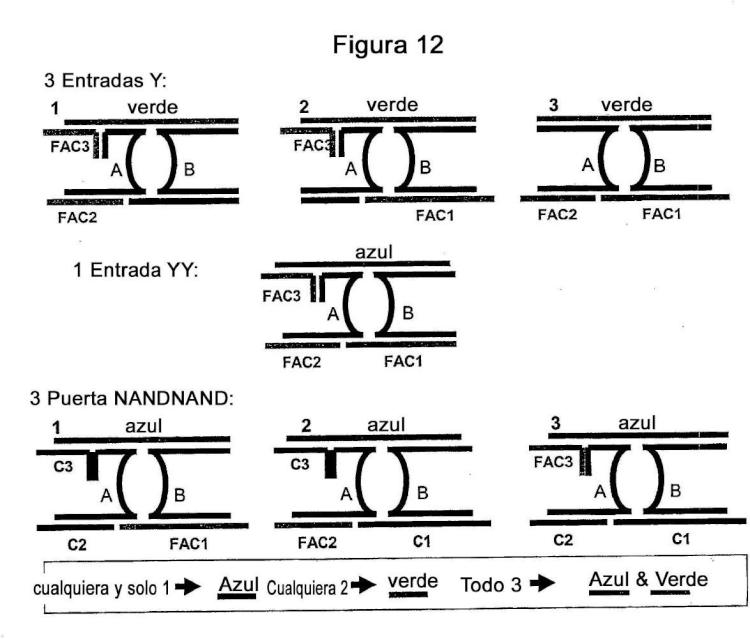
Dirección: Suite G16, National Innovation Centre, Australian Technology Park, 4 Cornwallis St. Eveleigh, NSW 2015 AUSTRALIA.
Inventor/es: TODD, ALISON, VELYIAN, MOKANY,Elisa, BIRKETT,Donald,John, DOAN,Tram,Bich, REID,CHRISTOPHER ROLAND.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/70 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen virus o bacteriófagos.
PDF original: ES-2520023_T3.pdf
Fragmento de la descripción:
Interruptores moleculares y métodos para su uso Descripción Ã?MBITO TÉCNICO
El presente invento trata de compuestos y métodos que permiten la manipulación de la actividad catalítica de los complejos de ácido nucleico multi-componente (MNA) . Además, se proporcionan métodos que usan estos compuestos y métodos para crear sensores moleculares, interruptores moleculares, y/o moduladores o propagadores de cascadas autocatalíticas autorreplicativas y otros procesos iterativos. Más en concreto, el invento trata de compuestos que permiten el auto-ensamblaje de complejos de ácido nucleico multicomponente inactivos y los métodos para su uso.
Contexto del invento [0002] Además de sus funciones evolutivas optimizadas, las extraordinarias propiedades físicas y funcionales de los ácidos nucleicos proporcionan la oportunidad de un sinfín de dispositivos y métodos bio-moleculares nuevos. Se ha contemplado el diseño de ácidos nucleicos para usos terapéuticos, biosensores, instrumentos a escala nano y herramientas para el cálculo molecular. Los métodos utilizan las características del auto-ensamblaje del ADN, la electro conductividad, elementos de información, amplificación, interruptores, detección molecular y actividad catalítica. Además, teniendo en cuenta que el ADN es sólido, estable y termoestable, proporciona un material perfecto para la ingeniería molecular de instrumentos mecánicos o informáticos.
Los ácidos nucleicos monocatenarios, como el ADN y el ARN, tienen la capacidad de multiplicarse en estructuras tridimensionales complejas que pueden funcionar como receptores altamente específicos (aptámeros) y catalizadores (ribozimas y and DNAzimas) . Además, la necesaria complementariedad entre cadenas de ácido nucleico para la hibridación conforma la base de una amplia gama de técnicas, que permiten la detección de objetivos (como el análisis de micromatriz, Northern blot o Southern blot) , y/o amplificación del objetivo (por ejemplo, reacción en cadena de la polimerasa) . Además, la hibridación proporciona la base para la construcción a nano escala de ácido nucleico y para estrategias computacionales basadas en el ADN.
La auto-replicación es un proceso por el cual los elementos pueden duplicarse (copiarse) a sí mismos. En dichos procesos, los productos de cada una de las reacciones dirigen la formación de las nuevas copias (replicones) del elemento a partir de las partes de los compuestos. Se ha desarrollado una amplia variedad de técnicas para la auto-replicación de las secuencias de ácido nucleico.
Los métodos para la réplica in vitro de las secuencias diana de ácido nucleico (amplificación de la diana) son muy conocidos. Muchos de estos métodos necesitan cebadores oligonucleótidos, capaces de hibridación específica con el ADN o ARN diana, que puede extenderse por la polimerasa de ADN o ARN para crear una nueva copia de la diana (un amplicón) , usando la diana como modelo para la síntesis directa. Dichas técnicas (revisadas por Schweitzer y Kingsmore, 2001) incluyen la reacción en cadena de polimerasa, amplificación del desplazamiento de cadena, amplificación de círculo rodante y amplificación isotérmica mediada por bucle, amplificación mediada por transcripción, auto-replicación de secuencia y amplificación basada en la replicación de secuencia de ácido nucleico. Un enfoque alternativo, conocido como reacción en cadena de la ligasa ("RCL") usa una ligasa de proteína para amplificar las dianas de ácido nucleico. La reacción depende de la capacidad de los productos de ligación de cada tanda para servir como modelos para dirigir la ligación de nuevas copias de la diana (Barany, 1991) .
Las tecnologías de amplificación de la diana, como las que se han indicado anteriormente, se han usado con frecuencia en investigación y/o en diagnóstico clínico. De todas formas, a pesar de su valor, cada una tiene sus desventajas inherentes. Todas ellas necesitan el uso de enzimas de proteína (por ejemplo, polimerasa de ADN, polimerasa de ARN, transcriptasa inversa, y o ligasa) . La inclusión de enzimas de proteína aumenta la complejidad y el coste de la fabricación del reactivo y disminuye el período de conservación de los kits que contienen reactivos. Otros retos técnicos asociados son la contaminación por replicones (amplicones de la diana) de reacciones previas que llevan a indicar falsos positivos y/o señales de fondo motivadas por la réplica de secuencia de cebadores (cebadores-dímeros) o un fondo provocado por la ligación independiente de diana.
En los últimos 20 años se ha descubierto una amplia variedad de moléculas de ácido nucleico, con actividad enzimática o catalítica. Las enzimas de ARN ("ribozimas") se dan en la naturaleza pero pueden manipularse para que de forma específica reconozcan y modifiquen un substrato diana de ARN (Haseloff y Gerlach, 1988) . Las técnicas de evolución in vitro han facilitado el descubrimiento y el desarrollo de muchos otros ácidos nucleicos catalíticos, incluidos los ácidos desoxirribonucleicos, a los que con frecuencia se hace referencia como "enzimas de ADN " o "DNAzimas" (revisado por Emilsson y Breaker, 2002) . Las DNAzimas y/o ribozimas con evolución in vitro se ha descubierto que tienen la capacidad de catalizar una amplia gama de reacciones, entre las que se incluye la división de ácidos nucleicos (Carmi et al., 1996; Raillard y Joyce, 1996; Breaker, 1997; Santoro y Joyce, 1998) , ligación de ácidos nucleicos (Cuenoud y Szostak, 1995, Prior et al., 2004) , metalización de porfirina (Li y Sen, 1996) , y la formación de cadenas de carbono-carbono (Tarasow et al., 1997) , uniones de éster (Illangasekare et al., 1995)
o uniones de amida (Lohse y Szostak, 1996) .
En concreto, las DNAzimas y las ribozimas se han caracterizado por específicamente dividir secuencias diferenciadas de ácido nucleico después de la hibridación a través de emparejamiento de bases de Watson Crick. Las DNAzimas son capaces de dividir tanto moléculas de ARN (Breaker y Joyce, 1994; Santoro y Joyce, 1997) como de ADN (Carmi et al., 1996) . Las ribozimas también son capaces de dividir secuencias diana tanto de ARN (Haseloff y Gerlach, 1988) como de ADN (Raillard y Joyce, 1996) . La tasa de división catalítica de muchas enzimas de ácido nucleico es dependiente de la presencia y el nivel de concentración de iones de metales divalentes como Ba2+, Sr2+, Mg2+, Ca2+, Ni2+, Co2+, Mn2+, Zn2+ y Pb2+ (Santoro y Joyce, 1998; Brown et al., 2003) .
Las DNAzimas "10:23" y "8:17" son capaces de dividir substratos de ácido nucleico en uniones de fosfodiéster de un ARN específico para crear un producto de reacción que tenga grupos de 2â?, 3â?-fosfato cíclico y 5â?hidroxil (Santoro y Joyce, 1997; revisado por Emilsson y Breaker, 2002) . Entre los ejemplos de desoxirribozimas (DNAzimas) , que pueden ligar productos de 2â?, 3â?-fosfato cíclico and 5â?-hidroxil se incluyen las ligas "7Z81" y "7Z48" (Prior, 2004) .
Hay varios ácidos nucleicos catalíticos, entre los que se incluye la ribozima en cabeza de martillo, las DNAzimas 10:23 y 8:17 y las ligasas "7Z81" y "7Z48" que tienen estructuras básicas semejantes con dominios múltiples. Estas enzimas de ácido nucleico tienen un dominio catalítico conservado (núcleo catalítico) flanqueado por dos dominios con de unión de substrato no conservados ("brazos") , que de forma específica reconocen y se hibridan al substrato. Mientras estas enzimas de ácido nucleico pueden funcionar como auténticas enzimas múltiples de transporte, cada una de las enzimas solo tiene capacidad para reconocer una molécula, en concreto el substrato que entonces puede modificar catalíticamente.
Se ha demostrado que los ácidos nucleicos catalíticos solo toleran ciertas modificaciones en el área que forma el núcleo catalítico (Perreault et al., 1990; Perreault et al., 1991; Zaborowska et al., 2002; Cruz et al., 2004; Silverman, 2004) ) . Dependiendo de la dureza de las condiciones de la reacción, podría tolerarse un cierto grado de desajuste dentro de los brazos del substrato. De todas formas, los requisitos para el emparejamiento de base Watson Crick es lo suficientemente estricto como para permitir el desarrollo de protocolos que usen ácidos nucleicos catalíticos para facilitar la discriminación de secuencias estrechamente relacionadas (Cairns et al., 2000) (WO 99/50452) .
Los "aptámeros" son secuencias de ADN, ARN o péptidos que tienen capacidad de reconocer uno o más ligandos con gran afinidad y especificidad debido a su estructura de alto nivel. Por ejemplo un dominio o bolsa unido por 3-D. Muchos aptámeros han sido evolucionados in vitro por su capacidad de unirse a ligandos, entre ellos, por ejemplo, los ácidos nucleicos, las proteínas,... [Seguir leyendo]
Reivindicaciones:
1. Una proencima inactiva de ácido nucleico multi-componente (MNAi) que comprende dos o más componentes oligonucleótidos y por lo menos una molécula inhibidora de la actividad, en la que por lo menos un primer
componente del oligonucleótido y un segundo componente del oligonucleótido son capaces de autoensamblarse en presencia de por lo menos un inhibidor de la activida de complejo de MNA donde cada uno de dichos componentes primero y segundo de oligonucleótido comprenden una porción de brazo se sustrato, una porción de núcleo catalítico y una porción de brazo sensor; Donde a continuación del auto-ensamblaje, la porción del brazo sensor de dichos primer y segundo componentes oligonucleótidos actúan como brazos sensores, la porción de brazo de sustrato del primer y segundo componente oligonucleótido actúan como brazos de sustrato y la porción de núcleo catalítico de los componentes primero y segundo oligonucleótido del núcleo catalítico no-funcional; y Donde a continuación del auto-ensamblaje por lo menos uno de los brazos sensores interactúa con dicho inhibidor de la actividad y dichos primer y segundo componentes oligonucleótidos se mantienen cerca por la
asociación de sus respectivas porciones del núcleo catalítico para formar un núcleo catalítico no funcional.
2. La MNAi de la reivindicación 1, cuando el inhibidor de la actividad sea un sustrato.
3. La MNAi de la reivindicación 1 o de la reivindicación 2, cuando por lo menos un componente de la MNAi comprenda un aptámero o porción suya donde dicho aptámero o porción suya una un ligando elegido del grupo que comprende ácidos nucleicos, proteínas, glicoproteínas, lípidos, lipoproteínas, células, virus, bacterias, arqueas, hongos, anticuerpos, metabolitos, patógenos, toxinas, contaminantes, venenos, pequeñas moléculas, polímeros, iones metálicos, sales de metales, priones o cualquier derivado, porción o combinaciones de los mismos.
4. El uso de la MNAi de la reivindicación 1 como interruptor molecular, donde dichas transiciones de MNAi transiciona de un complejo inactivo a uno activo de MNA en respuesta a un evento de entrada.
5. El uso de la reivindicación 4 cuando el evento de entrada se seleccione del grupo que comprende cambios e la temperatura, concentración de sal, fuerza iónica, pH, presencia o ausencia de catión divalente, tipo o concentración, carga eléctrica, carga magnética, manipulación física y cambios en la concentración de una MNA o componentes de modulador o componentes del microambiente o cualquier combinación de los mismos.
6. El uso de las reivindicaciones 4 o 5 siempre que la transición tenga como resultado un cambio en la señal de35 salida.
7. El uso de la reivindicación 6 siempre que se amplifique la señal de salida.
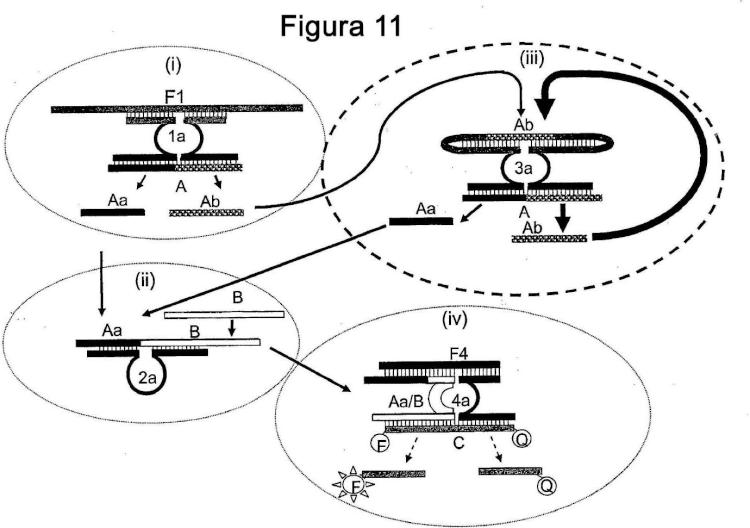
8. Un método de detección de diana usando cascada, cuando dicha cascada comprenda una MNAzima inicial que se forma en presencia de dicha diana; una primera MNAzima formada en presencia de un producto de dicha MNAzima inicial; y una MNAzima adicional que se ha formado en presencia de un producto de dicha primera MNAzima; donde dicho método comprenda los siguientes pasos:
(i) Modificación de un primer sustrato con dicha MNAzima inicial para generar un primer facilitador del 45 ensamblaje;
(ii) Ensamblaje de dicha primera MNAzima con dicho primer facilitador del ensamblaje, (iii) Modificación de un sustrato adicional con dicha primera MNAzima para generar un facilitador del ensamblaje adicional;
(iv) Ensamblaje de dicha MNAzima adicional con dicho facilitador del ensamblaje adicional;
(v) Modificación de dicho primer sustrato con dicha MNAzima adicional para generar dicho primer facilitador del ensamblaje;
(vi) Ensamblaje de dicha primera MNAzima con dicho primer facilitador del ensamblaje liberado de (v) , formando así una cascada de amplificación
Donde dicha modificación de como mínimo uno de dichos sustratos primero o adicional produzca un efecto detectable indicativo de dicha diana.
9. El método de la reivindicación 8 donde dicha MNAzima primera o adicional comprenda dos partzimas que se vuelven activas catalíticamente cuando están en presencia de ocmo mínimo dos componentes facilitadores del ensamblaje.
10. El método de la reivindicación 8 o de la reivindicación 9 donde dicho efecto detectable es detectado, como mínimo, por uno de los siguientes métodos: espectroscopio de fluorescencia, resonancia plasmónica de superficie, espectrocopio de masa, NMR, resonancia de espín electrónico, espectroscopio de fluorescencia de
polarización, dicroísmo circular, immunoanálisis, cromatografía, radiometría, fotometría, escintigrafía, métodos electrónicos, UV, espectroscopio de luz visible o infra roja, métodos enzimáticos o cualquier combinación de 63
ellos.
11. Un método para la detección de un facilitador del ensamblaje usando una cascada de señal que comprende
una primera MNAzima, un complejo de MNA inicialmente presente en una forma sustancialmente catalíticamente inactiva (MNAi) , un inhibidor de la actividad capaz de ser modificado por dicha primera
MNAzima para proporcionar un efecto detectable, donde dicho inhibidor de la actividad es tanto un inhibidor de
la actividad como un sustrato potencial y
Donde la asociación de dicho facilitador del ensamblaje con partzimas para dicha primera MNAzima en
condiciones que permitan la actividad catalítica de dicha primera MNAzima facilite la actividad catalítica de
dicha primera MNAzima proporcionando así la modificación de dicho inhibidor de la actividad para liberar un
dominio facilitador del activador del ensamblaje y un dominio inhibidor de la actividad de dicho inhibidor de la
actividad y donde dicha liberación proporcione dicho efecto detectable y
Donde dicho dominio facilitador del activador del ensamblaje, liberado, facilite el ensamblaje de una segunda
MNAzima de los componentes de dicho complejo de MNA y Donde la actividad catalítica de dicha segunda MNAzima modifique dicho inhibidor de la actividad para liberar
más dominios inhibidores de la actividad y más dominios facilitadores del activador del ensamblaje y donde
dicha liberación proporcione un mayor efecto detectable y;
Donde dichos otros dominios facilitadores del activador del ensamblaje faciliten el ensamblaje de las segundas
MNAzimas adicionales, proporcionando así dichas segundas MNAzimas activas catalíticamente,
proporcionando así un mayor efecto detectable, indicativo de la presencia de dicho facilitador del ensamblaje y
Donde el ensamblaje de las MNAzimas activas catalíticamente se regula por medio de un evento de entrada
seleccionado de entre los siguientes: cambio de la temperatura, concentración de sal, fuerza iónica, pH,
presencia o ausencia de catión divalente, tipo o concentración, carga eléctrica, carga magnética, manipulación
física y cambio en la concentración de una MNA o de componente modulador o de componente del microambiente o cualquier combinación de los anteriores.
Patentes similares o relacionadas:
Señal para el empaquetamiento de vectores del virus de la gripe, del 24 de Junio de 2020, de WISCONSIN ALUMNI RESEARCH FOUNDATION: Un vector del virus de la gripe para la expresión y empaquetamiento de ARNv recombinante, en el que el vector comprende: secuencias correspondientes […]
Métodos y sistemas para la amplificación de ácidos nucleicos, del 13 de Mayo de 2020, de Coyote Bioscience Co., Ltd: Un método para amplificar una secuencia de un ácido nucleico diana presente en una muestra biológica obtenida de un sujeto, que comprende: (a) proporcionar un recipiente de […]
Métodos para detectar un contaminante biológico, del 29 de Abril de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para detectar un contaminante biológico en una muestra de ensayo que comprende las etapas de: a) combinar ingredientes para preparar una mezcla […]
Procedimiento de prueba de la carga viral del VIH-1 automatizada para gotas secas, del 15 de Abril de 2020, de Abbott Molecular Inc: Un método automatizado para detectar ácidos nucleicos del VIH-1 en una muestra de sangre, comprendiendo el método: a) proporcionar: i) una muestra de sangre que […]
Métodos para diagnosticar enfermedades infecciosas usando medios de adsorción, del 15 de Abril de 2020, de ExThera Medical Corporation: Un método in vitro para concentrar patógenos infecciosos presentes en una muestra biológica obtenida de un sujeto que es sospechoso de estar […]
Métodos de discriminación entre el VIH-1 y vectores de lentivíricos, del 11 de Marzo de 2020, de Calimmune Inc: Un método de detección de una cantidad de un ácido nucleico lentivírico en una muestra, donde el ácido nucleico lentivírico comprende una deleción […]
Coronavirus de bovino atenuado y vacunas relacionadas, del 11 de Marzo de 2020, de INTERVET INTERNATIONAL B.V: Un coronavirus de bovino atenuado (BCoV) que codifica uno o más de los siguientes: - una proteína espicular que comprende la secuencia de aminoácidos de SEQ ID NO: 2, […]
Viriones de virus adenoasociados con cápside variante y métodos de uso de los mismos, del 19 de Febrero de 2020, de THE REGENTS OF THE UNIVERSITY OF CALIFORNIA: Un virión de virus adenoasociado recombinante (VAAr), o una composición farmacéutica que comprende dicho virión, para su uso en un método de tratamiento […]