SUSTRATO PARA LA ACTIVIDAD ENZIMÁTICA DE RPN 11.
Péptido que comprende una secuencia de aminoácidos al menos idéntica en un 70% a la SEQ ID NO:
1 y al menos un resto de ubiquitina que es al menos idéntico en un 90% a la secuencia de los primeros 76 aminoácidos de la SEQ ID NO: 2, en el que el péptido es un sustrato para una metaloproteasa Rpn11 que comprende un motivo JAMM
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2006/021080.
Solicitante: PROTEOLIX, INC.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 230 EAST GRAND AVENUE, SUITE A SOUTH SAN FRANCISCO CA 94080 ESTADOS UNIDOS DE AMERICA.
Inventor/es: BENNETT,MARK,K, PARLATI,FRANCESCO, AUJAY,MONETTE.
Fecha de Publicación: .
Fecha Solicitud PCT: 30 de Mayo de 2006.
Fecha Concesión Europea: 6 de Octubre de 2010.
Clasificación PCT:
- A61K38/17 NECESIDADES CORRIENTES DE LA VIDA. › A61 CIENCIAS MEDICAS O VETERINARIAS; HIGIENE. › A61K PREPARACIONES DE USO MEDICO, DENTAL O PARA EL ASEO (dispositivos o métodos especialmente concebidos para conferir a los productos farmacéuticos una forma física o de administración particular A61J 3/00; aspectos químicos o utilización de substancias químicas para, la desodorización del aire, la desinfección o la esterilización, vendas, apósitos, almohadillas absorbentes o de los artículos para su realización A61L; composiciones a base de jabón C11D). › A61K 38/00 Preparaciones medicinales que contienen péptidos (péptidos que contienen ciclos beta-lactama A61K 31/00; dipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina 2,5-dionas, A61K 31/00; péptidos basados en la ergolina A61K 31/48; que contienen compuestos macromoleculares que tienen unidades aminoácido repartidas estadísticamente A61K 31/74; preparaciones medicinales que contienen antígenos o anticuerpos A61K 39/00; preparaciones medicinales caracterizadas por los ingredientes no activos, p. ej. péptidos como soportes de fármacos, A61K 47/00). › que provienen de animales; que provienen de humanos.
- C07K14/00 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados.
- C07K19/00 C07K […] › Péptidos híbridos (Inmoglobulinas híbridas compuestas solamente de inmoglobulinas C07K 16/46).
- G01N33/68 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › en los que intervienen proteínas, péptidos o aminoácidos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia.
PDF original: ES-2356770_T3.pdf






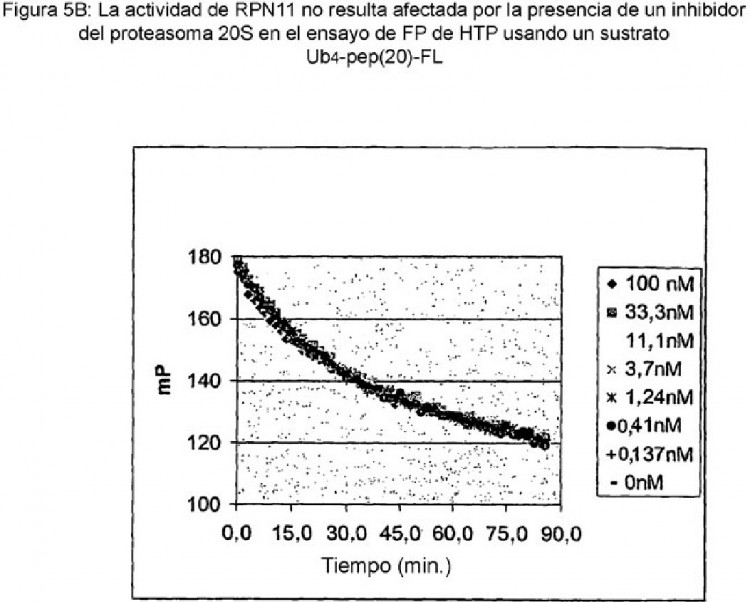
Fragmento de la descripción:
Antecedentes
La proteólisis por el proteasoma 26S avanza mediante la unión de una proteína sustrato ubiquitinada a la partícula reguladora 19S, seguido por su desubiquitinación, desplegado y translocación a la luz del núcleo 20S, donde se degrada. En los últimos años, se ha hecho evidente que el proteasoma es una diana atractiva para la intervención terapéutica en cáncer, trastornos relacionados con el sistema inmunitario, inflamación, estados isquémicos, trastornos neurodegenerativos y otras enfermedades. Hasta la fecha, el único inhibidor del proteasoma aprobado por la FDA (VELCADE™) funciona inhibiendo la actividad de las peptidasas del núcleo 20S. No obstante, la inhibición de otras actividades enzimáticas que residen dentro del complejo de proteasoma podría servir como un medio igualmente eficaz, si no mejor, de controlar la función del proteasoma.
Un candidato de diana es la metaloproteasa, Rpn11, que reside dentro de la partícula reguladora 19S. Es responsable de la desubiquitinación inicial de las proteínas diana (Eytan, et al. JBC 268(7):46684674 (1993); Verma, et al. Science 298:611-615 (2002)). El homólogo humano de esta proteína de levadura es POH1. Hasta la fecha, no se ha generado una forma recombinante de Rpn11. Sin embargo, la actividad desubiquitinante asociada con Rpn11 puede distinguirse en el contexto de un complejo de proteasoma 26S purificado por su sensibilidad a ATP-S y 1,10-fenantrolina (OPA) y su insensibilidad a inhibidores de proteasa del núcleo 20S y los inhibidores de DUB clásicos (ubiquitina aldehído (UB-Al) o ubiquitina vinilsulfona (UbVS)).
En la carrera por aprender más acerca de Rpn11 y su actividad desubiquitinante, se han empleado varios ensayos. La mayoría utilizan extractos del proteasoma 26S purificado como fuente de la enzima y observan la degradación en presencia o ausencia de un inhibidor de DUB. La detección de la actividad de Rpn11 requirió una proteína ubiquitinada tal como Ub-sic 1 (Verma et al., citado anteriormente) y la reacción se monitorizó mediante SDS-PAGE seguido por inmunotransferencia o bien con -ubiquitina (-Ub) o bien con -proteína tal como -sic 1. Los sustratos que estaban radiomarcados
o marcados de manera fluorescente todavía requerían una etapa que separaba el sustrato no escindido del producto escindido, tal como PAGE (Eytan et al., citado anteriormente) o precipitación con TCA (Yao et al., Nature 419: 403-407 (2002)), respectivamente.
Por tanto, es altamente deseable un ensayo de alto rendimiento (HTP) que emplee proteínas sustrato de Rpn11 novedosas con el fin de seleccionar grandes números de moléculas para identificar moduladores de la actividad de Rpn11.
Breve descripción de la solicitud
En consecuencia, esta invención proporciona péptidos que son sustratos de la actividad enzimática del proteasoma (por ejemplo, Rpn11) y diversos métodos que emplean los sustratos peptídicos, tales como ensayos que miden las actividades enzimáticas del proteasoma (por ejemplo, Rpn11) que seleccionan agentes que modulan las actividades enzimáticas del proteasoma, definiéndose dichos péptidos y métodos en las reivindicaciones. Los términos “péptido”, “polipéptido” y “proteínas” se usan en el presente documento de manera intercambiable. El término “proteasoma” se refiere al proteasoma 26S, a la partícula reguladora 19S, a un complejo proteico que comprende la partícula reguladora 19S, o a otros complejos proteicos o componentes que comprenden una Rpn11 o una proteína de tipo Rpn11 implicada en la proteólisis mediada por la ubiquitina. El término “Rpn11” también se usa en el presente documento de manera intercambiable con “POH1” y se refiere a cualquier polipéptido que confiera una actividad enzimática de Rpn11, por ejemplo, la actividad de desubiquitinación.
Un primer aspecto de la invención proporciona un sustrato peptídico para una proteasa del proteasoma 26S, el proteasoma 20S, o la partícula reguladora 19S. El sustrato peptídico comprende una secuencia de aminoácidos al menos idéntica en un 70%, 80%, 90%, 95% o 100% a la SEQ ID NO: 1 y un resto de ubiquitina que es al menos idéntico en un 90%, 95% o 100% a la secuencia de los primeros 76 aminoácidos de la SEQ ID NO: 2. En ciertas realizaciones, el sustrato peptídico comprende un resto de ubiquitina que es al menos idéntico en un 90%, 95% o 100% a la secuencia de los primeros 76 aminoácidos de la SEQ ID NO: 2, una primera secuencia de aminoácidos al menos idéntica en un 70%, 80%, 90%, 95% o 100% a la SEQ ID NO: 1 y una secuencia de aminoácidos adicional, que comienza en el extremo C-terminal de la primera secuencia de aminoácidos, que tiene al menos 3, 5, 8, 10, 12, 15, 18, 20, 25, 30, 40, 50, 75, 100, 200, 300, 500 o más aminoácidos.
También se describen sustratos peptídicos que comprenden una secuencia de aminoácidos al menos idéntica en un 50%, 60%, 70%, 80%, 90%, 95% o 100% a cualquiera de la SEQ ID NO: 3-15 y un resto de ubiquitina que es al menos idéntica en un 70%, 80%, 90%, 95% o 100% a la secuencia de los primeros 76 aminoácidos de la SEQ ID NO: 2.
En ciertas realizaciones, el sustrato peptídico comprende además uno o más restos de ubiquitina en el extremo N-terminal del péptido. En ciertas realizaciones, uno o más restos de ubiquitina pueden estar unidos al péptido a través de la(s) K(s) del péptido.
En ciertas realizaciones, un resto de ubiquitina comprende una secuencia de aminoácidos que es idéntica en un 90% o 100% a la secuencia de los primeros 76 aminoácidos de la SEQ ID NO: 2. La unidad de repetición de ubiquitina de 76 aminoácidos de la SEQ ID NO: 2 también se denomina Ub en el presente documento. En ciertas realizaciones, un resto de ubiquitina comprende 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 40, 50, 100 o más repeticiones de Ub. En ciertas realizaciones, un resto de ubiquitina comprende una o más repeticiones de Ub, excepto que el aminoácido 75º de al menos una repetición de Ub es A. En ciertas realizaciones, un resto de ubiquitina comprende una o más repeticiones de Ub, excepto que el aminoácido 76º de al menos una repetición de Ub es A. En ciertas realizaciones en las que un resto de ubiquitina comprende una o más repeticiones de Ub, el aminoácido 76º de al menos una repetición de Ub es G o A.
En ciertas realizaciones, el sustrato peptídico comprende además un agente detectable unido al péptido. El agente detectable puede unirse al péptido usando diferentes métodos, que pueden depender de la identidad del agente detectable y/o la parte del péptido que va a unirse con el agente detectable. Por ejemplo, el agente detectable puede unirse al péptido mediante residuos(s) de C del péptido. El agente detectable puede ser un marcador fluorescente, que puede unirse al sustrato peptídico mediante enlace covalente o no covalente. Un marcador fluorescente puede comprender un péptido fluorescente que puede unirse al péptido mediante enlace covalente o no covalente, por ejemplo, un enlace peptídico. Alternativamente, el agente detectable puede ser un marcador radiactivo, por ejemplo, un aminoácido radiomarcado tal como 35S-C o 35S-M.
En otras realizaciones, el sustrato peptídico puede comprender una parte de diana, es decir, una parte N-terminal o C-terminal, que se une específicamente a un agente de selección. El agente de selección puede ser un anticuerpo, que reconoce la parte de diana del sustrato peptídico. Alternativamente, el agente de selección puede ser un ión metálico divalente, que se asocia o interacciona específicamente con la parte de diana del sustrato peptídico.
Se describen además ácidos nucleicos que codifican para las diversas proteínas y sustratos peptídicos descritos en el presente documento.
Ciertas realizaciones proporcionan un método tal como se define en las reivindicaciones para seleccionar un agente que modula la actividad del proteasoma, por ejemplo, la actividad enzimática del proteasoma 26S o la partícula reguladora 19S. El método incluye proporcionar un sustrato peptídico de la solicitud, por ejemplo, un sustrato peptídico que comprende una secuencia de aminoácidos de la SEQ ID NO: 1 y uno o más restos de ubiquitina, y combinar el péptido con una mezcla de reacción adecuada para medir actividad del proteasoma en presencia de un agente de prueba. Un cambio en la actividad del proteasoma en presencia del agente de prueba en comparación con la actividad del proteasoma en ausencia del agente de prueba indica que el agente... [Seguir leyendo]
Reivindicaciones:
1. Péptido que comprende una secuencia de aminoácidos al menos idéntica en un 70% a la SEQ ID NO: 1 y al menos un resto de ubiquitina que es al menos idéntico en un 90% a la secuencia de los primeros 76 aminoácidos de la SEQ ID NO: 2, en el que el péptido es un sustrato para una metaloproteasa Rpn11 que comprende un motivo JAMM.
2. Péptido según la reivindicación 1, en el que el motivo JAMM es al menos idéntico en un 80% a la SEQ ID NO: 17.
3. Péptido según la reivindicación 1, en el que el motivo JAMM es al menos idéntico en un 80% a la SEQ ID NO: 18.
4. Péptido según una cualquiera de las reivindicaciones 1 a 3, en el que la metaloproteasa es al menos idéntica en un 80% a la secuencia de la SEQ ID NO: 16.
5. Péptido según una cualquiera de las reivindicaciones 1 a 4, que comprende además al menos 3 aminoácidos adicionales en el extremo C-terminal del péptido.
6. Péptido según la reivindicación 1, en el que la secuencia de aminoácidos tiene la secuencia de aminoácidos de la SEQ ID NO: 1, en el que al menos un aminoácido distinto de A se sustituye por A.
7. Péptido según la reivindicación 1, en el que la secuencia de aminoácidos tiene la secuencia de aminoácidos de la SEQ ID NO: 4, en el que al menos un aminoácido distinto de A se sustituye por A.
8. Péptido según una cualquiera de las reivindicaciones 1 a 5, que comprende además uno o más restos de ubiquitina en el extremo N-terminal del péptido.
9. Péptido según la reivindicación 8, en el que el resto de ubiquitina consiste en dos o más repeticiones de una secuencia de aminoácidos que es al menos idéntica en un 90% a la secuencia de los primeros 76 aminoácidos de la SEQ ID NO: 2.
10. Péptido según una cualquiera de las reivindicaciones 1 a 9, en el que el resto de ubiquitina comprende la secuencia de los primeros 76 aminoácidos de la SEQ ID NO: 2 modificada de manera que el aminoácido 75º es una A.
11. Péptido según una cualquiera de las reivindicaciones 1 a 9, en el que el resto de ubiquitina comprende la secuencia de los primeros 76 aminoácidos de la SEQ ID NO: 2 modificada de manera que el aminoácido 76º es una A.
12. Péptido según una cualquiera de las reivindicaciones 1 a 11, que comprende además un agente detectable unido al péptido.
13. Péptido según la reivindicación 12, en el que el agente detectable es un marcador fluorescente.
14. Péptido según la reivindicación 13, en el que el marcador fluorescente es un péptido fluorescente unido al extremo C-terminal del péptido mediante un enlace peptídico.
15. Péptido según la reivindicación 12, en el que el agente detectable es un marcador radiactivo.
16. Péptido según una cualquiera de las reivindicaciones 1 a 11, que comprende además una parte de diana N-terminal o parte de diana C-terminal que se une específicamente a un agente de selección.
17. Péptido según la reivindicación 16, en el que el agente de selección es un anticuerpo.
18. Péptido según la reivindicación 16, en el que el agente de selección es un ión metálico divalente.
19. Método para seleccionar un agente que modula la actividad del proteasoma de una metaloproteasa Rpn11, comprendiendo el método:
(a) proporcionar el péptido según la reivindicación 1; y
(b) combinar el péptido con un agente de prueba en una mezcla de reacción adecuada para medir la actividad de la metaloproteasa Rpn11 en condiciones que permiten que se escinda el resto de ubiquitina, en el que la metaloproteasa Rpn11 comprende una secuencia de aminoácidos al menos idéntica en un 80% a la secuencia de la SEQ ID NO: 16 y un motivo JAMM al menos idéntico en un 80% a la secuencia de la SEQ ID NO: 17;
en el que un cambio en la tasa o el grado de escisión del resto de ubiquitina a partir del péptido en presencia del agente de prueba en comparación con la tasa o el grado de escisión del resto de ubiquitina a partir del péptido en ausencia del agente de prueba indica que el agente de prueba modula la actividad del proteasoma.
20. Método según la reivindicación 19, en el que la Rpn11 está presente en una partícula reguladora 19S o un proteasoma 26S.
21. Método para seleccionar un agente que inhibe la actividad del proteasoma de una metaloproteasa Rpn11 que comprende un motivo JAMM, comprendiendo el método:
(a) proporcionar el péptido según la reivindicación 1 en una mezcla con ubistatina; y
(b) comparar la fluorescencia intrínseca de ubistatina en presencia de un agente inhibidor de prueba en la mezcla con la fluorescencia intrínseca de ubistatina en ausencia del agente inhibidor de prueba en la mezcla,
en el que una diferencia en la fluorescencia intrínseca de ubistatina en presencia del agente inhibidor de prueba en comparación con en ausencia del agente inhibidor de prueba indica que el agente inhibidor de prueba inhibe la actividad del proteasoma.
22. Método para seleccionar un agente que inhibe la actividad del proteasoma de una metaloproteasa Rpn11 que comprende un motivo JAMM, comprendiendo el método:
(a) proporcionar el péptido según la reivindicación 1;
(b) combinar el péptido de (a) con un agente inhibidor de prueba que tiene fluorescencia intrínseca en una mezcla; y
(c) medir la fluorescencia de la fluorescencia intrínseca del agente inhibidor de prueba en la mezcla;
en el que un cambio en la fluorescencia medida del agente inhibidor de prueba en la mezcla con respecto a la fluorescencia intrínseca indica que el agente inhibidor de prueba inhibe la actividad del proteasoma.
23. Método para seleccionar un agente que modula la actividad del proteasoma de una metaloproteasa Rpn11 que comprende un motivo JAMM, comprendiendo el método:
(a) proporcionar el péptido según la reivindicación 1 en una mezcla de reacción; y
(b) determinar si un agente de prueba produce un cambio en el tamaño y/o el peso molecular del péptido;
en el que un agente de prueba que produce un cambio en el tamaño y/o el peso molecular del sustrato peptídico modula la actividad del proteasoma.
24. Método según la reivindicación 23, en el que el sustrato peptídico que comprende además un marcador fluorescente.
25. Método según la reivindicación 24, en el que el cambio en el tamaño y/o el peso molecular del sustrato peptídico se determina mediante polarización de fluorescencia.
26. Método según la reivindicación 23, en el que el cambio en el tamaño y/o el peso molecular del sustrato peptídico se determina mediante electroforesis en gel en condiciones nativas, cromatografía de filtración en gel, o dispersión de la luz.
27. Método para seleccionar un agente que modula la actividad de diversos componentes de un proteasoma 26S, que comprende:
(a) proporcionar un primer sustrato peptídico que comprende el péptido según la reivindicación 1 marcado con un primer marcador fluorescente, un segundo sustrato peptídico que comprende un péptido no ubiquitinado marcado con un segundo marcador fluorescente, en el que el primer y el segundo marcadores fluorescentes son detectables a diferentes longitudes de onda;
(b) medir la fluorescencia a la longitud de onda del primer marcador detectable en presencia del proteasoma 26S y en presencia y ausencia de un agente de prueba; y
(c) medir la fluorescencia a la longitud de onda del segundo marcador detectable en presencia del proteasoma 26S y en presencia y ausencia de un agente de prueba; y
en el que un cambio en la fluorescencia del primer sustrato peptídico en presencia del agente de prueba en comparación con la fluorescencia en ausencia del agente de prueba indica que el agente de prueba modula una actividad asociada con la partícula reguladora 19S, y un cambio en la fluorescencia del segundo sustrato peptídico en presencia del agente de prueba en comparación con la fluorescencia en ausencia del agente de prueba indica que el agente de prueba modula una peptidasa del núcleo 20S.
28. Método según la reivindicación 27, en el que el proteasoma 26S, el primer sustrato peptídico y el segundo sustrato peptídico se combinan en una única mezcla de reacción.
Patentes similares o relacionadas:
Polipéptidos de unión específica novedosos y usos de los mismos, del 15 de Julio de 2020, de Pieris Pharmaceuticals GmbH: Muteína de lipocalina lagrimal humana que tiene especificidad de unión para IL-17A, en la que la muteína se une a IL-17A con una KD de aproximadamente 1 nM o menos, en la que […]
Formulaciones liofilizadas para antídoto del factor Xa, del 1 de Julio de 2020, de PORTOLA PHARMACEUTICALS, INC.: Una formulación acuosa, que comprende de 10 mM a 55 mM de arginina, de 1% a 3% de sacarosa (p/v), de 2% a 8% de manitol (p/v), y al menos 5 mg/ml de un polipéptido […]
Péptidos de unión beta amiloide y sus usos para el tratamiento y el diagnóstico de la demencia de Alzheimer, del 17 de Junio de 2020, de Priavoid GmbH: Péptido que contiene al menos una secuencia de aminoácidos que se une a especies beta amiloides y en el que la carga negativa del grupo carboxilo presente […]
Arginina desiminasa con reactividad cruzada reducida hacia anticuerpos para ADI - PEG 20 para el tratamiento del cáncer, del 6 de Mayo de 2020, de TDW Group: Una composición terapéutica que comprende una arginina desiminasa (ADI) aislada y un vehículo farmacéuticamente aceptable, en donde la ADI aislada comprende la secuencia de […]
Composiciones y métodos utilizando andamios de lisofosfatidilcolina, del 6 de Mayo de 2020, de NATIONAL UNIVERSITY OF SINGAPORE: Un método para seleccionar uno o más compuestos o ácidos grasos para determinar el transporte mediado por lisofosfatidilcolina (LPC) del uno o más compuestos o ácidos […]
Péptidos antimicrobianos y composiciones de los mismos, del 29 de Abril de 2020, de REVANCE THERAPEUTICS, INC: Composición farmacéutica o cosmética que comprende un péptido, en donde dicho péptido consiste en la secuencia de aminoácidos SEQ ID NO: 3 correspondiente […]
COMPOSICIÓN PARA LA ESTIMULACIÓN DEL SISTEMA INMUNE EN PECES QUE CONTIENE PÉPTIDOS RECOMBINANTES, PÉPTIDOS RECOMBINANTES INMUNOESTIMULANTES Y SECUENCIAS NUCLEOTÍDICAS QUE LOS CODIFICAN, del 23 de Abril de 2020, de UNIVERSIDAD DE CONCEPCION: La presente invención se refiere a una composición, una micropartícula y una formulación alimenticia que comprende péptidos recombinantes que estimulan el sistema inmune […]
Nuevo péptido con cuatro epítopos CTL unidos, del 15 de Abril de 2020, de TAIHO PHARMACEUTICAL CO., LTD.: Un péptido que consiste en 4 epítopos unidos, en el que los 4 péptidos de epítopo se seleccionan del grupo que consiste en los péptidos de epítopo CTL: el péptido como […]