SISTEMA DE ENSAYO DE TRES HÍBRIDOS.
Método de identificación de una secuencia polipeptídica que se une a un ligando especificado por el usuario,
que comprende: (i) proporcionar un ligando híbrido que tiene la fórmula general: R1-Y-R2, en la que: R1 es un primer ligando, R2 es un ligando especificado por el usuario e Y es un enlazador de polietileno que tiene la fórmula general (CH2-X-CH2)n, en la que X representa O, S, SO o SO2 y n es un número entero de 2 a 25; (ii) introducir el ligando híbrido en una población de células, conteniendo cada célula un sistema de exploración de ligando híbrido que incluye: (a) un gen indicador unido operativamente a una secuencia reguladora de la transcripción, incluyendo dicha secuencia reguladora una secuencia de ADN que se une a un dominio de unión a ADN; (b) un primer gen quimérico que codifica un primer polipéptido de fusión que comprende: un dominio de unión a ligando P1 y un dominio seleccionado de un dominio de unión a ADN o un dominio de activación de la transcripción, en el que el dominio de unión a ligando se une al primer ligando R1; y (c) un segundo gen quimérico que codifica un segundo polipéptido de fusión que comprende: un dominio de unión a ligando candidato P2 para el ligando especificado por el usuario R2 y un dominio seleccionado de un dominio de unión a ADN o un dominio de activación de la transcripción; en el que uno de los dos polipéptidos de fusión contiene un dominio de unión a ADN y el otro polipéptido de fusión contiene un dominio de activación de la transcripción; (iii) permitir que el ligando híbrido se una al dominio de unión a ligando del primer polipéptido de fusión a través del primer ligando R1 y contacte con el dominio de unión a ligando candidato del segundo polipéptido de fusión a través del ligando especificado por el usuario R2 de modo que, si R2 se une al dominio de unión a ligando candidato, se produce un aumento en el nivel de transcripción del gen indicador; (iv) identificar una célula de unión a ligando positiva en la que se haya producido un aumento en el nivel de transcripción del gen indicador; y (v) identificar la secuencia de ácido nucleico del segundo gen quimérico que codifica el dominio de unión a ligando candidato que se une al ligando especificado por el usuario R2, identificando de este modo una secuencia polipeptídica que se une a un ligando especificado por el usuario
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2002/006677.
Solicitante: Agennix USA Inc.
Agennix AG.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 101 College Road East Princeton, NJ 08540.
Inventor/es: BECKER,FRANK, COME,Jon,H, KLEY,Nikolai A, REICHEL,Christoph.
Fecha de Publicación: .
Fecha Solicitud PCT: 4 de Marzo de 2002.
Clasificación PCT:
- A61K31/01 NECESIDADES CORRIENTES DE LA VIDA. › A61 CIENCIAS MEDICAS O VETERINARIAS; HIGIENE. › A61K PREPARACIONES DE USO MEDICO, DENTAL O PARA EL ASEO (dispositivos o métodos especialmente concebidos para conferir a los productos farmacéuticos una forma física o de administración particular A61J 3/00; aspectos químicos o utilización de substancias químicas para, la desodorización del aire, la desinfección o la esterilización, vendas, apósitos, almohadillas absorbentes o de los artículos para su realización A61L; composiciones a base de jabón C11D). › A61K 31/00 Preparaciones medicinales que contienen ingredientes orgánicos activos. › Hidrocarburos.
- C07K14/00 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados.
- C12N15/62 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Secuencias de ADN que codifican proteínas de fusión.
- C12Q1/00 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones.
- G01N33/566 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › utilizando un soporte específico o proteínas receptoras como reactivos para la formación de uniones por ligando.
Clasificación antigua:
- A61K31/01 A61K 31/00 […] › Hidrocarburos.
- C07K14/00 C07K […] › Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados.
- G01N33/566 G01N 33/00 […] › utilizando un soporte específico o proteínas receptoras como reactivos para la formación de uniones por ligando.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
PDF original: ES-2360205_T3.pdf
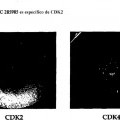


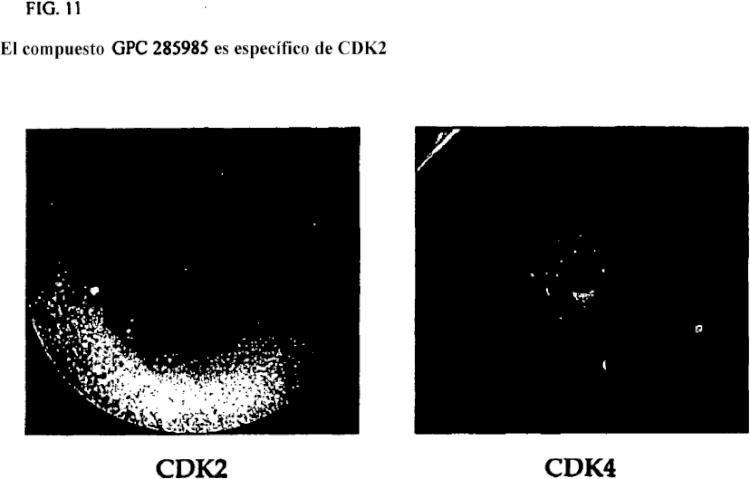
Fragmento de la descripción:
Referencia a solicitudes relacionadas
Esta solicitud reivindica prioridad respecto a la solicitud provisional de Estados Unidos 60/272.932 presentada el 2 de marzo de 2001; la solicitud provisional de Estados Unidos 60/278.233 presentada el 23 de marzo de 2001; y la solicitud provisional de Estados Unidos 60/329.437 presentada el 15 de octubre de 2001.
Antecedentes de la invención
Las interacciones de proteínas facilitan la mayoría de los procesos biológicos, incluyendo la transducción de señales y la homeostasia. Se ha avanzado en la elucidación de compañeros proteicos de interacción particulares que faciliten estos procesos biológicos mediante el desarrollo de métodos de “doble híbrido” o “trampa de interacción” para detectar y seleccionar compañeros proteicos de interacción (véase Fields y Songs (1989) Nature 340: 245-6; Gyuris et al. (1993) Cell 75: 791-803; Patente de Estados Unidos Nº 5.468.614; y Yang et al. (1995) Nucleic Acid Research 23, 1152-1156). Estos métodos dependen de la reconstitución de un activador de la transcripción nuclear a través de la interacción de dos polipéptidos compañeros de unión —es decir, un primer polipéptido fusionado a un dominio de unión a ADN (BD) y un segundo polipéptido fusionado a un dominio de activación de la transcripción (AD). Cuando el primer y el segundo polipéptidos interaccionan, la interacción puede detectarse por activación de un gen indicador que contiene sitios de unión para el dominio de unión a ADN. Para que funcione este método, ambas proteínas tienen que ser solubles y deben ser capaces de localizarse en el núcleo. Por consiguiente, la interacción de polipéptidos que normalmente se localizan en otros compartimentos puede no detectarse debido a la ausencia de otros componentes polipeptídicos no nucleares que faciliten la interacción o a modificaciones postraduccionales no nucleares particulares que no suceden en el núcleo o porque las proteínas de interacción no se pliegan apropiadamente cuando se localizan en el compartimento nuclear. En particular, el ensayo de doble híbrido nuclear es incompatible con la detección de interacciones proteicas que suceden dentro de o en la superficie de membranas celulares. Además, este ensayo no sirve para explorar interacciones de moléculas pequeñas-proteínas debido a que depende únicamente de proteínas de fusión codificadas genéticamente.
Un área fundamental de investigación en farmacología y medicina es la determinación de interacciones de ligando-receptor. La base farmacológica de la acción de fármacos a nivel celular es bastante frecuentemente la consecuencia de interacciones no covalentes entre moléculas orgánicas pequeñas terapéuticamente relevantes y proteínas de unión de alta afinidad dentro de un tipo celular específico. Estos ligandos orgánicos pequeños pueden funcionar como agonistas o antagonistas de acontecimientos reguladores clave que organizan funciones celulares tanto normales como anormales. Durante años la estrategia de la industria farmacéutica para descubrir dichos ligandos ha sido la de explorar aleatoriamente miles de moléculas pequeñas en ensayos in vitro e in vivo específicos para determinar un compuesto candidato potente para sus esfuerzos de descubrimiento de fármacos. Usando estas herramientas, puede encontrarse un compuesto candidato que ejerza efectos muy bien definidos con respecto a una función en un tipo celular particular (por ejemplo, inhibición de la producción de citocinas o de la replicación del ADN en una línea celular de cáncer particular). Sin embargo, dichos resultados pueden proporcionar escasos indicios en lo que se refiere al mecanismo de acción a nivel molecular (interacción de ligando-proteína). Además, la exploración de una acción potente sobre una función celular puede dejar pasar reactividades cruzadas de un compuesto candidato que dan origen a efectos secundarios no deseados. Dichos efectos secundarios son con frecuencia la consecuencia de proteínas con estructuras muy similares que tienen funciones diferentes, o de una proteína que cumple diferentes funciones cuando se expresa en diferentes tipos celulares, o incluso cuando se localiza en diferentes compartimentos subcelulares. Por lo tanto, la identificación de las posiblemente diversas dianas proteicas para un agente farmacológico que muestra una actividad dada es desafiante pero altamente deseable. Existe una necesidad no cumplida de un método general y eficaz para identificar las dianas celulares para estos agentes farmacológicos de modo que se acelere la búsqueda de nuevos fármacos tanto a nivel de la investigación básica como aplicada
De forma similar, existe la necesidad de una estrategia general para identificar una molécula pequeña capaz de unirse a cualquier diana celular seleccionada independientemente de su función biológica. Fowlkes et al. (WO 94/23025) y Broach et al. (WO/ 95/30012) describieron un ensayo de exploración para identificar moléculas capaces de unirse a receptores de la superficie celular de modo que se active una ruta de transducción de señales seleccionada. Estas referencias describen la modificación de rutas de señalización de levadura seleccionadas para mimetizar las etapas de la ruta de señalización de mamíferos. Esta última estrategia es específica para ciertas rutas de señalización y tiene una utilidad limitada para descubrir ampliamente moléculas pequeñas que interaccionen con cualquier diana celular. Por lo tanto, existe también una necesidad no cumplida de un método de exploración general para determinar la interacción entre moléculas pequeñas y proteínas diana para identificar nuevos fármacos que sean capaces de efectos terapéuticos específicos en una diversidad de patologías, así como para identificar agonistas y antagonistas que puedan interferir o competir con la unión de las moléculas pequeñas a estas dianas.
En este momento, existen pocas (si alguna) metodologías eficaces para identificar rápidamente una diana biológica, tal como una proteína para un ligando molecular pequeño particular. Las estrategias existentes incluyen el uso de cromatografía de afinidad, unión de ligando radiomarcado y marcaje por fotoafinidad en combinación con métodos de purificación de proteínas para detectar y aislar supuestas proteínas diana. Esto viene seguido de clonación del gen que codifica la proteína diana basándose en la secuencia peptídica de la diana aislada. Estas estrategias dependen de la abundancia de la supuesta proteína diana en la muestra y son laboriosas y meticulosas.
**(Ver fórmula)**
Crabtree et al. (documento WO/94/18317) describieron un método para activar un gen diana en células que comprenden (a) el suministro de células que contienen y son capaces de expresar (i) al menos una construcción de ADN que comprende al menos un dominio de receptor, capaz de unirse a un ligando seleccionado, fusionarse a una proteína adicional heteróloga capaz de iniciar un proceso biológico tras la exposición de la construcción de fusión al ligando, en el que el proceso biológico comprende la expresión del gen diana, en el que el ligando es capaz de unirse a dos o más proteínas de fusión, y en el que el proceso biológico sólo se inicia tras la unión del ligando a dos o más proteínas de fusión, siendo las dos proteínas de fusión iguales o diferentes, y (ii) el gen diana bajo el control de la expresión de un elemento de control que responde transcripcionalmente al inicio de dicho proceso biológico; y (b) exponer dichas células a dicho ligando en una cantidad eficaz para dar como resultado la expresión del gen indicador. Se describen además construcciones de ADN, ligandos y kits útiles para realizar dicho método. Los documentos relacionados US 5.830.462, US 5.869.337, US 6.165.787 muestran estas y otras realizaciones; en concreto, Holt et al. (documento WO 96/06097) describe la síntesis de ligandos híbridos para su uso con los presentes métodos.
El documento describe compuestos con restos de ligando de N-oxalil-pipecolilo y N-oxalil-prolilo para multimerización de inmunofilinas.
El fin previsto para estos métodos y composiciones se limita a la investigación de procesos celulares, la regulación de la síntesis de proteínas de importancia terapéutica o agrícola y la regulación de procesos celulares en terapia génica. Nada en los mismos sugiere el uso de estos métodos y composiciones para estudiar la interacción de proteínas con moléculas pequeñas, particularmente en su aplicación a la investigación farmacéutica y al desarrollo de fármacos.
Schreiber et al. (documento WO 96/13613) describen... [Seguir leyendo]
Reivindicaciones:
1. Método de identificación de una secuencia polipeptídica que se une a un ligando especificado por el usuario, que comprende:
(i) proporcionar un ligando híbrido que tiene la fórmula general: R1-Y-R2, en la que: R1 es un primer ligando, R2 es un ligando especificado por el usuario e Y es un enlazador de polietileno que tiene la fórmula general (CH2-X-CH2)n, en la que X representa O, S, SO o SO2 y n es un número entero de 2 a 25;
(ii) introducir el ligando híbrido en una población de células, conteniendo cada célula un sistema de exploración de ligando híbrido que incluye:
(a) un gen indicador unido operativamente a una secuencia reguladora de la transcripción, incluyendo dicha secuencia reguladora una secuencia de ADN que se une a un dominio de unión a ADN;
(b) un primer gen quimérico que codifica un primer polipéptido de fusión que comprende: un dominio de unión a ligando P1 y un dominio seleccionado de un dominio de unión a ADN o un dominio de activación de la transcripción, en el que el dominio de unión a ligando se une al primer ligando R1; y
(c) un segundo gen quimérico que codifica un segundo polipéptido de fusión que comprende: un dominio de unión a ligando candidato P2 para el ligando especificado por el usuario R2 y un dominio seleccionado de un dominio de unión a ADN o un dominio de activación de la transcripción;
en el que uno de los dos polipéptidos de fusión contiene un dominio de unión a ADN y el otro polipéptido de fusión contiene un dominio de activación de la transcripción;
(iii) permitir que el ligando híbrido se una al dominio de unión a ligando del primer polipéptido de fusión a través del primer ligando R1 y contacte con el dominio de unión a ligando candidato del segundo polipéptido de fusión a través del ligando especificado por el usuario R2 de modo que, si R2 se une al dominio de unión a ligando candidato, se produce un aumento en el nivel de transcripción del gen indicador;
(iv) identificar una célula de unión a ligando positiva en la que se haya producido un aumento en el nivel de transcripción del gen indicador; y
(v) identificar la secuencia de ácido nucleico del segundo gen quimérico que codifica el dominio de unión a ligando candidato que se une al ligando especificado por el usuario R2, identificando de este modo una secuencia polipeptídica que se une a un ligando especificado por el usuario.
2. Método de la reivindicación 1, en el que la secuencia de ácido nucleico que codifica el polipéptido de dominio de unión a ligando candidato del segundo polipéptido de fusión es de una biblioteca seleccionada de: una biblioteca de oligonucleótidos sintéticos, una biblioteca de ADNc, una biblioteca de fragmentos de ADN genómicos bacterianos o una biblioteca de fragmentos de ADN genómicos eucariotas.
3. Método de la reivindicación 1, que comprende además:
(a) determinar la afinidad de unión del ligando híbrido a los dominios de unión a ligando P1 y/o P2; o
(b) determinar los efectos del ligando híbrido que son independientes de la formación de un complejo trimérico que comprende el ligando híbrido, P1 y P2; o
(c) la etapa de realizar al menos un método separado adicional para confirmar que la transcripción del gen indicador depende de la presencia del ligando híbrido y los dominios de unión a ligando P1 y P2.
4. Método de la reivindicación 3, en el que la determinación de la afinidad de unión en la etapa (a) de la reivindicación 3 se realiza por resonancia de plasmón superficial.
5. Método de la reivindicación 3, en el que dicho método separado adicional en la etapa (c) de la reivindicación 3 se selecciona de: un método de ensayo de crecimiento en halo, un ensayo de crecimiento en placa de microtitulación o un ensayo de crecimiento por detección de la fluorescencia.
6. Método de la reivindicación 3, en el que dicho método separado adicional en la etapa (c) de la reivindicación 3 se realiza individualmente sobre más de aproximadamente 10, 100, 1000 ó 10000 tipos celulares de unión a ligando positivos diferentes identificados en la etapa (iv) de la reivindicación 1.
7. Método de identificación de un ligando de un polipéptido especificado por el usuario, que comprende:
(i) proporcionar al menos un ligando híbrido candidato que tiene la fórmula general R1-Y-R2, en la que R1 es un primer ligando, R2 es un ligando candidato e Y es un enlazador de polietileno que tiene la fórmula general (CH2-X-CH2)n, en la que X representa O, S, SO o SO2 y n es un número entero de 2 a 25;
(ii) introducir el ligando híbrido candidato en al menos una célula que contiene un sistema de exploración de ligando híbrido, que incluye:
(a) un gen indicador unido operativamente a una secuencia reguladora de la transcripción, incluyendo dicha secuencia reguladora una secuencia de ADN que se une a un dominio de unión a ADN;
(b) un primer gen quimérico que codifica un primer polipéptido de fusión, que comprende: un dominio de unión a ligando y un dominio seleccionado de: un dominio de unión a ADN o un dominio de activación de la transcripción, en el que el dominio de unión a ligando se une al primer ligando R1; y
(c) un segundo gen quimérico que codifica un segundo polipéptido de fusión, que comprende: un dominio de unión a ligando especificado por el usuario para el ligando candidato R2 y un dominio seleccionado de: un dominio de unión a ADN o un dominio de activación de la transcripción;
**(Ver fórmula)**
en el que uno de los dos polipéptidos de fusión contiene un dominio de unión a ADN y el otro polipéptido de fusión contiene un dominio de activación de la transcripción;
(iii) permitir que el ligando híbrido candidato se una al dominio de unión a ligando del primer polipéptido de fusión a través del primer ligando R1 y contacte con el dominio de unión a ligando especificado por el usuario del segundo polipéptido de fusión a través del ligando candidato R2 de modo que, si el dominio de unión a ligando especificado por el usuario se une al ligando candidato R2, se produce un aumento en el nivel de transcripción del gen indicador;
(iv) identificar el ligando híbrido candidato que causa un aumento en el nivel de transcripción del gen indicador en la célula, identificando de este modo el ligando candidato en el ligando híbrido candidato como un ligando para el polipéptido especificado por el usuario.
8. Método de la reivindicación 1 ó 7, en el que el gen indicador se selecciona de: HIS3, LEU2, TRP2, TRP1, ADE2, LYS2, URA3, CYH1, CAN1, lacZ, gfp o CAT.
9. Método in vitro para inducir o permitir la detección de un acontecimiento biológicamente detectable, que comprende:
(i) proporcionar al menos una célula que comprende al menos una secuencia de ácido nucleico que codifica un polipéptido de fusión, que incluye:
(a) al menos un dominio de unión a ligando; y
(b) un primer dominio funcional que por sí mismo no es capaz de inducir o permitir la detección del acontecimiento detectable;
(ii) proporcionar un ligando híbrido de fórmula general R1-Y-R2, en la que R1 es diferente de R2, al menos uno de R1 y R2 no es un péptido, R1 o R2 representa un ligando que se une a dicho dominio de unión a ligando; Y representa un enlazador de polietileno que tiene la fórmula general (CH2-X-CH2)n, en la que X representa O, S, SO o SO2 y n es un número entero de 2 a 25; y en el que la unión de dicho ligando híbrido a dicho dominio de unión a ligando pone al primer dominio funcional en las proximidades de un segundo dominio funcional, induciendo o permitiendo de este modo la detección del acontecimiento detectable; y
(iii) exponer dicha al menos una célula a una cantidad eficaz de dicho ligando híbrido para poner el primer dominio funcional en las proximidades de un segundo dominio funcional;
induciendo o permitiendo de este modo la detección del acontecimiento biológicamente detectable.
10. Método in vitro para investigar la relación de estructura-actividad de un ligando con un dominio de unión a ligando, que comprende:
(i) proporcionar un ligando híbrido R1-Y-R2, en el que
(a) R1 representa un primer ligando seleccionado de: un esteroide, ácido retinoico, antibiótico beta-lactámico, canabinoide, ácido nucleico, polipéptido, FK506, derivado de FK506, rapamicina, tetraciclina, metotrexato, novobiocina, maltosa, glutatión, biotina, vitamina D, dexametasona, estrógeno, progesterona, cortisona, testosterona, níquel, derivado de 2,4-diaminopteridina o ciclosporina, o un derivado de los mismos con modificaciones estructurales minoritarias;
(b) Y representa un enlazador de polietileno que tiene la fórmula general (CH2-X-CH2)n, en la que X representa O, S, SO o SO2 y n es un número entero de 2 a 25; y
(c) R2 representa un segundo ligando especificado por el usuario que es diferente de R1 y se selecciona de: un péptido, ácido nucleico, carbohidrato, polisacárido, lípido, prostaglandina, haluro de acilo, alcohol, aldehído, alcano, alqueno, alquino, alquilo, haluro de alquilo, alcaloide, amina, hidrocarburo aromático, éster de sulfonato, carboxilato ácido, haluro de arilo, éster, fenol, éter, nitrilo, anhídrido del ácido carboxílico, amida, sal de amonio cuaternario, imina, enamina, óxido de amina, cianohidrina, organocadmio, aldol, organometálico, hidrocarburo aromático, nucleósido o un nucleótido;
(ii) proporcionar células que comprenden una proteína de fusión que incluye:
(a) al menos un dominio de unión a ligando; y
(b) un primer dominio funcional heterólogo respecto al dominio de unión a ligando, que por sí mismo no es capaz de inducir o permitir la detección de un acontecimiento detectable, pero que es capaz de inducir o permitir la detección de un acontecimiento detectable cuando se pone en las proximidades de un segundo dominio funcional;
**(Ver fórmula)**
en el que se proporciona una pluralidad de ligandos híbridos que comprenden variantes estructurales de dicho segundo ligando R2 en la etapa (i), o se proporciona una pluralidad de proteínas de fusión que comprenden variantes estructurales de dicho dominio de unión a ligando en la etapa (ii);
(iii) exponer dichas células que comprenden cada proteína de fusión a una cantidad eficaz de cada ligando híbrido, de modo que el primer domino funcional puede ponerse en las proximidades de un segundo dominio funcional, induciendo o permitiendo de este modo la detección de un acontecimiento detectable;
(iv) medir la presencia, cantidad o actividad de cualquier acontecimiento detectable así inducido o permitido en la etapa (iii), investigando de este modo la relación de estructura-actividad entre dicho segundo ligando y el dominio de unión a ligando.
11. Método de la reivindicación 9 ó 10, en el que dicho primer dominio funcional se selecciona de: un dominio de unión a ADN, un dominio de activación de la transcripción, un subdominio carboxi-terminal de una ubiquitina de tipo salvaje, un subdominio amino-terminal de una ubiquitina o un subdominio amino-terminal de ubiquitina mutante de asociación reducida.
12. Método de cualquiera de las reivindicaciones 1, 7, 9 y 10, en el que dicha célula o células se seleccionan de células de levadura, mamífero o procariotas.
13. Método de la reivindicación 12, en el que dicha célula o células son levadura.
14. Método de la reivindicación 12, en el que dicha célula o células son de mamífero.
15. Método de la reivindicación 1, 7, 9 y 10, en el que el dominio de unión a ligando que se une a R1 es la dihidrofolato reductasa (DHFR).
16. Método para identificar un ligando híbrido que tiene la estructura general R1-Y-R2 adecuada para un ensayo in vivo, en el que dicho ensayo implica:
(i) el uso de un ligando híbrido y
(ii) de al menos un polipéptido de fusión que incluye:
(a) al menos un dominio de unión a ligando P; y
(b) un dominio funcional que por sí mismo no es capaz de inducir o permitir la detección de un acontecimiento detectable;
y en el que dicho método implica las etapas de:
(iii) sintetizar una pluralidad de ligandos híbridos R1-Y-R2 que difieren por una pluralidad de enlazadores Y diferentes, en el que:
(a) dicho enlazador tiene la estructura general (CH2-X-CH2)n, en la que X representa O, S, SO o SO2 y n es un número entero de 2 a 25, y la pluralidad de enlazadores difieren en n,
(b) R1 y R2 son diferentes, y
(c) al menos uno de R1 y R2 no es un péptido; y
(iv) ensayar cada ligando híbrido en dicha pluralidad de ligandos híbridos individualmente para determinar su eficacia para inducir o permitir la detección del acontecimiento detectable; y
(v) seleccionar un ligando híbrido con un enlazador particular que posea una eficacia adecuada para inducir o permitir la detección del acontecimiento detectable.
17. Composición que comprende:
(i) Un ligando híbrido representado por la fórmula general: R1-Y-R2, en la que:
(a) R1 representa un primer ligando seleccionado de: un esteroide, ácido retinoico, antibiótico beta-lactámico, canabinoide, ácido nucleico, polipéptido, FK506, derivado de FK506, rapamicina, tetraciclina, metotrexato, derivado de 2,4-diaminopteridina, novobiocina, maltosa, glutatión, biotina, vitamina D,
dexametasona, estrógeno, progesterona, cortisona, testosterona, níquel o ciclosporina, o un derivado de los mismos con modificaciones estructurales menores;
(b) Y representa un enlazador de polietileno que tiene la fórmula general (CH2-X-CH2)n, en la que X representa O, S, SO o SO2 y n es un número entero de 2 a 25;
(c) R2 representa un segundo ligando especificado por el usuario diferente de R1 seleccionado de: un péptido, ácido nucleico, carbohidrato, polisacárido, lípido, prostaglandina, haluro de acilo, alcohol, aldehído, alcano, alqueno, alquino, alquilo, haluro de alquilo, alcaloide, amina, hidrocarburo aromático, éster de sulfonato, carboxilato ácido, haluro de arilo, éster, fenol, éter, nitrilo, anhídrido del ácido carboxílico, amida, sal de amonio cuaternario, imina, enamina, óxido de amina, cianohidrina, organocadmio, aldol, organometálico, hidrocarburo aromático, nucleósido o nucleótido;
(ii) un polipéptido de fusión que incluye:
(a) al menos un dominio de unión a ligando; y
(b) un primer dominio funcional heterólogo al dominio de unión a ligando que por sí mismo no es capaz de inducir o permitir la detección de un acontecimiento detectable, pero que es capaz de inducir o permitir la detección de un acontecimiento detectable cuando se pone en las proximidades de un segundo dominio funcional.
**(Ver fórmula)**
18. Composición de la reivindicación 17, en la que la composición es un complejo.
19. Composición de la reivindicación 17, en la que la composición se proporciona en un entorno seleccionado de: una célula, un recipiente, un kit, una solución o un medio de cultivo.
20. Uso de un compuesto representado por la fórmula general R1-Y-R2, en la que:
(i) R1 representa un primer ligando seleccionado de: un esteroide, ácido retinoico, antibiótico beta-lactámico, canabinoide, ácido nucleico, polipéptido, FK506, derivado de FK506, rapamicina, tetraciclina, metotrexato, novobiocina, maltosa, glutatión, biotina, vitamina D, dexametasona, estrógeno, progesterona, cortisona, testosterona, níquel, 2,4-diaminopteridina o ciclosporina, o un derivado de los mismos con modificaciones estructurales menores;
(ii) Y representa un enlazador de polietileno que tiene la fórmula general (CH2-X-CH2)n, en la que X representa O, S, SO o SO2 y n es un número entero de 2 a 25; y
(iii) R2 representa un segundo ligando especificado por el usuario diferente de R1 seleccionado de: un péptido, ácido nucleico, carbohidrato, polisacárido, lípido, prostaglandina, haluro de acilo, alcohol, aldehído, alcano, alqueno, alquino, alquilo, haluro de alquilo, alcaloide, amina, hidrocarburo aromático, éster de sulfonato, carboxilato ácido, haluro de arilo, éster, fenol, éter, nitrilo, anhídrido del ácido carboxílico, amida, sal de amonio cuaternario, imina, enamina, óxido de amina, cianohidrina, organocadmio, aldol, organometálico, hidrocarburo aromático, nucleósido o un nucleótido;
para identificar la unión de un polipéptido a un ligando especificado por el usuario en un sistema de ensayo de triple híbrido;
para inducir o permitir la detección de un acontecimiento biológicamente detectable en un sistema de ensayo de triple híbrido;
para investigar la relación de estructura-actividad de un ligando respecto a un dominio de unión a ligando en un sistema de ensayo de triple híbrido;
para identificar un compuesto que inhibe una interacción entre un ligando y un polipéptido en un sistema de ensayo de triple híbrido; o
como ligando híbrido en cualquiera de los métodos de acuerdo con las reivindicaciones 1 a 16 y 36 a 38.
21. Método de cualquiera de las reivindicaciones 1, 7, 9 y 16 en el que:
(a) R1 se selecciona de: un esteroide, ácido retinoico, antibiótico beta-lactámico, canabinoide, ácido nucleico, polipéptido, FK506, derivado de FK506, rapamicina, tetraciclina, metotrexato, novobiocina, maltosa, glutatión, biotina, vitamina D, dexametasona, estrógeno, progesterona, cortisona, testosterona, níquel, derivado de 2,4-diaminopteridina o ciclosporina, o un derivado de los mismos con modificaciones estructurales menores; y
(b) R2 es diferente de R1 y se selecciona de: un péptido, ácido nucleico, carbohidrato, polisacárido, lípido, prostaglandina, haluro de acilo, alcohol, aldehído, alcano, alqueno, alquino, alquilo, haluro de alquilo, alcaloide, amina, hidrocarburo aromático, éster de sulfonato, carboxilato ácido, haluro de arilo, éster, fenol, éter, nitrilo, anhídrido del ácido carboxílico, amida, sal de amonio cuaternario, imina, enamina, óxido de amina, cianohidrina, organocadmio, aldol, organometálico, hidrocarburo aromático, nucleósido o un
**(Ver fórmula)**
nucleótido;
22. Método de cualquiera de las reivindicaciones 1, 10 y 21, o la composición de la reivindicación 17, o el uso de la reivindicación 20, en el que el primer ligando R1 del ligando híbrido se une al dominio de unión a ligando P1 con gran afinidad.
23. Método de la reivindicación 22, en el que la afinidad de unión corresponde a una constante de disociación de ligando/proteína de unión a ligando KD de menos de 1 M.
24. Método de cualquiera de las reivindicaciones 1, 10 y 21, o la composición de la reivindicación 17, o el uso de la reivindicación 20, en el que el primer ligando es capaz de formar un enlace covalente con el dominio de unión a ligando P1.
25. Método de cualquiera de las reivindicaciones 1, 10 y 21, o la composición de la reivindicación 17, o el uso de la reivindicación 20, en el que X es O.
26. Método de cualquiera de las reivindicaciones 1, 10 y 21, o la composición de la reivindicación 17, o el uso de la reivindicación 20, en el que Y es (CH2-O-CH2)n, en el que n = 2 a 5.
27. Método, composición o uso de la reivindicación 26, en el que n es 3, 4 ó 5.
28. Método, composición o uso de la reivindicación 27, en el que n es 3.
29. Método, composición o uso de la reivindicación 27, en el que n es 5.
30. Método de la reivindicación 10 ó 21, o la composición de la reivindicación 17, o el uso de la reivindicación 20, en el que R1 se selecciona de dexametasona, metotrexato, un derivado de metotrexato, FK506, un derivado de FK506 o un derivado de 2,4-diaminopteridina.
31. Método, composición o uso de la reivindicación 30, en el que R1 es metotrexato.
32. Método, composición o uso de la reivindicación 30, o el uso de la reivindicación 20, en el que R1 es dexametasona.
33. Método de la reivindicación 10 ó 21, o la composición de la reivindicación 17, o el uso de la reivindicación 20, en el que al menos uno de R1 y R2 no es un péptido.
34. Método de cualquiera de las reivindicaciones 1, 10 y 21, o la composición de la reivindicación 17, o el uso de la reivindicación 20, en el que R1 es metotrexato, Y es (CH2-O-CH2)n y n = 2 a 5.
35. Método de cualquiera de las reivindicaciones 1, 10 y 21, o la composición de la reivindicación 17, o el uso de la reivindicación 20, en el que R2 se une a o inhibe una quinasa.
36. Método in vitro de identificación de un compuesto que inhibe una interacción entre un ligando y un polipéptido, que comprende las etapas de:
(a) proporcionar un entorno en el que se produce dicha interacción, en el que el entorno es una célula que contiene una composición de cualquiera de las reivindicaciones 17, 18 y 22 a 35.
(b) poner en contacto el entorno con un compuesto de ensayo; y
(c) determinar si dicho compuesto de ensayo inhibe dicha interacción, identificando de este modo un compuesto que inhibe la interacción entre un ligando y un polipéptido.
37. Método para identificar un nuevo modulador de una proteína conocida determinada, incluyendo una quinasa, que usa una célula que contiene una composición de cualquiera de las reivindicaciones 17, 18 y 22 a 35.
38. Kit que comprende:
(i) al menos un polinucleótido que incluye un fragmento de ADN unido a una secuencia codificante de un primer dominio funcional heterólogo respecto al fragmento de ADN que por sí mismo no es capaz de inducir o permitir la detección de un acontecimiento detectable, pero que es capaz de inducir o permitir la detección de un acontecimiento detectable cuando se pone en las proximidades de un segundo dominio funcional;
(ii) un ligando híbrido de estructura general R1-Y-R2, en la que R2 es diferente de R1, uno de R1 y R2 es un ligando no peptídico e Y es un enlazador
en el que dicho kit se caracteriza además por el hecho de que:
(a) uno de R1 y R2 se une a o inhibe una quinasa;
(b) Y es de estructura general (CH2-O-CH2)n, en la que X representa O, S, SO o SO2 y n es un número entero de 2 a 25; o
(c) el primer dominio funcional es un subdominio carboxi-terminal de ubiquitina o un subdominio aminoterminal de ubiquitina.
Patentes similares o relacionadas:
Polipéptidos de unión específica novedosos y usos de los mismos, del 15 de Julio de 2020, de Pieris Pharmaceuticals GmbH: Muteína de lipocalina lagrimal humana que tiene especificidad de unión para IL-17A, en la que la muteína se une a IL-17A con una KD de aproximadamente 1 nM o menos, en la que […]
Formulaciones liofilizadas para antídoto del factor Xa, del 1 de Julio de 2020, de PORTOLA PHARMACEUTICALS, INC.: Una formulación acuosa, que comprende de 10 mM a 55 mM de arginina, de 1% a 3% de sacarosa (p/v), de 2% a 8% de manitol (p/v), y al menos 5 mg/ml de un polipéptido […]
Péptidos de unión beta amiloide y sus usos para el tratamiento y el diagnóstico de la demencia de Alzheimer, del 17 de Junio de 2020, de Priavoid GmbH: Péptido que contiene al menos una secuencia de aminoácidos que se une a especies beta amiloides y en el que la carga negativa del grupo carboxilo presente […]
Arginina desiminasa con reactividad cruzada reducida hacia anticuerpos para ADI - PEG 20 para el tratamiento del cáncer, del 6 de Mayo de 2020, de TDW Group: Una composición terapéutica que comprende una arginina desiminasa (ADI) aislada y un vehículo farmacéuticamente aceptable, en donde la ADI aislada comprende la secuencia de […]
Composiciones y métodos utilizando andamios de lisofosfatidilcolina, del 6 de Mayo de 2020, de NATIONAL UNIVERSITY OF SINGAPORE: Un método para seleccionar uno o más compuestos o ácidos grasos para determinar el transporte mediado por lisofosfatidilcolina (LPC) del uno o más compuestos o ácidos […]
Péptidos antimicrobianos y composiciones de los mismos, del 29 de Abril de 2020, de REVANCE THERAPEUTICS, INC: Composición farmacéutica o cosmética que comprende un péptido, en donde dicho péptido consiste en la secuencia de aminoácidos SEQ ID NO: 3 correspondiente […]
COMPOSICIÓN PARA LA ESTIMULACIÓN DEL SISTEMA INMUNE EN PECES QUE CONTIENE PÉPTIDOS RECOMBINANTES, PÉPTIDOS RECOMBINANTES INMUNOESTIMULANTES Y SECUENCIAS NUCLEOTÍDICAS QUE LOS CODIFICAN, del 23 de Abril de 2020, de UNIVERSIDAD DE CONCEPCION: La presente invención se refiere a una composición, una micropartícula y una formulación alimenticia que comprende péptidos recombinantes que estimulan el sistema inmune […]
Nuevo péptido con cuatro epítopos CTL unidos, del 15 de Abril de 2020, de TAIHO PHARMACEUTICAL CO., LTD.: Un péptido que consiste en 4 epítopos unidos, en el que los 4 péptidos de epítopo se seleccionan del grupo que consiste en los péptidos de epítopo CTL: el péptido como […]