PROCESO PARA GENERAR DIVERSIDAD IN VIVO CON USO DIAGNOSTICO, TERAPEUTICO O VACUNAL.
Se describe un proceso para generar diversidad in vivo con uso diagnóstico,
terapéutico o vacunal. Para ello se emplea una levadura que contiene el ARN de interés sobre el que se quiere obtener diversidad y las proteínas necesarias para replicarlo con una ARN polimerasa dependiente de ARN que introduce mutaciones y para empaquetarlo de forma heteróloga en partículas similares a virus. La población de los nuevos ARN sintetizados contiene variantes del original que son igualmente traducidas y sometidas a nuevas rondas de empaquetamiento y replicación. La presencia de la pared celular evita que las partículas similares a virus lisen la célula y se puedan transmitir de una generación a otra por citoducción. La diversidad así generada es acumulativa en el tiempo y limitada únicamente al ARN de interés, dejando estable el resto del genoma del vehiculo. El proceso ofrece además la posibilidad de secretar los productos generados al exterior o mantenerlos unidos a la pared celular, y de monitorizar la diversidad generada tanto genotípicamente como fenotípicamente
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P200700278.
Solicitante: GARCIA QUINTANILLA, ALBERTO.
Nacionalidad solicitante: España.
Provincia: SEVILLA.
Inventor/es: GARCIA QUINTANILLA,ALBERTO.
Fecha de Solicitud: 23 de Enero de 2007.
Fecha de Publicación: .
Fecha de Concesión: 12 de Enero de 2011.
Clasificación Internacional de Patentes:
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/63 C12N 15/00 […] › Introducción de material genético extraño utilizando vectores; Vectores; Utilización de huéspedes para ello; Regulación de la expresión.
- C12N15/81 C12N 15/00 […] › para levaduras.
Clasificación PCT:
- C12N15/10 C12N 15/00 […] › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/63 C12N 15/00 […] › Introducción de material genético extraño utilizando vectores; Vectores; Utilización de huéspedes para ello; Regulación de la expresión.
- C12N15/81 C12N 15/00 […] › para levaduras.
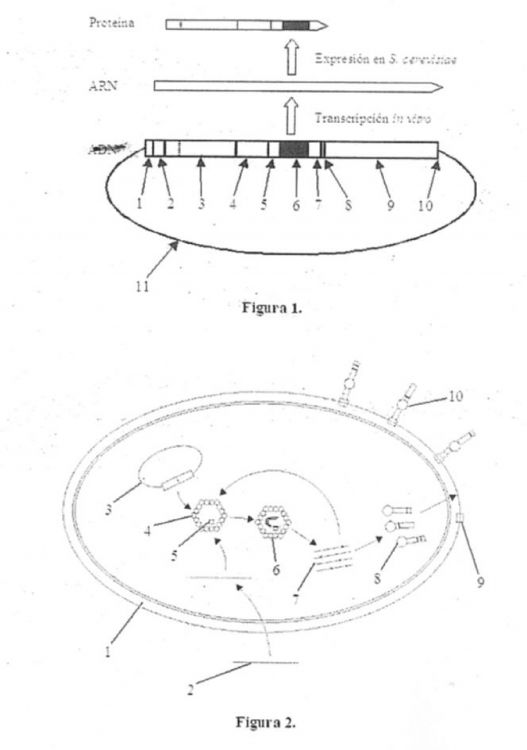
Fragmento de la descripción:
Proceso para generar diversidad in vivo con uso diagnóstico, terapéutico o vacunal.
Sector de la técnica
La invención se enmarca dentro del sector farmacéutico y está dirigida principalmente al desarrollo de vacunas humanas y veterinarias contra enfermedades causadas por agentes virales caracterizados por su elevada tasa de mutación y diversidad.
Estado de la técnica
Uno de los principales mecanismos de los virus para eludir el sistema inmunitario y adaptarse a distintos nichos es su capacidad de generar diversidad, tanto a nivel poblacional como en individuos infectados. Esta variabilidad se debe a la capacidad de recombinación y elevada tasa de mutación viral, siendo esta última mayor en aquellos virus que emplean una ARN polimerasa dependiente de ARN (RDRP) o una transcriptasa inversa durante su ciclo de replicación. Todo ello supone un obstáculo considerable en el diseño de vacunas eficaces. En el mejor de los casos, como ocurre con el virus de la gripe, implica variar anualmente la composición de la vacuna según sean las cepas circulantes. Sin embargo, para otros muchos virus como el virus de la inmunodeficiencia humana (VIH) todavía no existe ninguna.
Para tratar de dar solución a este problema se han desarrollado distintas aproximaciones que podrían agruparse en cuatro grandes grupos. Una de ellas consiste en la búsqueda de epítopos ampliamente conservados [De Groot et al. Vaccine. 2005;23:2136-481. Otra estrategia es el empleo de secuencias consenso que permiten reducir la distancia efectiva entre cepas a la mitad [Gao et al. Expert Rev Vaccines. 2004;3:S161-81. Una tercera línea de acción se basa en la creación de antígenos que resultan de la mezcla combinatoria de péptidos o ADN partiendo de poblaciones diversas, de las que destacan principalmente los "antígenos revueltos" basados en la mezcla de epítopos solapables [Thomson et al. Vaccine. 2005;23:4647-57] y el "barajado de ADN" que emplea técnicas de evolución molecular dirigida [Locher et al. Expert Opin Biol Ther. 2004;4:589-97].
Una cuarta vía considera la inclusión simultánea de múltiples cepas o variantes de una secuencia. Su candidato más representativo probablemente sea PolyEnvl [Hurwitz et al. Curr Drug Targets Infect Disord. 2005;5:143-56] que contiene más de 50 envueltas diferentes del VIH y que ha demostrado respuestas inmunitarias más amplias y duraderas que las formulaciones que contenían sólo una. En este mismo sentido y para solventar el uso tan limitado de secuencias, se han propuesto otros candidatos capaces de generar diversidad in vitro y que aumentan en varios órdenes de magnitud la diversidad incluida en los mismos. De entre éstos sobresalen los "mixotopos" de un mismo epítopo [Grass-Masse et al. Pept Res. 1992;5:211-6], las "construcciones hipervariables de epítopos" que se sintetizan usando para cada posición concreta distintas proporciones de aminoácidos según su frecuencia en las bases de datos [Anderson et al. Vaccine. 1994;12:736-40] y de modo similar los "inmunógenos de múltiples epítopos" [Hewer et al., J Theor Biol. 2005;233:85-90]. Así mismo, dado el gran número de variantes antigénicas que incluyen, estas aproximaciones abarcan también epítopos conformacionales (denominados mimotopos) y ofrecen un interés añadido para su uso en los ensayos diagnósticos [Hewer et al. Vaccine. 2005;23:2164-7]. Pese a todo, su síntesis resulta algo compleja y requieren de adyuvantes para desatar respuestas inmunes significativas en los modelos animales.
La producción de vacunas atenuadas o inactivadas conlleva la infección de huevos embrionarios, cultivos celulares o animales vivos, y consigo la generación de cierta variabilidad. Ésta, sin embargo, es puntual y se limita a cada ronda de infección, que suele provocar la lisis celular tras la cual se recogen los virus liberados. No obstante, hasta la fecha no se ha descrito ninguna estrategia capaz de generar diversidad de forma sostenida in vivo, y menos de forma limitada a la región de interés. Así, el uso de retrotransposones, de mutágenos o de vectores bacterianos con mutaciones en algún gen relacionado con su maquinaria de replicación permiten generar diversidad in vivo, pero afectan también al genoma del vehículo empleado, como sucede con la cepa de Escherichia coli XL-1 Red comercializada por Stratagene.
Otro problema clave es la necesidad de vacunas capaces de proteger en las mucosas. De este modo se controlaría el principal portal de entrada y reservorio de muchos patógenos, tal como ocurre con el tejido linfoide asociado al intestino (GALT), que comprende en tomo al 70% de las células del sistema inmunitario humano y queda gravemente infectado por el VIH durante las primeras semanas.
Existen distintas rutas para la inmunización de mucosas, como la nasal, oral, rectal, vaginal, transdérmica o respiratoria vía aerosol. La vía oral resulta particularmente atractiva por ser barata e ideal para campañas de vacunación masiva. Así mismo permite dirigir las células activadas al sistema respiratorio y genital [McDermott et al. J Immunol. 1979;122:1892-8] y a las glándulas salivares y mamarias [Holmgren et al. Nat Med. 2005;11:545-53] además de la mucosa intestinal, lo que facultaría su uso contra la transmisión sexual y en neonatos por amamantamiento.
El éxito para desatar una respuesta inmune por esta ruta depende principalmente de la elección de vectores apropiados, puesto que deben alcanzar la mucosa intestinal sin deteriorarse y poseer capacidad adyuvante intrínseca. Para ello, suelen usarse vectores virales o bacterianos que colonicen el tracto digestivo de forma natural, tales como poliovirus, rotavirus, Salmonella spp. o Lactobacilus spp. También el uso de levaduras [Stapanishcheva et al. Zh Microbiol Epidemiol Immunobiol. 1959;30:38-43] y concretamente de Saccharomyces cerevisiae ha sido descrito previamente [Duke et al. 1998. US Patent 5,830,463]. Las levaduras son fáciles de manipular, pueden producirse a gran escala de forma económica y someterse a procesos de liofilización para omitir la cadena de frío. Además soportan procesos que los vectores bacterianos no admiten y es posible la producción de glicoproteínas humanizadas [Wildt et al. Nat Rev Microbiol. 2005;3:119-28].
La mayoría de cepas de S. cerevisiae portan virus de ARN de doble cadena sin que éstos lisen ni disminuyan el crecimiento celular. Uno de los más comunes y mejor estudiados [Wickner. Microbiol Rev. 1996;60:250-65] es el virus citoplasmático L-A (también denominado L1 o ScV-L-A) del género Totivirus, que está presente en tomo a unas mil copias por célula y se transmite por citoducción a las células hijas. Este virus posee un ARN de unas 4,6 kilobases que codifica para una proteína mayor de la envuelta (Gag) y una polimerasa de ARN dependiente de ARN (RDRP) desfasada-1 base en la pauta de lectura, lo que asegura su incorporación en cada virión. El gen pol de la RDRP porta una secuencia de empaquetamiento y otra de unión a la RDRP. De ese modo, cada ARN (+) se encapsida, replica y transcribe dentro de la partícula viral donde las nuevas hebras (+) son secretadas al citoplasma y traducidas pese a no tener capuchón 5' ni cola poli-A en 3'. La incorporación in vivo [Fujimura et al. Cell. 1990;62:819-28] e in vitro [Ebihara et al. Biochem Biophys Res Commun. 1999;263:23-7] de transcritos heterólogos de ARN con el sitio de empaquetamiento dentro de los viriones ha sido descrita previamente, lo que posibilita el diseño de partículas similares a virus (VLPs) quiméricas.
El uso de VLPs se debe a la buena respuesta inmune que genera el organismo frente a antígenos de tamaño viral, así como a la demostración de inmunogenicidad tras su administración oral [Nicollier-Jamot et al. Vaccine. 2004;22:1079-86, Niikura et al. Virology. 2002;293:273-80, Tacket et al. Clin Immunol. 2003;108:241-7]. Su producción ha sido descrita desde células de insecto, a plantas o levaduras. Las de células de insecto se basan generalmente en el uso de baculovirus y están disponibles comercialmente. En el caso de levaduras, el modelo por excelencia es la vacuna contra la hepatitis B [Valenzuela et al. Nature. 1982;298:347-50], pero existen otros tipos descritos, como los basados en el retrotransposón Ty [Kingsman et al. Ann N Y Acad Sci. 1995;754:202-13] o virus...
Reivindicaciones:
1. Proceso para generar diversidad in vivo con uso diagnóstico, terapéutico o vacunal caracterizado por al menos tres componentes: (a) una cepa de levadura no patógena que ha sido previamente curada de posibles virus endógenos, (b) un ARN que incluye la región de interés sobre la cual se quiere generar diversidad, y (c) un vector que codifica para las VLPs y las RDRPs asociarlas que empaquetarán y replicarán el ARN de interés en trans, y que comprende las siguientes etapas: (1) transformación de la levadura con el vector de expresión que codifica para las VLPs y las RDRPs asociadas, (2) expresión de las VLPs y las RDRPs asociarlas, (3) transformación de la levadura con el ARN que incluye la región de interés así como las secuencias necesarias para ser empaquetado por las VLPs y replicado por las RDRPs. A partir de ahí el proceso ocurre por sí solo. (4) El ARN de interés es empaquetado por las VLPs y replicado por las RDRPs. (5) Las hebras recién sintetizadas contienen mutaciones al azar y son traducidas por la maquinaria celular en el citoplasma generándose una población cada vez más diversa formada por variantes del producto original. Dichas hebras (6) vuelven a ser empaquetadas y replicadas por otras VLPs, cerrando así el ciclo y generando más diversidad cada ronda de replicación del ARN de interés. Gracias a la pared celular de la levadura, las VLPs no tienen fase extracelular y se transmiten a las células hijas por citoducción sin usar la célula. (7) Por último se monitoriza la diversidad generada y selecciona si se desea los productos de interés antes de su uso final.
2. Proceso según reivindicación 1 donde la levadura empleada es preferiblemente Saccharomyces cerevisiae.
3. Proceso según reivindicación 1 donde la levadura empleada contiene otros marcadores, genes, modificaciones, patrones de glicosilación y/o mutaciones que facilitan la expresión correcta del producto de interés.
4. Proceso según reivindicación 1 donde la levadura contiene otros receptores, ligandos o dominios heterólogos de unión a dianas de interés.
5. Proceso según reivindicación 1 donde la levadura contiene otros genes heterólogos de interés.
6. Proceso según reivindicación 1 donde las VLPs que empaquetan y replican el ARN sujeto a diversidad se basan en cualquier virus de ARN que use una RDRP en su ciclo celular.
7. Proceso según reivindicación 1 donde las VLPs que empaquetan y replican el ARN sujeto a diversidad se basan preferiblemente en el totivirus L-A.
8. Proceso según reivindicación 1 donde las VLPs que empaquetan y replican el ARN sujeto a diversidad están codificadas por uno o varios vectores circulares o lineales, que se mantienen episomalmente o integrados en el genoma de la levadura, y se expresan bajo cualquier promotor constitutivo o inducible conocido.
9. Proceso según reivindicación 1 donde las VLPs que empaquetan y replican el ARN sujeto a diversidad portan epítopos o antígenos heterólogos intercalados.
10. Proceso según reivindicación 1 donde la región de interés es cualquier secuencia de la que se quiera obtener diversidad, sea natural o artificial, incluyendo epítopos, antígenos, VLPs, ARN interferentes o combinaciones de éstos.
11. Proceso según reivindicación 1 donde el ADN que codifica para el ARN sujeto a diversidad y que comprende la región de interés es un vector lineal o circular, con o sin origen de replicación, bajo un promotor inducible o constitutivo, que se integra o no en el genoma de la levadura, que se mantiene episomalmente o se pierde con el tiempo, y es introducido en la levadura por cualquier método descrito.
12. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés es sintetizado in vitro mediante transcripción por ARN polimerasas e introducido en la levadura por electroporación o cualquier otro método descrito.
13. Proceso según reivindicación 1 donde el ARN sujeto a diversidad comprende la región de interés, así como las secuencias y características necesarias para ser replicado por una RDRP y empaquetado en VLPs.
14. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés es único o segmentado.
15. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés contiene una secuencia que codifica para una proteína de anclaje a la pared celular de la levadura.
16. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés contiene una secuencia señal para secretar el producto al medio extracelular.
17. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés contiene dianas de corte de enzimas específicas.
18. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés contiene otras secuencias que codifican para adyuvantes, receptores, ligandos y/o dominios de unión a otras dianas de interés.
19. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés contiene una o varias secuencias espaciadoras.
20. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés contiene otros marcadores o epítopos para su purificación y/o de control.
21. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés contiene cambios en la pauta de lectura.
22. Proceso según reivindicación 1 donde el ARN sujeto a diversidad y que comprende la región de interés contiene genes esenciales o tóxicos para la levadura.
23. Proceso según reivindicación 1 donde la diversidad generada in vivo es acumulable en el tiempo.
24. Proceso según reivindicación 1 donde la diversidad generada in vivo se limita al ARN que codifica para la región de interés, manteniendo estable el resto del genoma de la levadura.
25. Proceso según reivindicación 1 donde la RDRP que replica el ARN de interés puede estar modificada en su especificidad o tasa de error.
26. Proceso según reivindicación 1 donde las secuencias de empaquetamiento de los vectores que codifican para las VLPs y las RDRPs asociadas han sido delecionadas o sustituidas con mutaciones sinónimas.
27. Proceso según reivindicación 1 donde la diversidad es monitorizada genotípicamente por cualquier método descrito.
28. Proceso según reivindicación 1 donde la diversidad es monitorizada fenotípicamente por cualquier método descrito.
29. Proceso según reivindicación 1 donde el candidato es la levadura entera, lisada o cualquier fracción de ésta purificada o secretada que contenga la diversidad generada.
30. Proceso según reivindicación 1 donde el candidato se usa como componente de un ensayo inmunodiagnóstico.
31. Proceso según reivindicación 1 donde el candidato se usa para terapia.
32. Proceso según reivindicación 1 donde el candidato se usa como vacuna.
33. Proceso según reivindicación 1 donde la inmunización con el candidato provoca una amplia respuesta inmune contra cepas divergentes del patógeno sobre el que está basado.
34. Proceso según reivindicación 1 donde el candidato se administra por cualquier ruta.
Patentes similares o relacionadas:
 Métodos para producir proteínas bicatenarias en bacterias, del 29 de Julio de 2020, de GENENTECH, INC.: Un método para producir un receptor de linfocitos T monoclonal de movilización inmunitaria contra el cáncer (ImmTAC) que comprende una cadena alfa del receptor de linfocitos […]
Métodos para producir proteínas bicatenarias en bacterias, del 29 de Julio de 2020, de GENENTECH, INC.: Un método para producir un receptor de linfocitos T monoclonal de movilización inmunitaria contra el cáncer (ImmTAC) que comprende una cadena alfa del receptor de linfocitos […]
Anticuerpo biespecífico o mezcla de anticuerpos con cadenas ligeras comunes, del 15 de Julio de 2020, de Jiangsu Alphamab Biopharmaceuticals Co., Ltd: Anticuerpo biespecífico o parte de unión a antígeno del mismo, en el que el anticuerpo biespecífico o la parte de unión a antígeno del mismo tiene una cadena […]
Vacunas y diagnóstico de torque teno virus porcino, del 18 de Junio de 2020, de VIRGINIA TECH INTELLECTUAL PROPERTIES, INC.: Composición inmunogénica que comprende una proteína según SEQ ID NO. 16.
Producción de FDCA catalizada por deshidrogenasa, del 17 de Junio de 2020, de PURAC BIOCHEM B.V.: Proceso para oxidar ácido 5-hidroximetil-2-furancarboxílico (HMFCA) a ácido 5-formil-2-furoico (FFA), donde el proceso comprende la etapa de incubar una […]
Vectores de AAV dirigidos a oligodendrocitos, del 10 de Junio de 2020, de THE UNIVERSITY OF NORTH CAROLINA AT CHAPEL HILL: Un ácido nucleico que codifica una cápside de AAV, comprendiendo el ácido nucleico una secuencia codificante de la cápside de AAV que es al menos el 96 % idéntica […]
Ácido nucleico que comprende o codifica para un tallo-bucle de histona y una secuencia poli(A) o una señal de poliadenilación para aumentar la expresión de un antígeno patogénico codificado, del 3 de Junio de 2020, de CureVac AG: Secuencia de ácido nucleico que comprende o codifica en la dirección 5' → 3' para: i) • una región codificante, que codifica para al menos […]
Microorganismo con productividad de l-lisina aumentada y procedimiento para producir l-lisina utilizando el mismo, del 27 de Mayo de 2020, de CJ CHEILJEDANG CORPORATION: Una subunidad beta prima (subunidad-β') mutante de la ARN polimerasa, en la que la subunidad beta prima (subunidad-β') mutante de la ARN polimerasa tiene […]
Agrupación de genes de biosíntesis de carrimicina, del 27 de Mayo de 2020, de Shenyang Fuyang Pharmaceutical Technology Co., Ltd: Agrupación de genes de biosíntesis de carrimicina, que consiste en 44 genes que comprende: 1) cinco genes de policétido sintasa, incluyendo los residuos […]