PROCEDIMIENTO DE ELIMINACIÓN DE SECUENCIAS DE GENES SELECCIONABLES.
Un proceso para la integración de ácido nucleico no marcado en el cromosoma de una célula procariota que comprende:
a) introducir un casete de ADN lineal en una célula, en el que dicho casete de ADN lineal comprende: i) un gen marcador de selección; ii) dos sitios de reconocimiento de recombinasas específicas de sitio de tipo dif que flanquean a dicho gen marcador de selección; y iii) dos regiones que flanquean dichos sitios de reconocimiento de recombinasas específicas de sitio de tipo dif que son homólogas a las dos regiones que flanquean el sitio de integración en el cromosoma de de la célula; b) cultivar dicha célula en condiciones de manera que el casete de ADN lineal se integre en el cromosoma celular por recombinación homóloga; y c) cultivar dicha célula en condiciones de manera que una recombinasa endógena específica de sitio, presente en el cromosoma de la célula, actúe para escindir el gen marcador de selección por recombinación específica de sitio entre los sitios de reconocimiento de recombinasas específicas de sitio de tipo dif
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2005/002590.
Solicitante: RPH PHARMACEUTICALS AB.
Nacionalidad solicitante: Suecia.
Dirección: LAGERVÄGEN, 7 136 50 HANINGE SUECIA.
Inventor/es: CRANENBURGH,Rocky MarcCobra Biologics Limited , BLOOR,Alexandra ElizabethCobra Biologics Limited.
Fecha de Publicación: .
Fecha Solicitud PCT: 1 de Julio de 2005.
Clasificación Internacional de Patentes:
- C12N15/867T
Clasificación PCT:
- C12N15/90 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Introducción estable de ADN extraño en el cromosoma.
Clasificación antigua:
- C12N15/90 C12N 15/00 […] › Introducción estable de ADN extraño en el cromosoma.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia.
Fragmento de la descripción:
CAMPO DE LA INVENCIÓN
La invención se refiere a un proceso para la eliminación de secuencias de genes marcadores de selección, en particular secuencias de genes de antibióticos, de moléculas de ácido nucleico. La invención se refiere 5 adicionalmente a la aplicación de este proceso en la integración y deleción de genes cromosómicos no marcados y en el control de la expresión génica.
ANTECEDENTES DE LA INVENCIÓN
Los genes de resistencia a antibióticos u otros genes marcadores de selección se usan rutinariamente para seleccionar la inserción cromosómica de genes heterólogos o la delección de genes nativos para crear nuevas 10 cepas de bacterias. Estos genes marcadores de selección también se usan rutinariamente para seleccionar la presencia de plásmidos en células bacterianas. Sin embargo, la conservación de genes marcadores de selección en células hospedadoras bacterianas, tanto integrados en el cromosoma como presentes en los plásmidos, causa varios problemas.
En primer lugar, la presencia de genes marcadores de selección en los cromosomas de hospedadores 15 reduce la diversidad de plásmidos que pueden propagarse en una célula, ya que estos también dependen de genes marcadores de selección para su selección y conservación. Adicionalmente, en la producción de agentes bioterapéuticos no se desean bacterias modificadas genéticamente que contengan genes de resistencia a antibióticos en los cromosomas, en particular para vacunas de ADN y en aplicaciones de terapia génica, ya que el ADN cromosómico representará un nivel contaminante bajo del producto final y conllevará el riesgo de transferir, al 20 paciente o al ambiente, el gen de resistencia a antibióticos mediante bacterias patógenas. Tampoco se desean genes de resistencia a antibióticos en plásmidos, ya que constituyen una carga metabólica en la producción de proteínas recombinantes y una preocupación en cuanto a bioseguridad cuando se está fabricando el plásmido para su uso en terapia génica y aplicaciones de vacunas de ADN.
Por lo tanto es muy deseable poder insertar o delecionar genes de cromosomas de células bacterianas sin 25 dejar atrás genes de resistencia a antibióticos u otros marcadores de selección y poder eliminar de los plásmidos, estos genes marcadores de selección, cuando ya no sean necesarios. Hasta ahora, se han desarrollado un par de estrategias para insertar y delecionar genes cromosómicos no marcados (es decir sin gen marcador de selección) en células bacterianas y para delecionar genes marcadores de plásmidos.
Una estrategia para la inserción y delección de genes no marcados se basa en integrar un plásmido, que 30 contiene un gen marcador de selección, en el cromosoma de una célula hospedadora bacteriana, mediante un solo evento de recombinación homóloga, seguido de la eliminación del plásmido por un segundo evento de recombinación (resolución) para producir de manera esperada el genotipo deseado (Leenhouts et al., 1996; Link et al., 1997). Una desventaja principal de esta estrategia es que si la inserción o la deleción reducen la eficacia biológica de la célula, en cuanto a su capacidad de supervivencia, el evento de resolución regenerará 35 invariablemente el tipo silvestre en lugar del genotipo mutante. Por lo tanto esta estrategia es muy ineficaz.
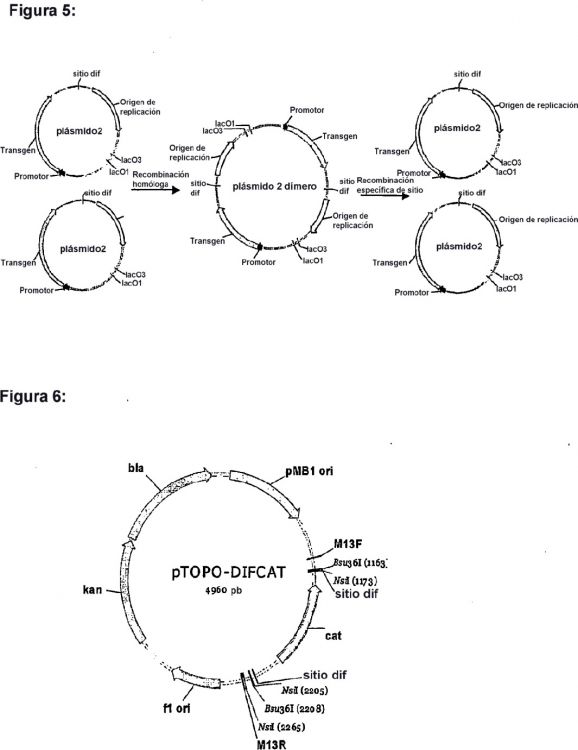
Una estrategia alternativa es usar un doble evento de recombinación para integrar eficazmente un casete génico de resistencia a antibiótico flanqueado por regiones de homología cromosómica en el cromosoma bacteriano. Los sitios de reconocimiento de una recombinasa específica de sitio (SSR) flanquean directamente al gen de resistencia a antibiótico. Después de la integración cromosómica, una recombinasa expresada en trans escinde el 40 gen de resistencia a antibiótico. Como ejemplos de sitios de recombinasas/dianas específicas de sitio usados para escindir el gen de resistencia a antibiótico se incluyen Cre/IoxP del bacteriófago P1 (Dale y Ow, 1991), FLP/FRT (Datsenko y Wanner, 2000) y R/RS (Sugita et al., 2000) de levaduras. De manera alternativa, el flanqueo de genes de resistencia a antibióticos con sitios de resolución internos permite la escisión por una transposasa expresada en trans (Sanchis et al., 1997). La desventaja de esta estrategia es que esto requiere la expresión de una recombinasa 45 o transposasa exógena, en la célula diana. Por lo tanto la célula debe transformarse dos veces.
Actualmente, los genes marcadores de selección también se eliminan de plásmidos usando recombinasas específicas de sitio como se describe para las aplicaciones cromosómicas anteriores, o más comúnmente, por digestión con endonucleasas de restricción. La estrategia con recombinasas requiere un gen adicional de recombinasa específica de sitio presente en cis o en trans en un plásmido auxiliar. La estrategia de digestión 50 mediante restricción requiere varias fases extra de manipulación del ADN del plásmido. Estas dos estrategias implican varias manipulaciones complejas.
Dada la creciente importancia en cuanto a la generación de moléculas de ácido nucleico sin genes marcadores de selección, en particular genes de resistencia a antibióticos, existe una necesidad de desarrollar procesos mejorados y más sencillos para la inserción y deleción de genes no marcados y para eliminar de plásmidos 55 genes marcadores de selección.
El documento WO01/18222 describe un método para la integración de ADN heterólogo en Escherichia coli con eliminación concreta de marcadores y replicones usados durante la construcción. Entre las secuencias a integrar se insertan secuencias de replicación y marcadores de resistencia entre dos sitios para una recombinasa específica de sitio. Mediante la expresión de la recombinasa éstos consiguen escindirse del cromosoma en el que están integradas las secuencias. El método se ilustra con FRT/FLP y otros sistemas tales como también dif/Xer. La 5 recombinasa se expresa a partir del plásmido, no se menciona otra forma de hacerlo. Por lo tanto D1 no anticipa innovación de ninguna de las reivindicaciones.
Barre et al., describen el análisis de la función de FtsK en la resolución del plásmido en E. coli mediante XerC y XerD. Los plásmidos indicadores, basados en pUC18 o pGB2 que comprenden dos sitios dif de reconocimiento de recombinasas específicas de sitio (por ejemplo, también la resistencia a antibióticos está dentro 10 de estos dos sitios) se resuelven en dos (plásmidos) in vivo. También se resuelve in vivo (por ejemplo se escinde) un casete de resistencia a Km insertado cromosómicamente flanqueado por dos sitios dif en una cepa que es cromosómicamente XerC y en la que la XerC endógena se expresa a partir del plásmido. Por lo tanto, se considera que D2 no anticipa innovación de ninguna de las reivindicaciones que requieran que la recombinasa esté presente en el cromosoma de las células. 15
Recchia et a., describen el análisis de la función de FtsK en la resolución del plásmido en E. coli mediante XerC y XerD. Los genes indicadores, basados en pUC18 o pBR322 que comprenden dos sitios dif, cer, psi o dib de reconocimiento de recombinasas específicas de sitio se resuelven en dos (plásmidos) en cepas RecA+ y RecA- in vivo en cepas XerC+. Además uno de los plásmidos que se resuelve de esta manera es pSDC124 en una cepa XerC+ WT (por ejemplo XerC en el cromosoma; Fig. 1) o en una cepa Xer+ en la que la XerC cromosómica se 20 expresa a partir del promotor WT p/ac (Fig. 2). Del protocolo de construcción en D8 (página 796, columna derecha, tercer párrafo; véase también la Tabla II o D3) es evidente que pSDC124 posee dos repeticiones directas de un sitio dit, y entre ellos se unen el gen de resistencia a kanamicina de pUC4K. Este fragmento KmR de 1,3 kb se escinde después de la resolución del plásmido mediante recombinación mediada por XerC, produciendo el plásmido pMIN33 (producto de resolución) y un círculo no-replicón de 1,3 kb que comprende el KmR. Por lo tanto la resolución de este 25 plásmido pSDC124 en una cepa WT con XerC sobre el cromosoma (D3, Fig. 1; y D8, Fig. 5) o en una cepa con una XerC cromosómica inducible (D3, Fig. 2) conduce a un producto de resolución (RP) que es pMIN33 y que carece de un gen de resistencia a kanamicina presente en el plásmido original pSDC124 y observado después de la recombinación en el círculo no-replicón. Por lo tanto D3 y D8 proporcionan, en una célula procariota, un proceso de eliminación de un marcador de selección, insertado entre dos sitios (de tipo) dif, a partir de un plásmido mediante 30 una recombinasa endógena...
Reivindicaciones:
1. Un proceso para la integración de ácido nucleico no marcado en el cromosoma de una célula procariota que comprende:
a) introducir un casete de ADN lineal en una célula, en el que dicho casete de ADN lineal comprende:
i) un gen marcador de selección;
ii) dos sitios de reconocimiento de recombinasas específicas de sitio de tipo dif que flanquean a dicho gen 5 marcador de selección; y
iii) dos regiones que flanquean dichos sitios de reconocimiento de recombinasas específicas de sitio de tipo dif que son homólogas a las dos regiones que flanquean el sitio de integración en el cromosoma de de la célula;
b) cultivar dicha célula en condiciones de manera que el casete de ADN lineal se integre en el cromosoma 10 celular por recombinación homóloga; y
c) cultivar dicha célula en condiciones de manera que una recombinasa endógena específica de sitio, presente en el cromosoma de la célula, actúe para escindir el gen marcador de selección por recombinación específica de sitio entre los sitios de reconocimiento de recombinasas específicas de sitio de tipo dif.
2. Un proceso de acuerdo con la reivindicación 1 en el que la célula es una célula bacteriana. 15
3. Un proceso de acuerdo con la reivindicación 2 en el que la célula es una célula bacteriana gram negativa.
4. Un proceso de acuerdo con la reivindicación 2 en el que la célula es una célula bacteriana gram positiva.
5. Un proceso de acuerdo con la reivindicación 3 en el que la célula es una célula de E. coli.
6. Un proceso de acuerdo con la reivindicación 4 en el que la célula es una célula de B. subtilis.
7. Un proceso de acuerdo con una cualquiera de las reivindicaciones 2 a 6 en el que la célula es una célula 20 RecA+.
8. Un proceso de acuerdo con una cualquiera de las reivindicaciones 1 a 7 en el que los sitios de reconocimiento de recombinasas específicas de sitio de tipo dif que flanquean al gen marcador de selección son iguales.
9. Un proceso de acuerdo con una cualquiera de las reivindicaciones 1 a 7 en el que los sitios de 25 reconocimiento de recombinasas específicas de sitio de tipo dif que flanquean al gen marcador de selección son diferentes.
10. Un proceso de acuerdo con una cualquiera de las reivindicaciones 1 a 9 en el que al menos uno de los sitios de tipo dif se selecciona del sitio dif de E. coli y del sitio dif de B. subtilis.
11. Un proceso de acuerdo con una cualquiera de las reivindicaciones 1 a 10 en el que al menos uno de los 30 sitios de tipo dif se selecciona de los sitios de tipo dif, cer y psi del plásmido y la molécula de ácido nucleico comprende adicionalmente secuencias accesorias.
12. Un proceso de acuerdo con una cualquiera de las reivindicaciones 1 a 11 en el que al menos uno de los sitios de tipo dif es el sitio híbrido de tipo dif, pif y la molécula de ácido nucleico comprende adicionalmente secuencias accesorias. 35
13. Un proceso de acuerdo con una cualquiera de las reivindicaciones 1 a 12 en el que el gen marcador de selección es un gen de resistencia a antibiótico.
14. Un proceso de acuerdo con una cualquiera de las reivindicaciones 1 a 13 para la delección de genes no marcados en el que las dos regiones que flanquean a dichos sitios de reconocimiento de recombinasas específicas de sitio de tipo dif son homólogas a las dos regiones que flanquean al gen a delecionar en el cromosoma de la 40 célula.
15. Un proceso de acuerdo con una cualquiera de las reivindicaciones 1 a 13 para la integración de genes no marcados en el que las dos regiones que flanquean a los sitios de reconocimiento de recombinasas específicas de sitio de tipo dif son homólogas a las dos regiones que flanquean al sitio de integración y el casete de ADN lineal comprende adicionalmente el gen exógeno a integrar, siempre que el gen exógeno no se localice entre los dos sitios 45 de reconocimiento de recombinasas específicas de sitio de tipo dif.
16. Un proceso de una cualquiera de las reivindicaciones 1 a 15 en el que la etapa b) comprende
adicionalmente cultivar la célula en presencia de una presión selectiva en el gen marcador de selección.
17. El proceso de acuerdo con la reivindicación 16 en el que la etapa c) comprende cultivar la célula en ausencia de cualquier presión selectiva.
18. Un proceso de acuerdo con una cualquiera de las reivindicaciones 1 a 17 en el que la molécula de ácido nucleico comprende adicionalmente un gen para selección positiva de células en las que se ha producido la 5 recombinación.
19. Un proceso para controlar la expresión de un gen de interés que comprende cultivar una célula procariota que comprende:
i) una primera molécula de ácido nucleico que comprende un gen de interés que está funcionalmente asociado a un operador; y 10
ii) una segunda molécula de ácido nucleico que comprende un gen marcador de selección y un gen represor flanqueados por sitios de reconocimiento de recombinasas específicas de sitio de tipo dif, en el que dicho represor es susceptible de unirse a dicho operador
en condiciones de manera que una recombinasa endógena específica de sitio presente en el cromosoma de la célula actúa para escindir el gen marcador de selección y dicho gen represor por recombinación específica de sitio 15 entre los sitios de reconocimiento de recombinasas específicas de sitio de tipo dif permitiendo de esta manera la expresión de un gen de interés.
20. Un proceso de acuerdo con la reivindicación 19 que comprende las características de una cualquiera de las reivindicaciones 2 a 13.
21. Un proceso de acuerdo con la reivindicación 19 o con la reivindicación 20 en el que la primera y segunda 20 molécula de ácido nucleico son casetes de ADN lineal integrados en el cromosoma de la célula.
22. Un proceso de acuerdo con la reivindicación 19 o con la reivindicación 20 en el que la primera y segunda molécula de ácido nucleicos son plásmidos.
23. Un proceso de acuerdo con la reivindicación 19 o con la reivindicación 20 en el que la primera molécula de ácido nucleico es un casete de ADN lineal integrado en el cromosoma de la célula y la segunda molécula de ácido 25 nucleico es un plásmido.
24. Un proceso de acuerdo con la reivindicación 19 o con la reivindicación 20 en el que la primera molécula de ácido nucleico es un plásmido y la segunda molécula de ácido nucleico es un casete de ADN lineal integrado en el cromosoma de la célula.
25. Un proceso para controlar la expresión de un gen de interés que comprende cultivar una célula procariota 30 que comprende una molécula de ácido nucleico que comprende
i) un gen de interés unido funcionalmente a un promotor; y
II) un gen marcador de selección y un terminador de la transcripción flanqueados por sitios de reconocimiento de recombinasas específicas de sitio de tipo dif, en el que dicho gen marcador de selección y terminador de la transcripción flanqueados por sitios de reconocimiento de recombinasas específicas de sitio de tipo dif se 35 localizan entre el gen de interés y el promotor de control de expresión de dicho gen de interés
en condiciones de manera que una recombinasa endógena específica de sitio presente, en el cromosoma de la célula, actúa para escindir, por recombinación específica de sitio, el gen marcador de selección y dicho terminador de la transcripción, entre los sitios de reconocimiento de recombinasas específicas de sitio de tipo dif, permitiendo de esta manera la expresión de un gen de interés. 40
26. Un proceso de acuerdo con la reivindicación 25 que comprende las características de una cualquiera de las reivindicaciones 2 a 13.
27. Un proceso de acuerdo con la reivindicación 25 o con la reivindicación 26 en el que la molécula de ácido nucleico es un plásmido o es un casete de ADN lineal integrado en el cromosoma.
M. Escalera de ADN Kb
1. Control negativo (sin ADN)
2. Tipo silvestre (ADNg DH1)
3. ADNg integrante (DH1::msbB-DifCAT)
4. 7. Clones resolutivos (DH1msbB)
M. Escalera de ADN Kb
1. Control negativo (sin ADN)
2. Tipo silvestre (ADNg DH1)
3. ADNg integrante (DH1::rbpA-DifCAT)
4. 7. Clones resolutivos (DH1lacdapD-rbpA)
Patentes similares o relacionadas:
MODULACIÓN DE LA PRODUCCIÓN DE RETROVIRUS POR APOBEC4, del 23 de Diciembre de 2011, de Bundesrepublik Deutschland, letztvertreten durch den Präsidenten des Paul-Ehrlich-Instituts Prof. Dr. Johannes Löwer: Una proteína APOBEC4 humana modificada caracterizada porque el extremo N está modificado para estabilizar la proteína APOBEC4 o el extremo C […]
 Animales no humanos que tienen un locus de cadena ligera lambda de inmunoglobulina modificado por ingeniería, del 29 de Julio de 2020, de REGENERON PHARMACEUTICALS, INC.: Un roedor cuyo genoma de la línea germinal comprende un locus de cadena ligera λ de inmunoglobulina endógeno que comprende:
(a) uno o más segmentos […]
Animales no humanos que tienen un locus de cadena ligera lambda de inmunoglobulina modificado por ingeniería, del 29 de Julio de 2020, de REGENERON PHARMACEUTICALS, INC.: Un roedor cuyo genoma de la línea germinal comprende un locus de cadena ligera λ de inmunoglobulina endógeno que comprende:
(a) uno o más segmentos […]
Métodos y composiciones para el tratamiento de enfermedades por almacenamiento lisosomal, del 27 de Mayo de 2020, de Sangamo Therapeutics, Inc: Uno o más transgenes y una o más nucleasas con dedos de zinc (ZFN) para su uso en un método de tratamiento de una enfermedad por almacenamiento lisosomal, el método […]
Terapias de aumento génico de la degeneración retiniana causada por mutaciones en el gen PRPF31, del 6 de Mayo de 2020, de MASSACHUSETTS EYE & EAR INFIRMARY: Un vector de virus adeno-asociado de tipo 2 (AAV2) que comprende una secuencia que codifica PRPF31 humano, operativamente enlazado a un promotor […]
Proteínas que tienen actividad nucleasa, proteínas de fusión y usos de estas, del 18 de Marzo de 2020, de HELMHOLTZ ZENTRUM MUNCHEN DEUTSCHES FORSCHUNGSZENTRUM FUR GESUNDHEIT UND UMWELT (GMBH): Una molécula de ácido nucleico que codifica (I) un polipéptido que tiene la actividad de una endonucleasa, que es (a) una molécula de ácido nucleico que […]
Animales no humanos que tienen una interrupción en un locus C9ORF72, del 19 de Febrero de 2020, de REGENERON PHARMACEUTICALS, INC.: Un roedor que comprende en su genoma una deleción de la porción codificante del exón 2 hasta la porción codificante del exón 11 de un locus C9orf72 endógeno, […]
Métodos y composiciones para modificar un locus objetivo, del 12 de Febrero de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para la modificación en serie de un locus objetivo en una célula, que comprende: (a) proporcionar la célula que comprende el locus objetivo, en donde el locus objetivo […]
Modelo de cerdo para la diabetes, del 29 de Enero de 2020, de AARHUS UNIVERSITET: Un cerdo transgénico que comprende un gen de polipéptido amiloide de los islotes (IAPP) mutado humano o parte del mismo, y que muestra al menos […]