RELACIONANDO AGREGACIÓN PROTEICA CON SUPERVIVENCIA DE LEVADURA.
Relacionando agregación proteica con supervivencia de levadura.
La invención se refiere a métodos para la identificación de compuestos capaces de prevenir la agregación de polipéptidos propensos a la agregación basados en el uso de proteínas de fusión. Las proteínas de fusión comprenden la proteína de interés y una enzima que es capaz de metabolizar un agente tóxico para la célula. Si el compuesto candidato previene la agregación de la proteína de fusión, el enzima se mantiene soluble y en forma activa, evitando así el efecto tóxico en la célula e incrementando la viabilidad celular respecto a la de células no expuestas al compuesto candidato. La invención también proporciona diferentes polipéptidos que contienen un polipéptido con tendencia a agregar y una enzima, y describe la utilización de los mismos.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2009/063213.
Solicitante: UNIVERSITAT AUTONOMA DE BARCELONA.
Nacionalidad solicitante: España.
Inventor/es: VENTURA ZAMORA,Salvador, AVILÉS PUIGVERT,Francesc Xavier, MORELL FERNÁNDEZ,Montserrat.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- G01N33/542 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › con inhibición estérica o modificación de la señal, p. ej. extinción de fluorescencia.
- G01N33/68 G01N 33/00 […] › en los que intervienen proteínas, péptidos o aminoácidos.
PDF original: ES-2418459_A2.pdf
Fragmento de la descripción:
RELACIONANDO AGREGACIÓN PROTEICA CON SUPERVIVENCIA DE LEVADURA
OBJETO DE LA INVENCIÓN
En la presente invención se relaciona la agregación proteica con la supervivencia de levaduras.
ANTECEDENTES DE LA INVENCIÓN
En los últimos años, la agregación de proteínas ha tomado una importancia creciente y es uno de los tópicos más estudiados en biología y medicina. Una gran cantidad de estudios sugieren que el mal plegamiento de proteínas y la formación de agregados amiloides insolubles es la causa fundamental de algunas enfermedades como el Alzheimer (A) , el Parkinson (P) , la diabetes tipo II y las encefalopatías espongiformes entre otras (Dobson, C.M. Protein-misfolding diseases: Getting out of shape. Nature 418, 729 -730 (2002) ) . Un punto en común de estas enfermedades es la presencia de depósitos de agregados proteicos en los órganos internos que interfieren con la función celular normal, algunas veces de manera letal. Además, la agregación proteica es uno de los mayores problemas en la expresión de proteínas, reduciendo el número de proteínas que pueden ser obtenidas de manera recombinante y dificultando la investigación en áreas como la genómica estructural y la proteómica. Por lo tanto, existe un gran interés en el desarrollo de métodos que detecten la agregación proteica y que permitan la selección de genes, compuestos químicos o condiciones de cultivo que permitan modular la agregación de proteínas.
Hasta la actualidad, diferentes ensayos han sido desarrollados para estudiar y detectar la agregación proteica en células procariotas, específicamente en Escherichia coli (Maxwell, K.L., Mittermaier, A.K., Forman-Kay, J.D. & Davidson, A.R. A simple in vivo assay for increased protein solubility, Protein Sci 8, 1908-1911 (1999) ) , (Waldo, G.S., Standish, B.M., Berendzen, J. & Terwilliger, T.C. Rapid protein-folding assay using green fluorescent protein. Nat Biotechnol 17, 691-695 (1999) ) , (Wigley, W.C., Stidham, R.D., Smith, N.M., Hunt, J.F. & Thomas, P.J. Protein solubility and folding monitored in vivo by structural complementation of a genetic marker protein. Nat Biotechnol 19, 131-136 (2001) ) . La mayoría de estos métodos se basan en la capacidad de las proteínas que agregan de causar el mal plegamiento de otra proteína marcador fusionada a ellas. De esta manera, diferentes proteínas han sido usadas como marcadores, permitiendo la detección de la agregación proteica utilizando diferentes señales como resistencia a ciertos antibióticos o la actividad !-galactosidasa.
Otra aproximación ha sido el uso de métodos basados en la detección de fluorescencia. Uno de los primeros en ser desarrollados utilizaba la proteína verde fluorescente (del inglés Green Flourescent protein, GFP) como proteína de fusión (Waldo, G.S., Standish, B.M., Berendzen, J. & Terwilliger, T.C. Rapid protein-folding assay using green fluorescent protein. Nat Biotechnol 17, 691-695 (1999) ) . Esta técnica está basada en la existencia de una correlación de la fluorescencia detectada con la tendencia a agregar de la proteína fusionada en el extremo N-terminal de la GFP. Fusiones con proteínas que no agregan permiten un correcto plegamiento de la GFP. Por lo tanto, las colonias bacterianas que expresan estas fusiones exhiben fluorescencia verde. Por el contrario, la GFP fusionada a proteínas con tendencia a agregar no se pliega correctamente y la emisión de fluorescencia disminuye significativamente. Como ejemplo, el péptido amiloide Aβ42 se fusionó con la GFP y se expresó en bacterias. Después de mutar de manera aleatoria el péptido, mutaciones que reducen la tendencia del péptido Aβ42 resultaron en una mayor fluorescencie en bacterias que contenían estas fusiones.
La transferencia de energía por resonancia de la fluorescencia (FRET) ha sido también utilizada para crear un método in vivo para la detección del plegamiento proteico y de mutaciones que aumentan la estabilidad termodinámica de las proteínas en el citoplasma de E.coli (Philipps, B., Hennecke, J. & Glockshuber, R. FRET-based in vivo screening for protein folding and increased protein stability. J Mol Biol 327, 239-249 (2003) ) . El método se basa en la fusión de la proteína fluorescente verde (GFP) en el extremo C-terminal de la proteína X en estudio y la fusión de la proteína fluorescente azul (del inglés, Blue Fluorescent Protein BFP) en el extremo N-terminal. Una transferencia FRET eficiente entre la proteína BFP y GFP solo se observará in vivo si la proteína X está bien plegada y permite la aproximación de las dos proteínas fluorescentes.
Por el contrario, la señal se pierde si BFP y GFP están alejadas debido al mal plegamiento o degradación de la proteína X.
Otros métodos utilizan la actividad intracelular de la enzima β-galactosidasa como señal de solubilidad proteica o agregación. Este método es una adaptación del tradicional ensayo de complementación proteica (Wigley, W.C., Stidham, R.D., Smith, N.M., Hunt,
J.F. & Thomas, P.J. Protein solubility and folding monitored in vivo by structural complementation of a genetic marker protein. Nat Biotechnol 19, 131-136 (2001) ) . Cada monómero de la enzima tetramérica se puede dividir en dos fragmentos, el más pequeño (α) y el más grande (ω) . En presencia del fragmento α, los dímeros de los fragmentos ω se estabilizan y forman un tetrámero con actividad enzimática. Si la proteína de interés es fusionada al fragmento α, el reparto del fragmento α entre la fracción soluble e insoluble conllevará una reducción de la actividad β-galactosidasa, obteniendo información sobre la solubilidad de la proteína diana.
Otro método ha sido desarrollado después de la identificación de genes específicos que responden a la agregación proteica en bacterias. En este método el promotor del gen responsable de esta actividad (IbpAB) se fusiona a la proteína βgalactosidasa para cuantificar la respuesta del promotor al mal plegamiento intracelular de proteínas (Lesley, S.A., Graziano, J., Cho, C.Y., Knuth, M.W. & Klock, H.E. Gene expression response to misfolded protein as a screen for soluble recombinant protein. Protein Eng 15, 153-160 (2002) ) . De esta manera, la expresión de la proteína βgalactosidasa (y su actividad) se vincula a la agregación de la proteína diana dentro de la célula. Recientemente ha sido demostrada la utilidad de este método para evaluar los diferentes factores que influyen en la solubilidad proteica durante la expresión recombinante de proteínas en E.coli (Schultz, T., Martinez, L. & de Marco, A. The evaluation of the factors that cause aggregation during recombinant expression in E. coli is simplified by the employment of an aggregation-sensitive reporter. Microb Cell Fact 5, 28 (2006) )
La resistencia a cloramfenicol también ha sido usada para detectar mutantes solubles de una proteína con tendencia a la agregación en E. coli (Maxwell, K.L., Mittermaier, A.K., Forman-Kay, J.D. & Davidson, A.R. A simple in vivo assay for increased protein solubility. Protein Sci 8, 1908-1911 (1999) ) . En este caso, la proteína que ofrece la señal es la cloranfenicol acetiltransferasa (CAT) . La resistencia a altos niveles de cloramfenicol será equivalente a la expresión de una variante soluble de la proteína de interés. La selección se lleva a cabo creciendo las bacterias en un medio con una alta concentración de antibiótico.
Todos estos métodos se basan en el uso de bacterias y por lo tanto, sufren de las desventajas relacionadas con la expresión de proteínas heterólogas en bacterias.
WO2007/103788 describe un método para la determinación de la agregación proteica en levadura. Está basado en la capacidad del factor Sup35p para formar agregados amiloides. Este factor manifiesta un fenotipo priónico llamado [PSI+] y está formado por tres dominios. El dominio N-terminal (N) es dispensable para la viabilidad y es necesario y suficiente para las propiedades prionicas de Sup35p. La función el dominio intermedio (M) no está determinada y el dominio C-terminal (RF) lleva a cabo el fin de la traducción proteica y es esencial para la viabilidad.
En este método, la actividad del factor Sup35p (NMRF) se mide in vivo examinando la eficiencia con la que la síntesis proteica se termina en un codón de terminación stop prematuro. El ensayo usa el alelo ade1-14. Cepas que presentan esta mutación y que expresan el NMRF activo producen una versión truncada e inactiva de la proteína Ade1p, y como consecuencia, no pueden crecer en un medio sintético sin adenina (-Ade) , mientras que normalmente crecen en medio suplementado con adenina (+Ade) . Además, estas células acumulan un intermediario... [Seguir leyendo]
Reivindicaciones:
1. Un método para la identificación de compuestos que son capaces de disminuir la agregación de un polipéptido propenso a agregar que comprende:
(i) poner en contacto una o más células de levadura con un compuesto candidato, donde las células de levadura expresan una proteína de fusión formada por el polipéptido con tendencia a agregar y la dihidrofolato reductasa humana (hDHFR) bajo condiciones en las cuales el polipéptido promueve en parte la deposición de hDHFR, dando lugar a la perdida de la actividad intracelular de hDHFR y un incremento de la sensibilidad de las células de levadura al MTX,
(ii) añadir MTX o un inhibidor especifico de la DHFR a las células de levadura del paso (i) en una cantidad que, sin la presencia de la actividad de la enzima hDHFR, que forma parte de la proteína de fusión utilizada en el paso (i) , puede afectar negativamente la viabilidad de las células de levadura, y determinar la viabilidad de las células de levadura
donde un incremento de la viabilidad de las células respecto las células que no han sido expuestas al compuesto candidato indica que este compuesto es capaz de disminuir la agregación del polipéptido con tendencia a agregar.
2. Un método, según la reivindicación 1, donde el polipéptido con tendencia a agregar es un péptido amiloidogénico.
3. Un método según la reivindicación 2, donde el péptido amiloidogénico es seleccionado del grupo de Aβ42, un péptido que comprende una expansión de Poliglutaminas y una variante de α-sinucleína.
4. Método según cualquiera de las reivindicaciones anteriores, donde el compuesto que afecta negativamente a la viabilidad celular de la levadura es el metotrexato (MTX) .
5. Un método según cualquiera de las reivindicaciones anteriores, donde el compuesto que afecta negativamente la viabilidad de las células de levadura está marcado con fluorescencia.
6. Un método según la reivindicación 5, donde el paso (iii) además comprende la detección de la fluorescencia de las células de levadura, donde un aumento de la fluorescencia intracelular es indicativo de que el compuesto es capaz de disminuir la agregación del polipéptido.
7. Un método, según cualquiera de las reivindicaciones de la 1 a la 6, donde las células de levadura son seleccionadas entre el grupo formado por Saccharomyces cererevisiae, Saccharomyces uvae, Saccharomyces kluyveri, Schizosaccharomyces pombe, Saccharomyces uvarum, Kluyveromyces lactis, Hansenula polymorpha, Pichia pastoris, Pichia methanolica, Pichia kluyveri, Yarrowia lipolytica, Candida sp., Candida utilis, Candida cacaoi, Geotrichum sp. y Geotrichum fermentans
8. Método, según las reivindicaciones de la 1 a la 7, donde la membrana de la célula de levadura muestra un aumento de permeabilidad.
9. Método según la reivindicación 8, donde la cepa de la levadura tiene una mutación que inactiva el gen erg6.
10. Método, según cualquiera de las reivindicaciones anteriores, donde la célula de levadura lleva una mutación de inactivación en una o más de una chaperona molecular seleccionada entre los miembros de las siguientes familias: Hsp100, Hsp90, Hsp70, Hsp40 o de pequeñas proteínas de choque térmico.
11. Un método según la reivindicación 10 donde el miembro de la familia de proteínas Hsp100 es la Hsp104, el miembro de la familia Hsp90 es seleccionado del grupo Hsc82 y Hsp82, el miembro de la familia Hsp70 es seleccionado del grupo de Ssa1, Ssa2, Ssa3 y Ssa4, el miembro de la familia de Hsp40 es seleccionado del
grupo de Ydj1 y Sis1 y/o de las proteínas pequeñas de choque térmico se han seleccionado del grupo de Hsp26 y Hsp42.
12. Un polipéptido formado por un polipéptido con tendencia a agregar y un polipéptido con actividad enzimática donde el polipéptido con tendencia a agregar es un péptido amiloidogénico y el polipéptido con actividad enzimática es la DHFR y dicho polipéptido con actividad enzimática es capaz de modificar un compuesto, que afecta negativamente a la viabilidad celular de la levadura, a un metabolito de dicho compuesto con un efecto adverso reducido sobre dicha viabilidad celular.
13. Un polipéptido, según la reivindicación 12 donde el péptido amiloidogénico se ha seleccionado entre el grupo de Aβ42, un polipéptido que comprende una repetición de poli-glutamina y α-sinucleína o una variante de la misma.
14. Un polipéptido como se define en las reivindicaciones 12 o 13 donde la DHFR es la DHFR humana.
15. Un polipéptido como se define en cualquiera de las reivindicaciones de la 12 a la 14 formado por un polipéptido marcador.
16. Un polipéptido, según la reivindicación 15 donde el polipéptido marcador es una proteína fluorescente.
17. Un polipéptido, según la reivindicación 16 donde la proteína fluorescente es la GFP.
18. Un polinucleótido que codifica un polipéptido definido en cualquiera de las reivindicaciones de la 12 a la 17.
19. Un vector que comprende un polinucleótido según la reivindicación 18.
20. Una célula huésped que comprende un polipéptido definido en cualquiera de las reivindicaciones de la 12 a la 17, un polinucleótido definido en la reivindicación 18
o un vector definido en la 19.
21. Una célula huésped definida en la reivindicación 20, donde la célula huésped es una célula de levadura.
22. Una célula huésped, según la reivindicación 21, donde la célula de levadura es Saccharomyces cerevisiae.
23. Una célula huésped como se define en cualquiera de las reivindicaciones entre la 20 a la 22, donde la célula de levadura muestra una mayor permeabilidad de la membrana.
24. Una célula huésped según la reivindicación 23, donde lleva una mutación que inactiva el gen erg6.
25. Una célula huésped según las reivindicaciones de la 21 a la 24, donde la célula lleva una mutación de inactivación de una o más de una chaperona molecular seleccionadas a partir de un miembro de la familia de las proteínas Hsp100, un miembro de la familia Hsp90, de la familia de las proteínas Hsp70, de la familia de las proteínas Hsp40 o una proteína pequeña de choque térmico.
26. Una célula huésped según la reivindicación 25, donde el miembro de la familia de proteínas Hsp100 es la Hsp104, el miembro de la familia Hsp90 es seleccionado del grupo Hsc82 y Hsp82, el miembro de la familia Hsp70 es seleccionado del grupo de Ssa1, Ssa2, Ssa3 y Ssa4, el miembro de la familia de Hsp40 es seleccionado del grupo de Ydj1 y Sis1 y/o las proteínas pequeñas de choque térmico se han seleccionado del grupo de Hsp26 y Hsp42.
27. Una célula huésped como se define en cualquiera de las reivindicaciones de la 21 a la 26, el uso de la cual permite identificar compuestos capaces de inhibir la agregación de los polipéptidos con tendencia a agregar.
28. Método de detección de un compuesto que disminuye la agregación de polipéptidos propensos a la agregación, donde el método comprende (a) el compuesto candidato se ponga en contacto con una o más células de levadura, donde las células de levadura expresen una proteína de fusión formada por un polipéptido con tendencia a agregar y la dihidrofolato reductasa humana (hDHFR) la cual inhibe la toxicidad del compuesto metotrexato (MTX) , el cual afecta a la viabilidad de las células de levadura (b) la adición del compuesto tóxico para la célula de levadura en una cantidad que, sin la presencia de la enzima, afectaría a la viabilidad celular.
29. Método, según la reivindicación 28, donde el polipéptido con tendencia a agregar es un péptido amiloidogénico.
30. Método, según la reivindicación 29, donde el polipéptido amiloidogénico es el Aβ42.
31. Método, según la reivindicación 29, donde el péptidos amiloidogénico es una expansión de poliglutaminas.
32. Método, según la reivindicación 29, donde el péptido amiloidogénico es una variante de α-sinucleína.
33. Método, según cualquiera de las reivindicaciones de la 28 a la 32, donde la enzima es la dihidrofolato reductasa humana (h-DHFR) y el compuesto tóxico es el metotrexato (MTX) .
34. Método según cualquiera de las reivindicaciones de la 28 a la 33, donde el compuesto tóxico está marcado con fluorescencia.
35. Método según cualquiera de las reivindicaciones de la 28 a la 34, donde las células de levadura han sido seleccionadas del grupo formado por Saccharomyces uvae, Saccharomyces kluyveri, Schizosaccharomyces pombe, Saccharomyces uvarum, Kluyveromyces lactis, Hansenula polymorpha, Pichia pastoris, Pichia methanolica,
Pichia kluyveri, Yarrowia lipolytica, Candida sp., Candida utilis, Candida cacaoi, Geotrichum sp. y Geotrichum fermentans 36. Método, según las reivindicaciones de la 28 a la 34, donde la célula de levadura es Saccharomyces cererevisiae. 10
37. Método según cualquiera de las reivindicaciones anteriores de la 28 a la 36, donde la agregación de los péptido amiloidogénicos da lugar a un grupo de enfermedades seleccionadas del grupo formado por la enfermedad del Alzheimer, la enfermedad de Parkinson, polineuropatía amiloidea familiar, una taupatía, la enfermedad de los Trinucleótidos, las encefalopatías espongiformes transmisibles (EET) , la enfermedad de Alzheimer (EA) , y la enfermedad de Huntington (EH) .
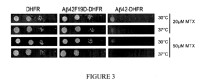
Figura 1
Figura 2
Figura 3
Figura 4
Figura 5
Figura 6
Figura 7
Figura 8
Figura 9
Figura 10
Figura 11
Figura 12
Figura 13
Figura 14
Figura 15
Patentes similares o relacionadas:
Inmunomoduladores, del 29 de Julio de 2020, de BRISTOL-MYERS SQUIBB COMPANY: Un compuesto de la fórmula (I) **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo, en donde: A se selecciona de **(Ver fórmula)** en donde: […]
Métodos y composiciones para el diagnóstico y pronóstico de lesión renal e insuficiencia renal, del 29 de Julio de 2020, de Astute Medical, Inc: Un método para evaluar el estado renal en un sujeto, que comprende: realizar una pluralidad de ensayos configurados para detectar una […]
Neuregulina para tratar la insuficiencia cardíaca, del 29 de Julio de 2020, de Zensun (Shanghai) Science & Technology, Co., Ltd: Neuregulina para usar en un método para tratar la insuficiencia cardíaca crónica en un paciente, donde el paciente tiene un nivel plasmático de NT-proBNP […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Método para llevar a cabo el seguimiento de la enfermedad de Gaucher, del 15 de Julio de 2020, de Centogene GmbH: Un método para determinar la evolución de la enfermedad de Gaucher en un sujeto, que comprende la etapa de determinar en varios puntos en el […]
Procedimiento para evaluación de la función hepática y el flujo sanguíneo portal, del 15 de Julio de 2020, de The Regents of the University of Colorado, a body corporate: Procedimiento in vitro para la estimación del flujo sanguíneo portal en un individuo a partir de una única muestra de sangre o suero, comprendiendo el procedimiento: […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos de monitorización terapéutica de profármacos de ácido fenilacético, del 24 de Junio de 2020, de Immedica Pharma AB: Glicerilo tri-[4-fenilbutirato] (HPN-100) para su uso en un método para tratar un trastorno del ciclo de la urea en un sujeto que tiene discapacidad […]