Evento DAS-59122-7 de maíz y métodos para su detección.
Una molécula de DNA aislada que comprende una secuencia de nucleótidos seleccionada de:
(a) la secuencia de nucleótidos descrita en la SEQ ID NO:23;
(b) la secuencia de nucleótidos descrita en la SEQ ID NO:21; y
(c) la secuencia de nucleótidos descrita en la SEQ ID NO:22.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2005/034947.
Solicitante: PIONEER HI-BRED INTERNATIONAL, INC..
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 7100 N.W. 62ND AVENUE P.O. BOX 1014 JOHNSTON, IA 50131-1014 ESTADOS UNIDOS DE AMERICA.
Inventor/es: ZHANG, JIAN, HAKIMI, SALIM, M., GUPTA,MANJU, BING,JAMES WAYNE, CRESSMAN,ROBERT F. JR, HONDRED,DAVID, KRONE,TODD L, HARTNETT LOCKE,MARY E, LUCKRING,ABIGAIL K, MEYER,SANDRA E, MOELLENBECK,DANIEL, NARVA,KENNETH EDWIN, OLSON,PAUL D, SANDERS,CRAIG D, WANG,JIMEI, ZHONG,GAN-YUAN.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/82 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › para células vegetales.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2432749_T3.pdf
Fragmento de la descripción:
Evento DAS-59122-7 de maíz y métodos para su detección Campo de la invención Las realizaciones de la presente invención se refieren al campo de la biología molecular de plantas, específicamente una realización de la invención se refiere a una construcción de DNA para conferir a una planta resistencia a los insectos. Las realizaciones de la invención se refieren más específicamente a una planta DAS59122-7 de maíz resistente a los insectos y a ensayos para detectar la presencia del DNA de la planta DAS-591227 de maíz en una muestra y en sus composiciones.
Antecedentes de la invención Una realización de esta invención se refiere al maíz resistente a los insectos, planta DAS-59122-7 (Zea mays) , también denominada línea DAS-59122-7 de maíz o evento DAS-59122-7 de maíz, y a la construcción de expresión en la planta del DNA de la planta DAS-59122-7 de maíz y a la detección de la región de inserción del transgén/flanqueante en la planta DAS-59122-7 de maíz y su progenie.
El maíz es un cultivo importante y es una fuente principal de alimento en muchas zonas del mundo. El daño causado por las plagas de insectos es un factor importante en la pérdida de los cultivos de maíz en el mundo, a pesar del uso de medidas protectora, tales como los plaguicidas químicos. En vista de esto, la resistencia a los insectos ha sido diseñada por ingeniería genética en cultivos, tales como el maíz con el fin de controlar el daño de los insectos y reducir la necesidad de los plaguicidas químicos tradicionales. Un grupo de genes que se han utilizado para la producción de cultivos transgénicos resistentes a los insectos son las delta-endotoxinas de Bacillus thuringiensis (B.t.) . Las delta-endotoxinas se han expresado con éxito en plantas de cultivo, tales como algodón, patatas, arroz, girasol, así como maíz y han demostrado proporcionar un excelente control sobre las plagas de insectos. (Perlak, F.J. et al., (1990) Bio/Technology 8, 939-943; Perlak, F.J. et al., (1993) Plant Mol. Biol. 22:313321; Fujimoto H. et al., (1993) Bio/Technology 11:1151-1155; Tu et al., (2000) Nature Biotechnology 18:1101-1104; publicación de patente PCT Nº WO 01/13731; y Bing JW et al., (2000) Efficacy of Cr y 1F Transgenic Maize, 14th Biennial International Plant Resistance to Insects Workshop, Fort Collins, CO) .
Se sabe que la expresión de genes extraños en plantas está influenciada por su localización en el genoma de la planta, debido tal vez a la estructura de la cromatina (por ejemplo, heterocromatina) o la proximidad de los elementos reguladores de la transcripción (por ejemplo, potenciadores) cerca del sitio de integración (Weising et al., Ann. Rev. Genet. 22:421-477, 1988) . Al mismo tiempo, la presencia del transgén en diferentes localizaciones en el genoma influirá de diferentes modos en el fenotipo global de la planta. Por esta razón, frecuentemente es necesario cribar un gran número de eventos con el fin de identificar un evento caracterizado por la expresión óptima de un gen de interés introducido. Por ejemplo, se ha observado en plantas y en otros organismos que puede haber una amplia variación entre eventos en los niveles de expresión de un gen introducido. También puede haber diferencias en los patrones de expresión espaciales o temporales, por ejemplo, diferencias en la expresión relativa de un transgén en diversos tejidos de la planta, que pueden no corresponder a los patrones esperados de los elementos reguladores de la transcripción presentes en la construcción del gen introducido. Por esta razón, es usual producir cientos a miles de diferentes eventos y cribar dichos eventos para obtener un solo evento que tenga los niveles y patrones deseados de expresión de transgenes para fines comerciales. Un evento que tiene los niveles o patrones deseados de la expresión de transgenes es útil para introgresar el transgén en otros antecedentes genéticos por cruzamiento sexual externo utilizando métodos de cultivo convencionales. La progenie de estos cruces mantiene las características de expresión del transgén del transformante original. Esta estrategia se utiliza para asegurar la expresión génica fiable en un número de variedades que están bien adaptadas a las condiciones locales de cultivo.
Sería ventajoso poder detectar la presencia de un evento particular con el fin de determinar si la progenie de un cruce sexual contiene un transgén de interés. Además, un método para detectar un evento particular sería de gran ayuda para el cumplimiento de las regulaciones que requieren la aprobación previa a la comercialización y el etiquetado de alimentos derivados de plantas de cultivos recombinantes, por ejemplo, o para su uso en la vigilancia ambiental, la vigilancia de rasgos en cultivos en el campo o la vigilancia de productos procedentes de una cosecha del cultivo, así como para su uso en asegurar el cumplimiento de las partes sometidas a los términos reglamentarios o contractuales.
Es posible detectar la presencia de un transgén por cualquier método de detección de ácidos nucleicos conocido en la técnica, incluyendo, aunque sin limitación, la reacción en cadena de la polimerasa (PCR) o hibridación de DNA usando sondas de ácidos nucleicos. Estos métodos de detección generalmente se centran en elementos genéticos utilizados con frecuencia, tales como promotores, terminadores, genes marcadores, etc., debido a que en muchas construcciones de DNA, la región codificadora es intercambiable. Como resultado, dichos métodos pueden no ser útiles para discriminar entre diferentes eventos, en particular los producidos usando la misma construcción de DNA
o construcciones muy similares a menos que se conozca la secuencia de DNA del DNA flanqueante adyacente al DNA heterólogo insertado. Por ejemplo, un ensayo de PCR específico para el evento se describe en la Patente de EE.UU. Nº 6.395.485 para la detección del evento élite GAT-ZM1. En consecuencia, sería deseable disponer de un método sencillo y discriminatorio para la identificación del evento DAS-59122-7.
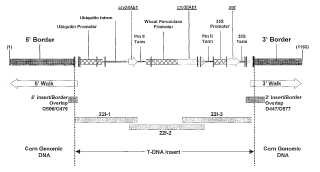
Sumario de la invención Las realizaciones de esta invención se refieren a métodos para producir y seleccionar una planta de cultivo monocotiledónea resistente a los insectos. Más específicamente, se proporciona una construcción de DNA que cuando se expresa en células vegetales y en plantas confiere resistencia a los insectos. De acuerdo con un aspecto de la invención, se proporciona una construcción de DNA, capaz de introducción y replicación en una célula hospedante, que cuando se expresa en células vegetales y en plantas confiere a las células vegetales y plantas resistencia a los insectos. La construcción de DNA está constituida por una molécula de DNA denominada PHI17662A e incluye tres (3) casetes de expresión del transgén. El primer casete de expresión comprende una molécula de DNA que incluye el promotor, el exón no traducido en 5' y el primer intrón del gen de la ubiquitina (Ubi1) de maíz (Christensen et al., (1992) Plant Mol. Biol., 18:675-689 y Christensen and Quail (1996) Transgenic Res. 5:213-218) conectada operativamente a una molécula de DNA que codifica una δ-endotoxina de B.t. identificada como Cr y 34Ab1 (Patentes de EE.UU. Nº 6.127.180, 6.624.145 y 6.340.593) conectada operativamente a una molécula de DNA que comprende un terminador de la transcripción Pin II aislado de patata (Gyheung An et al., (1989) Plant Cell. 1:115-122) . El segundo casete de expresión del transgén de la construcción de DNA comprende una molécula de DNA que codifica el promotor de peroxidasa de trigo (Hertig et al., (1991) Plant Mol. Biol.. 16:171174) conectada operativamente a una molécula de DNA que codifica una δ-endotoxina de β.t. identificada como Cr y 35Ab1 (Patente de EE.UU.. Nº 6.083.499, 6.548.291 y 6.340.593) conectada operativamente a una molécula de DNA que comprende un terminador de la transcripción Pin II aislado de patata (Gyheung An et al., (1989) Plant Cell. 1:115-122) . El tercer casete de expresión del transgén de la construcción de DNA comprende una molécula de DNA del promotor 35S del virus del mosaico de la coliflor (CaMV) (Odell J.T. et al., (1985) Nature 313:810-812; Mitsuhara et al., (1996) Plant Cell Physiol. 37:49-59) conectada operativamente a una molécula de DNA que codifica un gen de fosfinotricina-acetiltransferasa (PAT) (Wohlleben W. et al., (1988) Gene 70:25-37) conectada operativamente a una molécula de DNA que comprende una terminador de la transcripción en 3' del promotor 35S de CaMV (véase Mitsuhara et al., (1996) Plant Cell Physiol. 37:49-59) . También se proporcionan plantas que contienen la construcción del DNA.
De acuerdo con otra realización de la invención, se proporcionan composiciones y métodos para identificar una nueva planta de... [Seguir leyendo]
Reivindicaciones:
1. Una molécula de DNA aislada que comprende una secuencia de nucleótidos seleccionada de:
(a) la secuencia de nucleótidos descrita en la SEQ ID NO:23;
(b) la secuencia de nucleótidos descrita en la SEQ ID NO:21; y
(c) la secuencia de nucleótidos descrita en la SEQ ID NO:22.
2. Un kit para identificar el evento DAS-59122-7 en una muestra biológica que detecta una región específica de DAS-59122-7, comprendiendo dicho kit al menos un primer cebador, que reconoce una secuencia dentro de la SEQ ID NO:19 o dentro de la SEQ ID NO:20, y que comprende además al menos un segundo cebador que reconoce una secuencia de nucleótidos dentro de la secuencia SEQ ID NO:24 .
3. El kit de la reivindicación 2, en donde dichos al menos primero y segundo cebadores, respectivamente, comprenden un par de secuencias seleccionadas de:
(a) las secuencias SEQ ID NO:18 y SEQ ID NO:1;
(b) las secuencias SEQ ID NO:10 y SEQ ID NO:9;
(c) las secuencias SEQ ID NO:2 y SEQ ID NO:17;
(d) las secuencias SEQ ID NO:8 y SEQ ID NO:17; y
(e) las secuencias SEQ ID NO:36 y SEQ ID NO:37.
4. Un kit de detección de DNA específico para el DNA de unión del evento DAS-59122-7 de maíz y su progenie, que comprende al menos una molécula de DNA de una longitud suficiente de polinucleótidos de DNA contiguos para funcionar en un método de detección de DNA, en donde dicha molécula de DNA se hibrida en condiciones de hibridación rigurosas con una secuencia de DNA de unión del evento DAS-59122-7 de maíz, abarcando dicha secuencia de unión la unión entre el DNA heterólogo insertado en el genoma y el DNA flanqueante, y no se hibrida en dichas condiciones de hibridación rigurosas con un DNA de planta de maíz que no es DAS-59122-7 y dicha molécula de DNA es homóloga o complementaria de una secuencia seleccionada de:
(a) la secuencia de nucleótidos descrita en la SEQ ID NO:21; y
(b) la secuencia de nucleótidos descrita en la SEQ ID NO:22.
5. Un método para identificar el evento DAS-59122-7 en una muestra biológica, que comprende detectar una región específica de DAS-59122-7 por amplificación de un fragmento de DNA de un ácido nucleico presente en dicha muestra biológica usando una reacción en cadena de la polimerasa con al menos dos cebadores, en donde un primer cebador reconoce una secuencia dentro de la SEQ ID NO:19 o la SEQ ID NO:20, y un segundo cebador reconoce una secuencia dentro de la SEQ ID NO:24.
6. El método de la reivindicación 5, en donde dichos primero y segundo cebadores comprenden las secuencias SEQ ID NO:18 y SEQ ID NO:1, respectivamente.
7. El método de la reivindicación 5, en donde dichos primero y segundo cebadores comprenden las secuencias SEQ ID NO:10 y SEQ ID NO:9, respectivamente.
8. El método de la reivindicación 5, en donde dichos primero y segundo cebadores comprenden las secuencias SEQ ID NO:2 y SEQ ID NO:17, respectivamente.
9. El método de la reivindicación 5, en donde dichos primero y segundo cebadores comprenden las secuencias SEQ ID NO:8 y SEQ ID NO:17, respectivamente.
10. El método de la reivindicación 5, en donde dichos primero y segundo cebadores comprenden las secuencias SEQ ID NO:36 y la SEQ ID NO:37, respectivamente .
11. Un método para detectar la presencia del evento DAS-59122-7 de maíz o su progenie en una muestra biológica, que comprende:
(a) extraer una muestra de DNA de dicha muestra biológica;
(b) proporcionar un par de moléculas de cebadores de DNA seleccionado de:
(i) las secuencias SEQ ID NO:18 y SEQ ID NO:1;
(ii) las secuencias SEQ ID NO:10 y SEQ ID NO:9;
(iii) las secuencias SEQ ID NO:2 y SEQ ID NO:17; y
(iv) las secuencias SEQ ID NO:8 y SEQ ID NO:17;
(c) proporcionar condiciones de reacción de amplificación del DNA;
(d) realizar dicha reacción de amplificación de DNA, produciendo con ello una molécula de amplicón de DNA; y
(e) detectar dicha molécula de amplicón de DNA, en donde la detección de dicha molécula de amplicón de DNA en dicha reacción de amplificación de DNA indica la presencia del evento DAS-59122-7 de maíz.
12. Una molécula de DNA aislada que comprende uno cualquiera de los amplicones producidos por el método de la reivindicación 11.
13. Un método para detectar la presencia de DNA correspondiente al evento DAS-59122-7 en una muestra, comprendiendo el método:
(a) poner en contacto la muestra que comprende DNA de maíz con una sonda de polinucleótidos que se hibrida en condiciones de hibridación rigurosas con DNA de unión del evento DAS-59122-7 de maíz, dicho DNA de unión la unión entre el DNA heterólogo insertado en el genoma y el DNA flanqueante, y no se hibrida bajo dichas condiciones de hibridación rigurosas con un DNA de planta de maíz que no es DAS-59122-7 y dicha sonda es de una longitud suficiente de nucleótidos contiguos homólogos o complementarios de una secuencia seleccionada de:
(i) la secuencia de nucleótidos descrita en la SEQ ID NO:21; y
(ii) la secuencia de nucleótidos descrita en la SEQ ID NO:22;
(b) someter la muestra y la sonda a condiciones de hibridación rigurosas; y
(c) detectar la hibridación de la sonda con el DNA de unión, en donde la detección de la hibridación indica la presencia del evento DAS-5912-7.
14. Un par de moléculas de DNA que comprende:una primera molécula de DNA y una segunda molécula de DNA, en donde las moléculas de DNA tienen una longitud suficiente de nucleótidos contiguos de una secuencia seleccionada de:
(a) la secuencia descrita en la SEQ ID NO:21 o su complemento; y
(b) la secuencia descrita en la SEQ ID NO:22 o su complemento;
en donde el par de moléculas de DNA es capaz de amplificar, en una reacción en cadena de la polimerasa, un amplicón que comprende una región específica de DAS-59122-7 procedente del evento DAS-59122-7 de maíz y su progenie, en donde dicha región específica de DAS-59122-7 comprende al menos una secuencia de unión dentro de la SEQ ID NO:23, y dicha secuencia de unión abarca la unión entre el DNA heterólogo insertado en el genoma y el DNA flanqueante.
15. Un par de moléculas de DNA de la reivindicación 14, en donde cada molécula de DNA es un cebador que comprende una secuencia seleccionada de las SEQ ID NO:2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 36 y 37 o sus complementos.
16. Un par de moléculas de DNA de la reivindicación 15, en donde cada cebador comprende una secuencia seleccionada de las SEQ ID NO:2, 8, 9, 10, 17 y 18 o sus complementos.
17. Una molécula de DNA aislada que comprende una secuencia de unión que comprende una secuencia seleccionada de las SEQ ID NO:32, 33, 34 y 35 y sus complementos.
18. Un método para confirmar la pureza de semillas, que comprende la detección de una región específica de DAS59122-7 con un cebador o sonda específico que se hibrida específicamente en condiciones de hibridación rigurosas con una secuencia de unión dentro de la SEQ ID NO:21 o la SEQ ID NO:22, abarcando dicha secuencia de unión la unión entre el DNA heterólogo insertado en el genoma y el DNA flanqueante o su complemento, en una muestra de semillas, y no se hibrida en dichas condiciones de hibridación rigurosas con el DNA de una planta de maíz que no es no DAS-59122-7.
19. Un método para cribar semillas para detectar la presencia del evento DAS-59122-7, que comprende la detección de una región específica de DAS-59122-7 con un cebador o sonda específico que se hibrida específicamente en
condiciones de hibridación rigurosas con una secuencia de unión dentro de la SEQ ID NO:21 o la SEQ ID NO:22, abarcando dicha secuencia de unión la unión entre el DNA heterólogo insertado en el genoma y el DNA flanqueante o su complemento, en una muestra de un lote de semillas, y no se hibrida en dichas condiciones de hibridación rigurosas con el DNA de una planta que no es DAS-59122-7.
20. Una planta de maíz resistente a los insectos, en donde el DNA que comprende la secuencia de nucleótidos de la SEQ ID NO:23 forma parte del genoma de la planta.
21. Una planta descendiente de la planta de maíz resistente a los insectos de la reivindicación 20, en donde el DNA que comprende la secuencia de nucleótidos de la SEQ ID NO:23, forma parte del genoma de la planta.
22. Semilla de una planta de la reivindicación 20 o 21, en donde dicha semilla comprende la SEQ ID NO:23.
23. Un método para detectar la presencia de la inserción del evento DAS-59122-7 en el tejido de maíz que comprende:
(a) seleccionar un par de cebadores comprendiendo cada uno AL menos diez nucleótidos de la SEQ ID NO:21 o la SEQ ID NO:22, en donde cada miembro del par está en lados opuestos de una secuencia indicativa de la presencia de dicha inserción del evento DAS-59122-7, comprendiendo dicha secuencia al menos una secuencia de unión de DAS-59122-7;
(b) poner en contacto una muestra de dicho tejido de maíz con dicho par de cebadores; y
(c) realizar la amplificación del DNA y analizar los amplicones.
24. El método de la reivindicación 23, en donde cada cebador de dicho par se selecciona de las SEQ ID NO:1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 30, 36 y 37 o sus complementos.
25. Un método para detectar la presencia de la inserción del evento DAS-59122-7 en el tejido de maíz, que comprende:
(a) poner en contacto una muestra de dicho tejido de maíz con una sonda de polinucleótidos que se hibrida en condiciones de hibridación rigurosas con una o más secuencias de DNA seleccionadas de las SEQ ID NO:32, 33, 34 y 35 y sus complementos, en donde la sonda se hibrida específicamente con una región dentro de la región flanqueante en 5' o 3' del evento DAS-59122-7 y también comprende una parte del DNA extraño contiguo a dicha región;
(b) someter dichas muestra y sonda a condiciones de hibridación rigurosas; y
(c) analizar la hibridación de la sonda .
26. Un kit de detección de DNA que comprende una sonda de polinucleótidos que se hibrida en condiciones de hibridación rigurosas con una o más secuencias de DNA seleccionadas de las SEQ ID NO:32, 33, 34 y 35 y sus complementos, y en donde la sonda se hibrida específicamente con una región dentro de la región flanqueante en 5'
o 3' del evento DAS-59122-7 y comprende también una parte del DNA extraño contiguo a dicha región.
27. Un kit de detección de DNA que comprende un par de cebadores, comprendiendo cada cebador al menos 10 nucleótidos dentro de la SEQ ID NO:21 y la SEQ ID NO:22, en donde cada cebador está en los lados opuestos de una secuencia indicativa de la presencia de la inserción del evento DAS-59122-7, comprendiendo dicha secuencia al menos una secuencia de unión de DAS-59122-7, abarcando dicha secuencia de unión la unión entre el DNA heterólogo insertado en el genoma y el DNA flanqueante.
28. El kit de detección de DNA de la reivindicación 27, en donde cada cebador de dicho par se selecciona de las SEQ ID NO:1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 36 y 37 y sus complementos.
Patentes similares o relacionadas:
Composiciones para la supresión de la formación de inhibidores contra el factor VIII en pacientes con hemofilia A., del 22 de Julio de 2020, de THE TRUSTEES OF THE UNIVERSITY OF PENNSYLVANIA: Una composición que comprende material vegetal liofilizado que comprende al menos un fragmento de FVIII conjugado con la subunidad B de la toxina […]
Plantas con rendimiento incrementado y método de obtención de dichas plantas, del 15 de Julio de 2020, de CENTRE NATIONAL DE LA RECHERCHE SCIENTIFIQUE (CNRS): Método para mejorar por lo menos un carácter fenotípico seleccionado de entre el rendimiento en semillas, la velocidad de germinación o el crecimiento […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Moléculas de ácido nucleico de Nucampholin para controlar plagas de insectos coleópteros, del 17 de Junio de 2020, de FRAUNHOFER-GESELLSCHAFT ZUR FORDERUNG DER ANGEWANDTEN FORSCHUNG E.V.: Una molécula de ácido nucleico aislada que comprende un polinucleótido que codifica una molécula de ácido ribonucleico de horquilla (hpARN) con una estructura […]
Polinucleótidos aislados y métodos y plantas que usan los mismos para regular la acidez de las plantas, del 10 de Junio de 2020, de The State of Israel, Ministry of Agriculture and Rural Development, Agricultural Research Organization, (A.R.O.), Volcani Cent: Una célula de planta o una planta que comprende una construcción de ácido nucleico que comprende una secuencia de ácido nucleico que codifica un polipéptido […]
Genes de Chromobacterium subtsugae, del 27 de Mayo de 2020, de Marrone Bio Innovations, Inc: Una célula vegetal que comprende un polipéptido que tiene la secuencia de aminoácidos expuesta en las SEQ ID NO: 1, 2 o 3.
Moléculas polinucleotídicas para la regulación génica en plantas, del 6 de Mayo de 2020, de MONSANTO TECHNOLOGY, LLC: Un procedimiento para regular la expresión de un gen diana endógeno en plantas en crecimiento, que comprende: aplicar por vía tópica sobre […]
Producción de partículas similares al virus de la gripe en plantas, del 6 de Mayo de 2020, de MEDICAGO INC.: Un ácido nucleico que comprende una región reguladora activa en una planta y un potenciador de la expresión activo en una planta, la región […]