Método para medir la afinidad de biomoléculas.
Un método para medir la afinidad de primera y segunda biomoléculas en el que una primera biomolécula está unida por una primera parte de unión que tiene una longitud de primera parte de unión y una segunda biomolécula está unida por una segunda parte de unión que tiene una longitud de segunda parte de unión,
determinar la unión de primera y segunda biomoléculas adyacentes entre sí, variar al menos una de las longitudes de las primera y segunda partes de unión y determinar la unión de las primera y segunda biomoléculas, en el que las biomoléculas están unidas a un soporte y/o unidas entre sí.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2006/004208.
Solicitante: Nanotether Discovery Science Limited.
Nacionalidad solicitante: Reino Unido.
Dirección: 8th Floor Eastgate House, 35-43 Newport Road Cardiff CF24 0AB REINO UNIDO.
Inventor/es: DALE,TREVOR CLIVE, HARWOOD,ADRIAN JOHN, BORRI,PAOLA.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- G01N33/50 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › Análisis químico de material biológico, p. ej. de sangre o de orina; Ensayos mediante métodos en los que interviene la formación de uniones bioespecíficas con grupos coordinadores; Ensayos inmunológicos (procedimientos de medida o ensayos diferentes de los procedimientos inmunológicos en los que intervienen enzimas o microorganismos, composiciones o papeles reactivos a este efecto, procedimientos para preparar estas composiciones, procedimientos de control sensibles a las condiciones del medio en los procedimientos microbiológicos o enzimáticos C12Q).
- G01N33/53 G01N 33/00 […] › Ensayos inmunológicos; Ensayos en los que interviene la formación de uniones bioespecíficas; Materiales a este efecto.
- G01N33/543 G01N 33/00 […] › con un soporte insoluble para la inmovilización de compuestos inmunoquímicos.
PDF original: ES-2509344_T3.pdf




Fragmento de la descripción:
Método para medir la afinidad de biomoléculas Campo de la invención
La presente invención se refiere en particular, aunque no exclusivamente, al campo del análisis de las rutas bioquímicas y a la interacción entre biomoléculas tales como proteínas y polipéptidos.
Antecedentes de la invención
Ha habido una explosión de información biológica en los últimos años resultante del uso de técnicas experimentales de alto rendimiento. Estas técnicas varían de secuenciación del genoma, análisis de micromatrices, ensayos de interacciones proteína-proteína de 2 híbridos de levadura, hasta exploraciones de ARNi y el uso de análisis de imágenes automático para estudiar procesos de biología celular.
Después de la implantación exitosa de cada técnica "ómica", se crean oportunidades y cuellos de botella en la interfaz entre un conjunto de técnicas y el siguiente. Un cuello de botella clave es el de la bioquímica.
Las máquinas moleculares, formadas a partir de complejos de proteínas, son los componentes básicos que subyacen en la mayoría de los procesos celulares. Se han usado técnicas tales como la técnica de dos híbridos de levadura para identificar interacciones binarias entre pares de proteínas en una escala de todo el genoma, mientras que los análisis complementarios que usan purificación de complejos y espectrometría de masas están comenzando a identificar las combinaciones de componentes que comprenden cada máquina molecular. Sin embargo, el estudio detallado de interacciones bioquímicas requiere la identificación de constantes de velocidad y unión de afinidad, junto con técnicas para estudiar cómo se regulan fisiológicamente estas propiedades.
Las técnicas bioquímicas de "tubo de ensayo" convencionales, incluyendo calorimetría y anisotropía de fluorescencia, requieren la producción y purificación de cantidades de microgramos a miligramos de proteínas solubles que consume tiempo y es por lo tanto poco probable que se aumenten de escala para aplicaciones de alto rendimiento. Una técnica potencialmente más prometedora, la resonancia de plasmón superficial (Biacore®) requiere algo menos de proteína y puede tolerar la presencia de impurezas (en algunos formatos), pero requiere el flujo cuidadosamente temporalizado de proteína de ensayo, seguido de soluciones de lavado sobre un compañero de unión inmovilizado y es por lo tanto poco probable que se adapte fácilmente para análisis de alto rendimiento. Se describen a continuación requisitos proteicos típicos para resonancia de plasmón superficial y calorimetría:
Calorimetría de valoración isotérmica:
Medidas: Kd, estequiometría (n), AG, AN
Requiere: 1 mi de solución 1 pM; 2 nmoles; 1 mg de proteína 5 KDa.
Resonancia de plasmón superficial (Biacore®)
Medidas: Kon, Kff, Kd
Requiere:_______2 mi de solución 1 nM; 2 pmoles; 1 pg de proteína 5 KDa*.
* Cálculo basado en una serie típica de experimentos requeridos para establecer una afinidad de unión en el intervalo de ~1 nM.
Los altos niveles de proteínas que se requieren en estos ensayos resultan del requisito de "saturar" las concentraciones de ligando de unión 4-1 veces más que la constante de disociación. Esto impone los requisitos típicos mostrados en la Tabla 1.
Tabla 1
| Kd | 4x Kd | pg/ml para proteína de 5 KDa |
| 1 nM | 4 nM | ,2 |
| nM | nM | |
| 1 nM | 4 nM | |
| 1 pM | 4 pM | 2 |
Estos niveles de proteína requieren sistemas de síntesis y purificación de proteínas convencionales relativamente caros y que consumen tiempo.
Se conocen métodos de ensayo en los que una biomolécula está unida a una superficie y una segunda biomolécula está en solución, no unida con la superficie o con la otra biomolécula, a partir del documento WO 24/38415 y Goldstein ef al. 1999 (Journal of Molecular Recognition, 12: 293-299).
Pueden usarse experimentos de "sedimentación" de traducción in vitro para identificar interacciones de unión entre cantidades pequeñas de proteína radiomarcada producida en un extracto de traducción (1-15 ng producidos). Sin embargo, como la técnica de 2 híbridos de levadura, padecen la desventaja de que no son cuantitativos, exploran con respecto a intervalos de afinidades de picomolar a bajo nanomolar y no consiguen explorar interacciones de afinidad menores.
Existe un requisito de nuevas técnicas bioquímicas de alto rendimiento que sean cuantitativas, sensibles, pueden usar volúmenes pequeños de analito, pueden requerir un número bajo de moles de analito, pueden conseguir concentraciones altas (hasta micromolares intermedias normalmente de aproximadamente 1-5 pM a aproximadamente 1-2 pM) y pueden ser adaptables para análisis masivamente paralelos.
Sumario de la invención
De acuerdo con un aspecto de la invención se proporciona un método para medir la afinidad de la primera y segunda biomoléculas en el que una primera biomolécula está unida con una primera parte de unión que tiene una longitud de primera parte de unión y una segunda biomolécula está unida con una segunda parte de unión que tiene una longitud de segunda parte de unión, determinando la unión de las primera y segunda biomoléculas adyacentes entre sí, variando al menos una de las longitudes de primera y segunda partes de unión y determinando la unión de las primera y segunda biomoléculas en el que las biomoléculas están unidas con un soporte y/o unidas entre sí.
El método de la invención es ventajoso porque permite un control muy preciso de la concentración de cantidades de biomoléculas y también porque necesita solamente cantidades muy pequeñas de las biomoléculas, permite que se lleven a cabo estudios de unión en equilibrio en volúmenes de pico a attolitros. Los bajos volúmenes permiten realizar una serie de ensayos bioquímicos dependientes de concentración con cantidades de aporte muy bajas de cada biomolécula. Como ejemplo, en una determinación teórica comparando un método de acuerdo con la invención con técnicas convencionales, la calorimetría convencional requeriría 2 nanomoles de proteína o 1 mg de proteína de una proteína de 5 KDa; la Resonancia de Plasmón Superficial requeriría 2 picomoles de proteína correspondientes a 1 microgramos para una proteína de 5 KDa. Por el contrario, un método comparable de acuerdo con la invención requeriría solamente 1 attomoles y 5 picogramos para una proteína de 5 KDa. Normalmente, se usan nano a zeptolitros, preferentemente pico a attolitros, volúmenes de primera y/o segunda biomoléculas.
En este contexto, el término "biomolécula" incluye tanto moléculas de origen natural como molécula sintéticas.
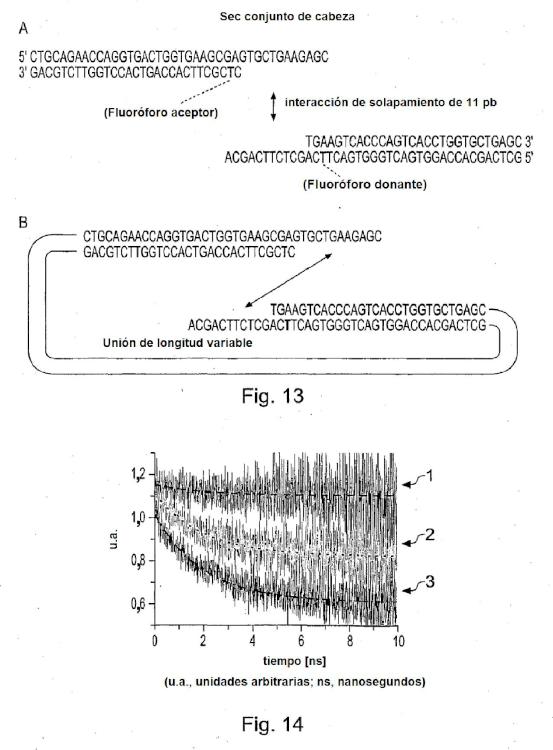
Las primera y segunda biomoléculas pueden unirse con un soporte sólido tal como un portaobjetos de vidrio o microperla. En dicha configuración, las primera y segunda biomoléculas están unidas por separado con el mismo soporte sólido. Como alternativa, las primera y segunda biomoléculas pueden estar unidas entre sí en una disposición "en forma de Y". En otra realización, las primera y segunda biomoléculas pueden estar unidas por una unión pero no estar unidas con un soporte sólido, en una disposición de "molécula lineal". Por ejemplo, las primera y segunda biomoléculas pueden estar unidas entre sí y presentes en una solución.
Cuando las biomoléculas están unidas con un soporte sólido, las biomoléculas pueden disponerse de forma aleatoria sobre el soporte sólido. Como alternativa, las moléculas pueden estar unidas con partes discretas de, o áreas definidas en, el soporte sólido de modo que las primera y segunda biomoléculas pueden interaccionar solamente si se "estiran" para abarcar el hueco entre las partes discretas. Controlando la distancia entre las partes discretas y/o la longitud de la unión, la proporción de biomoléculas unidas y libres puede alterarse permitiendo la determinación de la afinidad descrita anteriormente. Las partes discretas de la superficie pueden acoplarse con las primera y segunda biomoléculas usando una serie de técnicas incluyendo litografía de haz de iones, electrones o fotones para desproteger secuencialmente partes para el acoplamiento. Como alternativa, puede usarse el grabado/modificación directa usando puntas microscópicas de fuerza atómica para introducir una modificación de superficie regional selectiva. Una ventaja de este tipo de enfoque es que las distancias intermoleculares pueden controlarse directamente... [Seguir leyendo]
Reivindicaciones:
1. Un método para medir la afinidad de primera y segunda biomoléculas en el que una primera biomolécula está
unida por una primera parte de unión que tiene una longitud de primera parte de unión y una segunda biomolécula está unida por una segunda parte de unión que tiene una longitud de segunda parte de unión, determinar la unión de primera y segunda biomoléculas adyacentes entre sí, variar al menos una de las longitudes de las primera y segunda partes de unión y determinar la unión de las primera y segunda biomoléculas, en el que las biomoléculas están unidas a un soporte y/o unidas entre sí.
2. Un método de acuerdo con la reivindicación 1 en el que la unión es en forma de Y.
3. Un método de acuerdo con la reivindicación 2 en el que la longitud de cada rama se varía de forma independiente.
4. Un método de acuerdo con la reivindicación 1 en el que las primera y segunda biomoléculas están unidas entre sí
en una disposición molecular lineal.
5. Un método de acuerdo con la reivindicación 1 en el que tanto la primera como la segunda biomoléculas están unidas a un soporte.
6. Un método de acuerdo con la reivindicación 5 en el que las primera y segunda biomoléculas están unidas entre sí y al soporte.
7. Un método de acuerdo con las reivindicaciones 5 o 6, en el que el soporte lo proporciona un soporte sólido.
8. Un método de acuerdo con cualquier reivindicación anterior en el que las primera y segunda partes de unión se proporcionan por medio de una única unión.
9. Un método de acuerdo con cualquier reivindicación anterior en el que las primera y segunda biomoléculas están estrechamente adyacentes entre sí, de modo que los volúmenes de barrido sustancialmente hemisféricos definidos por el movimiento de cada biomolécula se solapan, de modo que las primera y segunda biomoléculas pueden unirse entre sí.
1. Un método de acuerdo con cualquier reivindicación anterior en el que variando las longitudes de primera y segunda partes de unión pueden variarse las concentraciones efectivas de las primera y/o segunda biomoléculas.
11. Un método de acuerdo con la reivindicación 1 en el que se determina una serie de reacciones de unión a las que afecta una tercera biomolécula determinando la unión de las primera y segunda biomoléculas.
12. Un método de acuerdo con cualquier reivindicación anterior en el que al menos una parte de unión comprende nucleótidos.
13. Un método de acuerdo con la reivindicación 12 en el que la o cada parte de unión comprende ADN bicatenario.
14. Un método de acuerdo con una cualquiera de las reivindicaciones 1 a 11 en el que la unión, o cada una de ellas, comprende un nanotubo de carbono, fibrilla amiloide o un polímero.
15. Un método de acuerdo con la reivindicación 14 en el que el polímero es un complejo de cruce de ADN tal como un híbrido DX.
16. Un método de acuerdo con una cualquiera de las reivindicaciones 5 a 15 en el que al menos una parte de unión está unida al soporte sólido por medio de un anclaje oligonucleotídico.
17. Un método de acuerdo con la reivindicación 16 en el que se sintetizan en serie oligonucleótidos de anclaje para las primera y segunda uniones.
18. Un método de acuerdo con cualquier reivindicación anterior en el que la proporción de las primera y segunda biomoléculas que están molecularmente cerca entre sí indica la proporción de primera y segunda biomoléculas que interaccionan.
19. Un método de acuerdo con cualquier reivindicación anterior en el que la proporción de primera y segunda biomoléculas de unión se determina por la intensidad de transferencia de energía por resonancia de Forster (FRET) entre los primer y segundo fluoróforos respectivamente unidos a, o integrados con, las primera y segunda biomoléculas.
2. Un método de acuerdo con una cualquiera de las reivindicaciones 7 a 16 en el que el soporte sólido es un portaobjetos de vidrio y están impresos anclajes o uniones oligonucleotídicos en matrices en una superficie del
portaobjetos de vidrio.
21. Un método de acuerdo con una cualquiera de las reivindicaciones 9 a 2 en el que el soporte lo proporciona una microperla acoplada a una combinación específica de primera y segunda biomoléculas unidas.
22. Un método de acuerdo con una cualquiera de las reivindicaciones 12 a 2 en el que al menos una de las primera y segunda biomoléculas es una biomolécula proteica o polipeptídica conectada con una primera unión de ácido nucleico por medio de una reacción de traducción in vitro para unir covalentemente un péptido naciente por su extremo C terminal cerca de, o en, el extremo 3 de un conjugado de ARNm-ADN.
23. Un método de acuerdo con cualquier reivindicación anterior en el que la Kd de una interacción entre las primera y segunda biomoléculas se determina determinando la proporción de primera y segunda biomoléculas unidas para una serie de concentraciones de las primera y segunda biomoléculas y determinando después la concentración (Kd) de la primera o segunda biomolécula requerida para unión semimáxima de las primera y segunda biomoléculas.
24. Un método de acuerdo con la reivindicación 1 en el que el valor de Kff para una interacción entre las primera y segunda biomoléculas se determina proporcionando concentraciones de saturación iniciales de las primera y segunda biomoléculas, escindiendo una segunda parte de unión o anclaje y controlando cualquier cambio en los niveles de primera y segunda biomoléculas unidas.
25. Un método de acuerdo con una cualquiera de las reivindicaciones 1 a 17 que comprende proporcionar una concentración de primera y segunda biomoléculas en torno a la Kd de una interacción entre las primera y segunda biomoléculas en el que se determina el efecto de un modulador de la interacción entre las primera y segunda biomoléculas.
Patentes similares o relacionadas:
Método de determinación de la presencia y/o cantidad de moléculas diana, del 22 de Julio de 2020, de Canopy Biosciences, LLC: Método para el análisis de células individuales en una muestra de sangre mediante la determinación de la presencia y/o cantidad de una o más moléculas […]
Kit de reactivos utilizado para detectar gastrina-17 y método de preparación y aplicación para el kit de reactivos, del 15 de Julio de 2020, de Shenzhen New Industries Biomedical Engineering Co., Ltd: Un kit para detectar gastrina-17, caracterizado porque comprende un componente A y un componente B, en donde el componente A es un primer […]
Un sustrato para inmovilizar sustancias funcionales y un método para preparar las mismas, del 15 de Julio de 2020, de Temasek Polytechnic: Un sustrato sólido que tiene compuestos dispuestos sobre el mismo, en donde se inmoviliza una molécula funcional sobre los compuestos, teniendo cada compuesto una cadena que […]
Análisis biológico autónomo de alta densidad, del 1 de Julio de 2020, de BioFire Diagnostics, LLC: Recipiente para realizar reacciones de amplificación en un sistema cerrado que comprende una porción flexible que tiene una serie de blísteres […]
Chip de análisis y aparato de análisis de muestras, del 1 de Julio de 2020, de Takano Co., Ltd: Un chip de análisis que comprende: un sustrato conformado sustancialmente en forma de disco; un puerto de inyección formado […]
Dispositivo para la detección de analitos, del 1 de Julio de 2020, de TECHLAB, INC.: Un dispositivo para detectar por lo menos una sustancia de interés en una muestra líquida, comprendiendo el dispositivo: (a) una unidad que […]
Estructuras para controlar la interacción de luz con dispositivos microfluídicos, del 3 de Junio de 2020, de Opko Diagnostics, LLC: Un sistema de ensayo múltiplex que comprende: un artículo que soporta ensayos en fase sólida, comprendiendo dicho artículo un sustrato plano rígido y comprendiendo […]
Uso de un estándar para la detección de agregados de proteínas de una enfermedad por plegamiento incorrecto de proteínas, del 27 de Mayo de 2020, de FORSCHUNGSZENTRUM JULICH GMBH: Uso de un estándar, que comprende una nanopartícula inorgánica con un tamaño de 2 a 200 nm, en cuya superficie ya sea mediante i) ácido carboxílico-espaciador-maleimida […]