Análisis de espectro de masas.
Un procedimiento para analizar un analito diana, donde el procedimiento comprende:
(a) proporcionar una pluralidad de muestras que puede comprender el analito diana, donde cada muestra estamarcada de forma diferencial con un marcador de masa o una combinacion de marcadores de masa, donde losmarcadores de masa proceden de un conjunto de marcadores de masa, donde cada marcador de masa es unmarcador de masa isobarico que comprende un grupo marcador de masa con espectrometria de masas distinta,de forma que las muestras se pueden distinguir mediante espectrometria de masas.
(b) mezclar la pluralidad de las muestras marcadas para producir una mezcla de analisis e introducir la mezcla deanalisis en un espectrometro de masas;
(c) seleccionar iones que tienen una primera proporcion masa-carga equivalente a un ion del analito dianamarcado con un numero especifico de los marcadores de masa;
(d) fragmentar iones de la primera proporcion masa-carga en una pluralidad de iones de fragmentos, donde laproporcion de la pluralidad de iones de fragmentos comprende al menos un marcador de masa intacto.
(e) seleccionar iones que tienen una segunda proporcion masa-carga equivalente a un ion de un fragmento delanalito diana que comprende al menos un marcador de masa intacto;
(f) fragmentar iones de la segunda proporcion masa-carga en una pluralidad de iones de fragmentos adicionales,donde la proporcion de los iones de fragmentos adicionales son iones de los grupos marcadores de masa;
(g) producir un espectro de masas de los iones de fragmentos adicionales producidos en la etapa (f); y
(h) determinar a partir del espectro de masas la cantidad del analito diana en cada muestra.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2009/056010.
Solicitante: ELECTROPHORETICS LIMITED.
Nacionalidad solicitante: Reino Unido.
Dirección: COVEHAM HOUSE DOWNSIDE BRIDGE ROAD COBHAM SURREY KT11 3EP REINO UNIDO.
Inventor/es: SCHULZ-KNAPPE, PETER, BYERS,HELEN, BAUMANN,CHRISTIAN, WARD,MALCOLM.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- G01N33/68 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › en los que intervienen proteínas, péptidos o aminoácidos.
PDF original: ES-2444722_T3.pdf
Fragmento de la descripción:
Análisis de espectro de masas La presente invención se refiere a un procedimiento de análisis de un analito diana por espectrometría de masas, en particular biomoléculas, tales como ácidos nucleicos y proteínas. La invención se refiere específicamente a un procedimiento de espectrometría de masas en tándem multiplexada utilizando marcadores de masa isobáricos. La presente divulgación se refiere también a un dispositivo de espectrometría de masas para analizar uno o más analitos objetivo.
En la técnica se conocen diversos procedimientos para marcar moléculas de interés, incluyendo átomos radiactivos, tintes fluorescentes, reactivos luminiscentes, reactivos de captura de electrones y tintes absorbedores de luz. Cada uno de estos sistemas de marcaje tiene características que lo hacen adecuado para ciertas aplicaciones y no para otras. Más recientemente, se han producido avances en el campo de la espectrometría de masas como procedimiento para detectar marcadores que están unidos de forma escindible a su molécula de interés asociada.
Para muchas aplicaciones, tales como el análisis del ácido nucleico, la estructura del analito puede determinarse a partir del marcaje indirecto. Esto es particularmente ventajoso en relación con el uso de la espectrometría de masas, porque las biomoléculas complejas, tales como el ADN, tienen espectros de masas complejos y se detectan con una sensibilidad relativamente mala. Detección indirecta significa que una molécula marcadora asociada puede utilizarse para identificar el analito original, en la que el marcador está diseñado para la detección sensible y un simple espectro de masas. Espectros de masas simples significa que se pueden utilizar múltiples marcadores para analizar múltiples analitos de forma simultánea.
El documento PCT/GB98/00127 describe conjuntos de sondas de ácido nucleico unidas covalentemente a marcadores escindibles que son detectables por espectrometría de masas, que identifican la secuencia de la sonda de ácido nucleico unida covalentemente. Las sondas marcadas de la presente solicitud tienen la estructura Nu-L-M, en la que Nu es un ácido nucleico unido covalentemente a L, un ligador escindible, unido covalentemente a M, un marcador de masa. Los ligadores escindibles preferidos en la presente solicitud se escinden dentro de la fuente de iones del espectrómetro de masas. Los marcadores de masa preferidos son éteres de poliarilo sustituidos. La presente solicitud divulga una variedad de procedimientos de ionización y análisis por analizadores de masa de cuadrupolo, analizadores de tiempo de vuelo (TOF) e instrumentos del sector magnético como procedimientos específicos para analizar marcadores de masa por espectrometría de masas.
El documento PCT/GB94/01675 divulga ligandos, y específicamente ácidos nucleicos, unidos de forma escindible a moléculas marcadoras de masa. Los ligadores escindibles preferidos son fotoescindibles. La presente solicitud describe espectrometría de masas de Ionización por Desorción Láser Asistida por Matriz, (MALDI) Tiempo de vuelo (TOF) , como un procedimiento específico para analizar los marcadores de masa por espectrometría de masas.
El documento PCT/US97/22639 divulga moléculas marcadoras de masa no volátiles liberables. En realizaciones preferidas estos marcadores comprenden polímeros, típicamente biopolímeros, que pueden fijarse de forma escindible a un grupo reactivo o ligando, es decir, una sonda. Ligadores escindibles preferidos parecen ser química o enzimáticamente escindibles. La presente solicitud divulga espectrometría de masas MALDI TOF como un procedimiento específico para analizar los marcadores de masa por espectrometría de masas.
Los documentos PCT/US97/01070, PCT/US97/01046 y PCT/US97/01304 divulgan ligandos y específicamente ácidos nucleicos unidos de forma escindible a moléculas marcadoras de masa. Ligadores escindibles preferidos parecen ser química o enzimáticamente escindibles. Las presentes solicitudes divulgan una variedad de procedimientos de ionización y análisis por analizadores de masa de cuadrupolo, analizadores de TOF e instrumentos del sector magnético como procedimientos específicos para analizar marcadores de masa por espectrometría de masas.
Ninguna de estas solicitudes en la técnica anterior menciona el uso de análisis de masas en tándem o en serie de biomoléculas marcadas.
Gygi et al. (Nature Bioteclmology 17; 994-999, quot;Quantitative analysis of complex protein mixtures using isotopecoded affinity tagsquot; 1999) divulgan el uso de “marcadores de afinidad codificados en isótopos” para la captura de péptidos a partir de proteínas, para permitir el análisis de la expresión proteica. En este artículo los autores describen el uso de un ligador de biotina, que es reactivo a los tioles, para la captura de péptidos con cisteína en su interior. Una muestra de proteína de una fuente se hace reaccionar con el ligador de biotina y se escinde con una endopeptidasa. Los péptidos que contienen cisteína biotinilada pueden aislarse después sobre perlas con avidina para análisis posterior por espectrometría de masas. Dos muestras pueden compararse cuantitativamente por marcaje de una muestra con el ligador de biotina y marcaje de la segunda muestra con una forma adulterada del ligador de biotina. Cada péptido en las muestras se representa después como un par de picos en el espectro de 65 masas, La integración de los picos en el espectro de masas correspondiente a cada marcador indica los niveles relativos de expresión del péptido único a los marcadores.
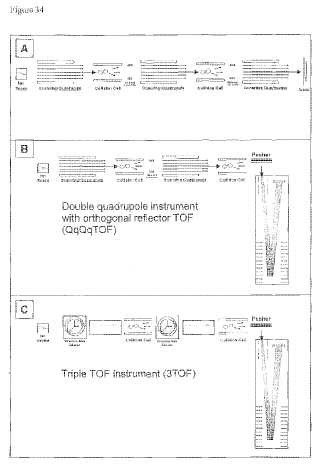
La monitorización selectiva de reacción (SRM) y la monitorización de reacción múltiple (MRM) proporcionan procedimientos altamente selectivos de espectrometría de masas tándem, que tienen la capacidad de filtrar y eliminar de forma efectiva todas las moléculas y contaminantes excepto el analito deseado. Esto es particularmente beneficioso si se analizan muestras complejas que tienen a tener presentes varias especies isobáricas dentro de 5 una ventana analítica definida. Normalmente, una combinación de selección del precursor (ion parental) en la primera etapa del espectrómetro de masas (en el presente documento denominado Q1: cuadrupolo 1, pero también equivalente para las fases respectivas en espectrómetros de masas no cuadrupolos, como trampas de iones, etc.) , fragmentación del ion parental en muchos fragmentos de los cuales se selecciona uno o varios fragmentos específicos en las etapas siguientes de la medición de espectrómetro de masas (normalmente en cuadrupolo 3, Q3) y se detecta con el detector de iones. Esta selección en dos etapas asegura que se mide el analito deseado y a cualquier otra especie de ion se le reduce la intensidad. La relación señal-ruido es muy superior a los experimentos convencionales EM/EM, que seleccionan una ventana de masa en Q1, y después miden todos los fragmentos generados en el detector de iones. En principio, este sistema basado en EM puede proporcionar una absoluta especificidad estructural para el analito, y, en combinación con los patrones internos marcados con isótopos (SIS)
estables adecuados, puede proporcionar una cuantificación absoluta de la concentración de analito.
En los experimentos convencionales de tipo SRM/MRM, se utiliza la referencia a un isótopo estable marcado para generar un par analito/referencia que se utilizará para la cuantificación del analito frente a la referencia. Para el análisis de proteínas, tal péptido de referencia difiere del analito que hay que medir solo por incorporación de isótopos, para hacerlo claramente diferente en masa para la selección de Q1, pero sin embargo idéntico en la composición química y en el comportamiento físico-químico. En un experimento típico, se selecciona el par analito/referencia, esto es en Q1 conectando canales de selección de masas entre estas dos masas. La posterior fragmentación de estos dos iones conduce a fragmentos con masas distintas (específicas) . Después se escogen una o más masas de fragmentos adecuadas, en las que el filtro Q3 permanece en la posición de los iones del
fragmento seleccionados, asegurando de este modo la transición de este ión al analizador de masas, y filtrando y eliminando otras especies de iones.
Los recientes trabajos de diseño mejoraron los marcadores de masa para la identificación de analitos por medio del uso de la espectrometría de masas se han centrado en los marcadores de masa que se identifican más fácilmente en el espectro de masas sin otros contaminantes.
El documento WO 01/68664 divulga un conjunto de dos o más marcadores de masa, comprendiendo cada marcador en el conjunto un resto marcador de masa unido por medio de un ligador escindible a... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento para analizar un analito diana, donde el procedimiento comprende:
(a) proporcionar una pluralidad de muestras que puede comprender el analito diana, donde cada muestra está marcada de forma diferencial con un marcador de masa o una combinación de marcadores de masa, donde los marcadores de masa proceden de un conjunto de marcadores de masa, donde cada marcador de masa es un marcador de masa isobárico que comprende un grupo marcador de masa con espectrometría de masas distinta, de forma que las muestras se pueden distinguir mediante espectrometría de masas.
(b) mezclar la pluralidad de las muestras marcadas para producir una mezcla de análisis e introducir la mezcla de análisis en un espectrómetro de masas;
(c) seleccionar iones que tienen una primera proporción masa-carga equivalente a un ion del analito diana marcado con un número específico de los marcadores de masa;
(d) fragmentar iones de la primera proporción masa-carga en una pluralidad de iones de fragmentos, donde la 15 proporción de la pluralidad de iones de fragmentos comprende al menos un marcador de masa intacto.
(e) seleccionar iones que tienen una segunda proporción masa-carga equivalente a un ion de un fragmento del analito diana que comprende al menos un marcador de masa intacto;
(f) fragmentar iones de la segunda proporción masa-carga en una pluralidad de iones de fragmentos adicionales, donde la proporción de los iones de fragmentos adicionales son iones de los grupos marcadores de masa;
(g) producir un espectro de masas de los iones de fragmentos adicionales producidos en la etapa (f) ; y
(h) determinar a partir del espectro de masas la cantidad del analito diana en cada muestra.
2. El procedimiento de acuerdo con la reivindicación 1, donde una muestra es una muestra de ensayo y una muestra es una muestra de calibración, donde la muestra de calibración comprende uno o más alícuotas diferentes del
analito diana, teniendo cada alícuota una cantidad conocida del analito, donde la muestra de ensayo y cada alícuota de la muestra de calibración está marcados de forma diferencial, y/o donde la pluralidad de las muestras puede comprender una pluralidad de diferentes analitos diana y el procedimiento comprende una etapa de repetir etapas (c) a (h) para cada analito diana, preferentemente donde una muestra es una muestra de ensayo y una muestra de calibración se proporciona para cada analito diferente, donde cada muestra de calibración comprende uno o más alícuotas diferentes de un analito diana, donde la muestra de ensayo y cada alícuota de cada muestra de calibración se marcan de forma diferencial. y/o donde la o cada muestra de calibración comprende dos o más alícuotas diferentes del analito diana. y/o donde una pluralidad de las muestras de ensayo se analizan para determinar un analito, preferentemente donde cada una de la pluralidad de muestras de ensayo se analiza para el mismo analito y/o preferentemente donde cada muestra de ensayo se marca de forma diferencial con uno o más de los marcadores de masa isobáricos, y/o donde el procedimiento comprende una etapa adicional antes que la etapa (a) de cada muestra de ensayo marcada de forma diferencial y cada alícuota de la muestra de calibración con uno o más marcadores de masa isobáricos, preferentemente donde el procedimiento comprende una etapa adicional de combinar los alícuotas marcados de forma diferencial para producir una muestra de calibración antes de la etapa (a) , y/o donde la pluralidad de las muestras son muestras de ensayo, y/o donde la cantidad determinada en la etapa (h) es la cantidad relativa del analito diana en cada muestra o donde la cantidad determinada en la etapa (h) es la cantidad absoluta del analito diana en cada muestra, y/o donde el procedimiento comprende una etapa adicional tras la etapa (d) de producir un espectro de masas de la pluralidad de iones de fragmentos de la etapa (d) , preferentemente donde la identidad del analito diana se determina 45 identificando uno o más iones de fragmentos característicos del analito diana en el espectro de masas. y/o donde el procedimiento comprende una etapa adicional tras la etapa (f) de seleccionar iones de un intervalo de proporciones masa-carga equivalente al intervalo de proporciones masa-carga de los grupos indicadores de masa, y/o donde en la etapa (c) , la primera proporción masa-carga es equivalente a la proporción masa-carga del ion parental no fragmentado del analito diana marcado con un número específico de marcadores de masa o donde en la etapa ©, la primera proporción masa-carga es equivalente a la proporción masa-carga de un ion de fragmento del analito diana marcado con un número específico de marcadores de masa. y/o donde en la etapa (e) , la segunda proporción masa-carga es equivalente a un ion de un fragmento del analito diana que comprende al menos un marcador de masa intacto cuyo ion de fragmento es único del analito diana, y/o donde en la etapa (e) , la segunda proporción masa-carga es la proporción masa-carga de un ion de serie y que 55 comprende un marcador de masa intacto o donde la etapa (e) , la segunda proporción masa-carga es la proporción masa-carga de un ion de serie b que comprende un marcador de masa intacto, preferentemente donde el ion de serie y y el ion de serie b tiene una proporción masa-carga más alta en comparación con la primera proporción masa-carga seleccionada en la etapa (c) y/o donde el analito diana se selecciona de una proteína, un polipéptido, un péptido, un aminoácido o un ácido nucleico, o fragmentos de los mismos, y/o donde las etapas (c) a (g) se llevan a cabo en cuadrupolos separados en un espectrómetro de masas o donde las etapas (c) a (g) se llevan a cabo secuencialmente en la misma zona de un espectrómetro de masas, y/o donde una de las muestras comprende un alícuota desencadenante que comprende un analito desencadenante y el procedimiento comprende una etapa adicional después de la etapa (b) y antes de la etapa (c) de detectar iones 65 que tienen una proporción carga-masa equivalente a la proporción carga-masa del analito desencadenante, donde cuando se detectan iones que tienen una proporción carga-masa equivalente a la proporción carga-masa del analito desencadenante, se inicia la etapa (c) , preferentemente donde el analito desencadenante en el alícuota desencadenante está marcado con un marcador de masa isobárico, o donde el analito desencadenante en el alícuota desencadenante está marcado con un marcador de masa que es químicamente idéntico pero isotópicamente distinto y diferente en masa de los marcadores de masa isobáricos de los otros analitos en las muestras.
3. Un procedimiento de acuerdo con la reivindicación 1 o la reivindicación 2, donde el marcador de masa comprende la estructura siguiente:
X-L-M
donde X es un resto marcador de masa que comprende el grupo siguiente:
donde la unidad cíclica es aromática o alifática y comprende de 0-3 dobles enlaces de forma independiente entre dos cualquiera átomos adyacentes; cada Z es, de forma independiente N, N (R1) , C (R1) , CO, CO (R1) (es decir, -OC (R1) -o -C (R1) -O-) , C (R1) 2, O o S; X es N, C o C (R1) ; cada R1 es de forma independiente H, un grupo alquilo C1-C6 lineal o ramificado sustituido o insustituido, un grupo cíclico alifático sustituido o insustituido, un grupo aromático sustituido o insustituido, un grupo heterocíclico sustituido o insustituido; e y es un número entero de 0-10, L es un ligador escindible y M es un resto de normalización de masa, preferentemente donde el ligador escindible que une el resto marcador de masa al resto de normalización de masa es un ligador escindible mediante colisión, preferentemente donde el ligador es escindible mediante DIC, DTE, DCE
o DIS usando espectrometría de masas. 25
4. Un procedimiento de acuerdo con la reivindicación 2, donde la etapa de marcaje comprende una etapa de hacer reaccionar el analito con un marcador de masa reactivo, donde el marcador de masa reactivo comprende un marcador de masa y una funcionalidad reactiva, preferentemente donde la funcionalidad reactiva es capaz de reaccionar con cualquier grupo amino en el polipéptido y comprende un nucleófilo o un electrófilo. y/o preferentemente, donde el marcador de masa es un marcador de masa reactivo para marcar y detectar un polipéptido mediante espectrometría de masas, donde el marcador de masa comprende una funcionalidad reactiva para unir el marcador de masa al polipéptido, donde la funcionalidad reactiva comprende le grupo siguiente:
donde cada R2 es, de forma independiente H, un grupo alquilo C1-C6 lineal o ramificado sustituido o insustituido, un grupo cíclico alifático sustituido o insustituido, un grupo aromático sustituido o insustituido o un grupo heterocíclico sustituido o insustituido.
5. Un procedimiento de acuerdo con cualquiera de las reivindicaciones 2-4, donde el marcador de masa es un marcador de masa de un conjunto de dos o más marcadores de masa, donde cada resto de normalización de masa garantiza que un marcador de masa tiene una masa agregada deseada y donde el conjunto comprende marcadores de masa que tienen un resto marcador de masa, teniendo cada resto marcador de masa una masa diferente de la de 45 los demás grupos marcadores de masa en el conjunto y teniendo cada marcador en el conjunto una masa agregada común; y donde los marcadores de masa en el conjunto se pueden distinguir entre sí mediante espectroscopia de masas,
preferentemente, donde cada marcador de masa en el conjunto tiene un resto ajustador de masa, seleccionado de:
(a) un sustituyente isotópico situado dentro del resto marcador de masa y/o dentro del resto de normalización de masa, y
(b) átomos o grupos sustituyentes unidos al resto marcador de masa y/o unidos al resto de normalización de masa.
más preferentemente, donde el resto ajustador de masa se selecciona de un sustituyente átomo de halógeno, un grupo metilo sustituyente y sustituyentes isotópicos 2H, 15N, 13C o 18O, más preferentemente donde el resto ajustador 10 de masa es 15N o 13C y el conjunto comprende dos marcadores de masa que tienen las estructuras siguientes:
más preferentemente, donde el resto ajustador de masa es 15N and 13C y el conjunto comprende cinco marcadores de masa que tienen las estructuras siguientes:
6. Un procedimiento de acuerdo con la reivindicación 5, donde el resto ajustador de masa es 15N y 13C y el conjunto comprende seis marcadores de masa que tienen las estructuras siguientes:
Patentes similares o relacionadas:
Neuregulina para tratar la insuficiencia cardíaca, del 29 de Julio de 2020, de Zensun (Shanghai) Science & Technology, Co., Ltd: Neuregulina para usar en un método para tratar la insuficiencia cardíaca crónica en un paciente, donde el paciente tiene un nivel plasmático de NT-proBNP […]
Inmunomoduladores, del 29 de Julio de 2020, de BRISTOL-MYERS SQUIBB COMPANY: Un compuesto de la fórmula (I) **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo, en donde: A se selecciona de **(Ver fórmula)** en donde: […]
Métodos y composiciones para el diagnóstico y pronóstico de lesión renal e insuficiencia renal, del 29 de Julio de 2020, de Astute Medical, Inc: Un método para evaluar el estado renal en un sujeto, que comprende: realizar una pluralidad de ensayos configurados para detectar una […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Método para llevar a cabo el seguimiento de la enfermedad de Gaucher, del 15 de Julio de 2020, de Centogene GmbH: Un método para determinar la evolución de la enfermedad de Gaucher en un sujeto, que comprende la etapa de determinar en varios puntos en el […]
Procedimiento para evaluación de la función hepática y el flujo sanguíneo portal, del 15 de Julio de 2020, de The Regents of the University of Colorado, a body corporate: Procedimiento in vitro para la estimación del flujo sanguíneo portal en un individuo a partir de una única muestra de sangre o suero, comprendiendo el procedimiento: […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos de monitorización terapéutica de profármacos de ácido fenilacético, del 24 de Junio de 2020, de Immedica Pharma AB: Glicerilo tri-[4-fenilbutirato] (HPN-100) para su uso en un método para tratar un trastorno del ciclo de la urea en un sujeto que tiene discapacidad […]