HUELLA GENÓMICA COMO PREDICTOR DE RESPUESTA A TRATAMIENTO.
Huella genómica como predictor de respuesta a tratamiento.
La invención se relaciona con un método de predicción de la respuesta al tratamiento con un taxano más quimioterapia con un antimetabolito,
un agente intercalante y un agente alquilante del ADN, en pacientes con cáncer de mama. En concreto, los autores han desarrollado un modelo mediante regresión logística en que el test genómico de 40 genes, combinado con parámetros clínicos, predice con precisión la respuesta patológica completa (RCp) al tratamiento neoadyuvante de paclitaxel más quimioterapia con fluorouracilo, adriamicina y ciclofosfamida (T/FAC) en pacientes de cáncer de mama con una expresión normal del gen HER-2. Dicho modelo tiene mejor rendimiento que las variables clínicas utilizadas rutinariamente.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P200931311.
Solicitante: CENTRO DE INVESTIGACIONES ENERGETICAS, MEDIOAMBIENTALES Y TECNOLOGICAS (CIEMAT).
Nacionalidad solicitante: España.
Inventor/es: GARCIA ESCUDERO,RAMON, PARAMIO GONZALEZ,JESUS MARIA.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
- G01N33/50 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › Análisis químico de material biológico, p. ej. de sangre o de orina; Ensayos mediante métodos en los que interviene la formación de uniones bioespecíficas con grupos coordinadores; Ensayos inmunológicos (procedimientos de medida o ensayos diferentes de los procedimientos inmunológicos en los que intervienen enzimas o microorganismos, composiciones o papeles reactivos a este efecto, procedimientos para preparar estas composiciones, procedimientos de control sensibles a las condiciones del medio en los procedimientos microbiológicos o enzimáticos C12Q).
- G01N33/53 G01N 33/00 […] › Ensayos inmunológicos; Ensayos en los que interviene la formación de uniones bioespecíficas; Materiales a este efecto.
- G06F19/00
Fragmento de la descripción:
Huella genómica como predictor de respuesta a tratamiento.
Campo de la invención
La presente invención se relaciona con métodos in vitro para la predicción de la respuesta de un sujeto con cáncer de mama al tratamiento de un taxano más quimioterapia con un antimetabolito, un agente intercalante y un agente alquilante.
Antecedentes de la invención
A nivel mundial, el cáncer de mama es el segundo tipo más común de cáncer (10,4%; tras el cáncer de pulmón) y la quinta causa más común de muerte por cáncer (tras el cáncer de pulmón, cáncer de estómago, cáncer de hígado, y cáncer de colon). Entre las mujeres en el mundo, el cáncer de mama es la causa más común de muerte por cáncer. En 2005, el cáncer de mama produjo 502.000 muertes en el mundo (el 7% de las muertes por cáncer; casi el 1% de todas las muertes). El número de casos mundiales ha aumentado significativamente desde la década de 1970, un fenómeno del que se echa la culpa parcialmente a los estilos de vida modernos en el mundo occidental. Las mujeres de Norteamérica tienen la mayor incidencia de cáncer de mama en el mundo.
Debido a que la mama está compuesta de tejidos idénticos en hombres y mujeres, el cáncer de mama también se da en hombres. La incidencia de cáncer de mama en hombres es aproximadamente 100 veces menor que en mujeres, aunque se considera que los hombres con cáncer de mama tienen estadísticamente las mismas tasas de supervivencia que las mujeres.
El cáncer de mama se clasifica en fases según el sistema TNM. El pronóstico está íntimamente unido a los resultados de la clasificación en fases, y la clasificación en fases también se usa para asignar pacientes a tratamientos tanto en ensayos clínicos como en la práctica médica. Los diferentes estadios se pueden clasificar siguiendo el sistema TNM desarrollado por la AJCC (American Joint Committee on Cáncer) en colaboración con la UICC (Unión Internacional Contra Cáncer):
• TX: El tumor primario no se puede evaluar. T0: No hay evidencia de tumor. Tis: Carcinoma in situ, no invasión. T1: El tumor es de 2 cm o menor. T2: El tumor es mayor de 2 cm pero menor de 5 cm. T3: El tumor es mayor de 5 cm. T4: Tumor de cualquier tamaño que crece en la pared del pecho o piel, o cáncer de mama inflamatorio.
• NX: Los ganglios linfáticos cercanos no se pueden evaluar. N0: El cáncer no se ha extendido a los ganglios linfáticos regionales. N1: El cáncer se ha extendido a 1 a 3 ganglios linfáticos de la axila o a uno mamario interno. N2: El cáncer se ha extendido a 4 a 9 ganglios linfáticos de la axila o a múltiples ganglios mamarios internos. N3: Aplica uno de los siguientes:
• MX: La presencia de extensión distante (metástasis) no se puede evaluar. M0: No existe metástasis distante. M1: Se ha producido la extensión a órganos distantes, que no incluyen el ganglio linfático supraclavicular.
Por tanto, la clasificación de los tumores de mama basada en los criterios TNM sería:
El pilar principal del tratamiento de cáncer de mama es la cirugía cuando el tumor está localizado, con posible terapia adyuvante hormonal (con tamoxifeno o un inhibidor de aromatasa), quimioterapia, y/o radioterapia. En la actualidad, las recomendaciones de tratamiento después de la cirugía (terapia adyuvante) siguen un patrón. Este patrón está sujeto a cambio, ya que cada dos años, tiene lugar una conferencia mundial en St. Gallen, Suiza, para discutir los resultados reales de los estudios multicentros mundiales. Asimismo, dicho patrón también se revisa según el criterio consenso del National Institute of Health (NIH). Basándose en estos criterios, más del 85-90% de los pacientes que no presentan metástasis en nódulos linfáticos serían candidatos para recibir terapia sistémica adyuvante.
La quimioterapia neoadyuvante es la administración de quimioterapia antes de cirugía de cáncer de mama, con el objetivo de reducir la extensión de la cirugía o impedir la cirugía por completo. La respuesta a quimioterapia neoadyuvante se dicotomiza usualmente como respuesta patológica completa (RCp) ó enfermedad residual (ER). RCp es un punto final clínico significativo para predecir, ya que estos pacientes poseen supervivencia global y supervivencia libre de enfermedad prolongada cuando se compara con pacientes con respuesta menor. Debido a que la terapia neoadyuvante o adyuvante con trastuzumab es efectiva en pacientes de cáncer de mama con sobreexpresión del gen HER-2 junto con quimioterapia, este régimen combinado se recomienda actualmente para pacientes positivos para HER-2.
Hess et al. (Hess et al, 2006. J. Clin. Oncol. 24: 4236-44) describen el desarrollo de un "predictor" multigénico de Respuesta Patológica completa (RCp) de pacientes con cáncer de mama para tratamientos con paclitaxel/fluorouracilo, adriamicina y ciclofosfamida (T/FAC) y determinaron su precisión predictiva en casos independientes. Dicho estudio incluía 99 pacientes con niveles normales de HER-2. De esta manera, identificaron un predictor con 30 sondas que predecía la RCp a la quimioterapia T/FAC. Otros tests genómicos de cáncer de mama capaces de identificar tumores con mala prognosis basal en respuesta a quimioterapia han sido descritos conocidos en el estado de la técnica incluyen test 21-gene (Paik et al, 2004, N Engl J Med. 351: 2817-2826), intrinsic subtypes (Parker et al, 2009, J. Clin. Oncol. 27:1160-1167) o Genomic Grade Index (Liedtke et al, 2009, J. Clin. Oncol. 27: 3185-3191). Sin embargo, dichos test genómicos no presentan una alta sensibilidad ni especificidad.
Por tanto, existe la necesidad de desarrollar nuevos métodos que permitan predecir la respuesta a un tratamiento con paclitaxel/fluorouracilo, adriamicina y ciclofosfamida en pacientes con cáncer de mama y que presenten mayor sensibilidad que los métodos existentes.
Compendio de la invención
En un primer aspecto, la presente invención se refiere a un método in vitro para predecir la respuesta de un sujeto diagnosticado con cáncer de mama al tratamiento con un taxano y quimioterapia con un antimetabolito, un agente intercalante y un agente alquilante del ADN, en adelante el método de la invención, que comprende determinar los niveles de expresión de los genes identificados en la Tabla 1 y en la Tabla 2 en una muestra de tejido tumoral procedente de dicho sujeto, en donde un aumento de la expresión de los genes identificados en la Tabla 1 y una disminución de la expresión de los genes identificados en la Tabla 2 con respecto a un valor de referencia es indicativo de una mejor respuesta al tratamiento.
Breve descripción de las figuras
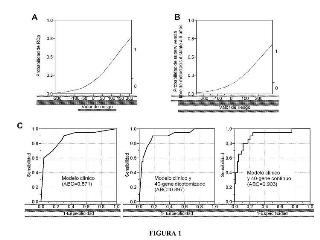
La Figura 1 muestra la relación entre el test 40 genes y la respuesta a quimioterapia T/FAC. (A) Valor de Riesgo derivado del test 40 genes frente a probabilidad de RCp. El eje Y a la derecha muestra la proporción de pacientes RCp (0) versus pacientes ER (1). (B) Valor de Riesgo derivado del test 40 genes frente a probabilidad de supervivencia libre de metástasis distante a 5 años en los datasets de Loi (GSE6532 y GSE9195). El eje Y a la derecha muestra la proporción de pacientes que desarrollaron metástasis distante a 5 años (0) versus aquellos pacientes que no (1). (C) Análisis de...
Reivindicaciones:
1. Un método in vitro para predecir la respuesta de un sujeto diagnosticado con cáncer de mama al tratamiento con un taxano y quimioterapia con un antimetabolito, un agente intercalante y un agente alquilante del ADN, que comprende determinar los niveles de expresión de los genes identificados en la Tabla 1 y en la Tabla 2 en una muestra de tejido tumoral procedente de dicho sujeto, en donde un aumento de la expresión de los genes identificados en la Tabla 1 y una disminución de la expresión de los genes identificados en la Tabla 2 con respecto a un valor de referencia es indicativo de una mejor respuesta al tratamiento.
2. Método según la reivindicación 1, en donde el taxano es paclitaxel.
3. Método según cualquiera de las reivindicaciones 1 a 2, en donde el antimetabolito es 5-fluorouracilo.
4. Método según cualquiera de las reivindicaciones 1 a 3, en donde el agente intercalante es adriamicina.
5. Método según cualquiera de las reivindicaciones 1 a 4, en donde el agente alquilante del ADN es ciclofosfamida.
6. Método según cualquiera de las reivindicaciones 1 a 5, en donde el sujeto diagnosticado con cáncer de mama presenta niveles normales de HER-2.
7. Método según cualquiera de las reivindicaciones 1 a 6, en donde el cáncer de mama es cáncer de mama avanzado.
8. Método según cualquiera de las reivindicaciones 1 a 7, en donde el tratamiento es neoadyuvante.
9. Método según cualquiera de las reivindicaciones 1 a 8, que comprende además determinar al menos un parámetro clínico seleccionado entre la edad del sujeto, el estado del receptor de estrógenos y el grado tumoral, en donde a menor edad del sujeto, sujetos negativos para receptor de estrógenos y grado tumoral más avanzado, es indicativo de una mejor respuesta al tratamiento.
10. Método según cualquiera de las reivindicaciones la 9, en donde la cuantificación de los niveles de expresión de los genes identificados en las Tablas 1 y 2 comprende la cuantificación del ARN mensajero (ARNm) de dichos genes, o un fragmento de dicho ARNm, el ADN complementario (ADNc) de dichos genes, o un fragmento de dicho ADNc, o sus mezclas.
11. Método según la reivindicación 10, en donde la cuantificación de los niveles de expresión de los genes identificados en las Tablas 1 y 2 se realiza mediante una reacción en cadena de la polimerasa (PCR) cuantitativa multiplex o un array de ADN o ARN.
12. Método según la reivindicación 11, donde la determinación de los niveles de expresión de los genes identificados en la Tabla 1 y la Tabla 2 se realiza mediante un array de ADN que comprende las sondas identificadas en las Tablas 3 y 4.
13. Método según la reivindicación 12, en donde la determinación de la predicción de la respuesta al tratamiento comprende un análisis de regresión de riesgos proporcionales de dicho pronóstico en función de los niveles de expresión de los genes identificados en la Tabla 1 y en la Tabla 2.
14. Método según la reivindicación 13, donde dicho análisis de regresión de riesgos proporcionales es un análisis de tipo Cox.
15. Método según la reivindicación 14, donde en dicho análisis de tipo Cox se establece la respuesta patológica completa (RCp) como variable pronóstico.
16. Método según la reivindicación 15, donde la determinación de la predicción de la respuesta al tratamiento se lleva a cabo aplicando la siguiente fórmula:
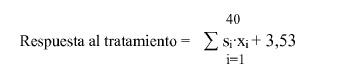
donde xi es el valor del nivel de expresión en log2 de cada uno de dichos genes identificados en la Tabla 1 y 2; y
si es el valor del estadístico de Wald del análisis de regresión de tipo Cox de cada uno de dichos genes identificados en la Tabla 1 y 2 según las reivindicaciones 14 ó 15,
en donde si dicho valor es mayor que cero entonces es indicativo de una mejor respuesta al tratamiento.
17. Método según la reivindicación 16, en donde la cuantificación de los niveles de expresión de los genes identificados en las Tablas 1 y 2 comprende la cuantificación de los niveles de proteína codificada por dichos genes o de una variante de la misma.
18. Método según la reivindicación 17, en donde la cuantificación de los niveles de proteína se realiza mediante western blot, ELISA o un array de proteínas.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]