MÉTODO PARA DIAGNOSTICAR CÁNCER DE PULMÓN DE CÉLULAS NO PEQUEÑAS.
Método para diagnosticar cáncer de pulmón de células no pequeñas (NSCLC) o una predisposición a desarrollar cáncer de pulmón de células no pequeñas en un sujeto,
que comprende determinar el nivel de expresión de un gen asociado a cáncer de pulmón de células no pequeñas en una muestra biológica derivada del sujeto, en el que un aumento de dicho nivel de expresión en comparación con un nivel de control normal de dicho gen indica que dicho sujeto padece o está en riesgo de desarrollar NSCLC, en el que dicho gen asociado a NSCLC es KIF11
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/JP2005/005613.
Solicitante: ONCOTHERAPY SCIENCE, INC..
Nacionalidad solicitante: Japón.
Dirección: 2-1, SAKADO 3-CHOME, TAKATSU-KU KAWASAKI-SHI, KANAGAWA 213-0012 JAPON.
Inventor/es: NAKAMURA, YUSUKE, NAKATSURU,Shuichi, DAIGO,Yataro.
Fecha de Publicación: .
Fecha Solicitud PCT: 18 de Marzo de 2005.
Clasificación Internacional de Patentes:
- A61K31/731 NECESIDADES CORRIENTES DE LA VIDA. › A61 CIENCIAS MEDICAS O VETERINARIAS; HIGIENE. › A61K PREPARACIONES DE USO MEDICO, DENTAL O PARA EL ASEO (dispositivos o métodos especialmente concebidos para conferir a los productos farmacéuticos una forma física o de administración particular A61J 3/00; aspectos químicos o utilización de substancias químicas para, la desodorización del aire, la desinfección o la esterilización, vendas, apósitos, almohadillas absorbentes o de los artículos para su realización A61L; composiciones a base de jabón C11D). › A61K 31/00 Preparaciones medicinales que contienen ingredientes orgánicos activos. › Carragenanos.
- C12Q1/68M6B
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania.
PDF original: ES-2360073_T3.pdf

Fragmento de la descripción:
Esta solicitud reivindica el beneficio de la solicitud provisional estadounidense n.º de serie 60/555.789 presentada el 23 de marzo de 2004.
Campo de la Invención
Esta invención se refiere al campo de la ciencia biológica, más específicamente al campo del diagnóstico y terapia contra el cáncer. En particular a métodos para diagnosticar cánceres de pulmón de células no pequeñas, dados a conocer en la presente memoria, usando genes, KIF11, GHSR1b, NTSR1 y FOXM1, que muestran expresión elevada en tales células cancerosas.
Antecedentes de la invención
El cáncer de pulmón es uno de los tumores humanos más comúnmente mortal. Se han notificado muchas alteraciones genéticas asociadas al desarrollo y la evolución del cáncer de pulmón. Aunque los cambios genéticos pueden ayudar a esfuerzos de pronóstico y predicciones de riesgo metastásico o responden a ciertos tratamientos, la información acerca de un número único o limitado de marcadores moleculares generalmente no proporciona resultados satisfactorios para el diagnóstico clínico de cáncer de pulmón de células no pequeñas (NSCLC) (Mitsudomi et al., Clin Cancer Res 6: 405563 (2000); Niklinski et al., Lung Cancer. 34 Suppl 2: S53-8 (2001); Watine, BMJ320: 379-80 (2000)). El NSCLC es con diferencia la forma más común, justificando casi 80% de tumores de pulmón (Society, A.C. Cancer Facts and Figures 2001 (2001)). La tasa de supervivencia de 10 años global sigue siendo tan sólo de 10% a pesar de los recientes avances en la terapia de modalidad múltiple, porque la mayor parte de los NSCLC no se diagnostican hasta estadios avanzados (Fry, W.A. et al., Cancer 86; 1867-76 (1999)). Aunque los regímenes de quimioterapia basados en platino se consideran los patrones de referencia para el tratamiento de NSCLC, esos fármacos pueden extender la supervivencia de pacientes con NSCLC avanzado sólo aproximadamente seis semanas (Non-small Cell Lung Cancer Collaborative Group, BMJ. 311: 899-909 (1995)). Están investigándose para esta enfermedad numerosas terapias seleccionadas como objetivo, incluyendo inhibidores de tirosina cinasa, pero hasta el momento se han logrado resultados prometedores en sólo un número limitado de pacientes y algunos receptores padecen reacciones adversas graves a (Kris M.N.R., Herbst R.S. Proc. Am. Soc. Clin. Oncol. 21: 292a(A1166) (2002)).
Se han notificado muchas alteraciones genéticas asociadas al desarrollo y a la evolución del cáncer de pulmón, pero siguen siendo inciertos los mecanismos moleculares precisos (Sozzi, G. Eur. J. Cancer 37: 63-73 (2001)). A lo largo de la última década han surgido agentes citotóxicos recientemente desarrollados incluyendo paclitaxel, docetaxel, gemcitabina y vinorelbina para ofrecer múltiples elecciones terapéuticas para pacientes con NSCLC avanzado; sin embargo, cada uno de los nuevos regímenes puede proporcionar beneficios de supervivencia modestos en comparación con las terapias a base de cisplatino (Schiller, J.H. et al., N. Engl. J. Med. 346: 92-98 (2002); Kelly, K. et al., J. Clin. Oncol. 19: 3210-3218 (2001)). Por tanto, los médicos esperan con entusiasmo nuevas estrategias terapéuticas, tales como el desarrollo de agentes seleccionados como diana moleculares.
Un enfoque eficaz para identificar moléculas desconocidas implicadas en rutas de carcinogénesis es el análisis sistemático de niveles de expresión de miles de genes en microalineamientos de ADNc (Kikuchi, T. et al., Oncogene 22: 2192-2205 (2003); Kakiuchi, S. et al., MoI. Cancer Res. 1: 485-499 (2003); Zembutsu, H. et al., Int. J. Oncol. 23: 29-39 (2003); Suzuki, C. et al., Cancer Res. 63: 7038-7041 (2003)) y puede poner de manifiesto dianas candidatas para el desarrollo de marcadores tumorales y fármacos anticancerígenos novedosos. Para aislar dianas moleculares novedosas para el diagnóstico, el tratamiento y la prevención de NSCLC, se prepararon poblaciones puras de células tumorales a partir de 37 tejidos de cáncer mediante microdisección con captura por láser y se analizaron perfiles de expresión de todo el genoma de células de NSCLC en un microalineamiento de ADNc que contenía 23.040 genes (Kikuchi, T. et al., Oncogene 22: 2192-2205 (2003)). En el transcurso de estos experimentos, se identificaron KOC1 (n.º de registro de GenBank NM_006547) y neuromedina U (NMU; n.º de registro de GenBank NM_006681) como genes que se sobreexpresaban frecuentemente en tumores de pulmón y eran indispensables para el crecimiento de células de NSCLC.
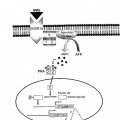
La comunicación entre células es un requisito previo para el desarrollo y mantenimiento de organismos multicelulares. Se han observado desde hace tiempo varios sistemas de intercambio de información intercelular tales como la sinapsis química, uniones de huecos y plasmodesmos en células vegetales, pero un nuevo sistema de transporte que implica una estructura nanotubular altamente sensible, nanotubos de tunelización (TNT) entre las células, se notificó sólo recientemente en células de mamífero (Rustom, A. et al., Science 303, 1007- 1010 (2004). Una estructura de este tipo facilitaría la transferencia selectiva de orgánulos y vesículas de membrana; por tanto los TNT en células somáticas de mamífero podrían contribuir a un sistema(s) de transporte entre células portando factores de transcripción o ribonucleopartículas (RNP), como en las plantas (Nakajima, K. et al., Nature 413, 307-311 (2001); Lucas, WJ. et al., Nat. Rev. MoI. Cell Biol. 2, 849-857 (2001)). Algunos investigadores han documentado interacciones entre algunas proteínas de unión a ARN y proteínas motoras como cinesina y dineína dentro de las células somáticas de mamífero, así como también transporte de ARNm intercelular en células embrionarias de mamífero (Brendza, R.R et al., Science 289, 21202122 (2000); Chennathukuzhi, V. et al., Proc. Natl. Acad. Sd. USA 100, 15566-15571 (2003); Villace, P. et al., Nucleic Acids Res. 32, 2411-2420 (2004). ; Morales, CR. et al. Dev. Biol.246, 480-494 (2002).). Sin embargo, no ha surgido ninguna notificación describiendo un sistema de transporte de ARNm intercelular en células somáticas de mamífero que implican un complejo de proteínas de unión a ARN y proteínas motoras.
Se ha notificado el fenómeno de localización de ARNm en ovocitos y embriones en desarrollo de Drosophila y Xenopus y en células somáticas tales como fibroblastos y neuronas (King, MX. et al., Bioessays 21: 546-557 (1999); Mowry, K.L., Cote, CA. FASEB J. 13: 435-445 (1999); Lasko, P. J. Cell Biol. 150: F51-56 (2000); Steward, O. Neuron 18: 9-12 (1997)). El ARNm de beta-actina (ACTB) se localiza en las laminillas conductoras de fibroblastos embrionarios de pollo (CEF) (Lawrence, J.B., Singer, R.H. Cell 45: 407-415 (1986)) y en el cono de crecimiento de neuronas en desarrollo (Bassell, GJ. et al., J. Neurosci. 18: 251-265 (1998)). La localización de ARNm de ACTB depende del zip-code, un elemento que actúa en cis ubicado en la UTR en 3' del ARNm (Kislauskis, E.H. et al., J. Cell Biol. 123: 165-172 (1993)). El factor que actúa en trans, proteína de unión a zip-code 1 (ZBP1), se purificó mediante afinidad con el zip-code de ARNm de ACTB (Ross, A.F. et al., MoI. Cell Biol. 17, 2158-2165 (1997)). Tras la identificación de ZBP1, se identificaron homólogos adicionales en un amplio rango de organismos incluyendo Xenopus, Drosophila, ser humano y ratón (Mueller-Pillasch, F. et al., Oncogene 14: 2729-2733 (1997); Deshler, J.O. et al., Science 276: 1128-1131 (1997); Doyle,
G.A. et al., Nucleic Acids Res. 26: 5036-5044 (1998)). Los miembros de la familia de ZBP1 se expresan en fibroblastos embrionarios germinales y en varios tipos de cáncer (Mueller-Pillasch, F. et al., Oncogene 14: 2729-2733 (1997); Mueller, F. et al., Br. J. Cancer 88; 699-701 (2003)). Las proteínas de tipo ZBP1 contienen dos motivos de reconocimiento de ARN (RRM) en la parte NH2-terminal de la proteína y cuatro dominios de homología de hnRNP K (KH) en el extremo COOH-terminal.
KOC1 (alias proteína de unión a ARNm de IGF-II 3: IMP-3) es una de las IMP (IMP-I, IMP-2 y IMP-3), que pertenecen a los miembros de la familia ZBP1 y muestran múltiples uniones a ARNm de IGF-II líder 3 y el ARN de H19 con huella genética recíproca (Mueller-Pillasch, F. et al., Oncogene 14: 2729-2733 (1997)). Aunque inicialmente se notificó que KOC1 se sobreexpresaba en cáncer pancreático (Mueller-Pillasch, F. et al., Oncogene 14: 2729-2733 (1997); Mueller, F. et al., Br. J. Cancer 88: 699-701 (2003)), su función precisa en células de cáncer o incluso en células somáticas de mamífero normales sigue siendo incierta.
KOC1 es ortóloga a la proteína de unión a ARN de Vg1 de Xenopus (Vg1RBP/Vera), que media... [Seguir leyendo]
Reivindicaciones:
1. Método para diagnosticar cáncer de pulmón de células no pequeñas (NSCLC) o una predisposición a desarrollar cáncer de pulmón de células no pequeñas en un sujeto, que comprende determinar el nivel de expresión de un gen asociado a cáncer de pulmón de células no pequeñas en una muestra biológica derivada del sujeto, en el que un aumento de dicho nivel de expresión en comparación con un nivel de control normal de dicho gen indica que dicho sujeto padece o está en riesgo de desarrollar NSCLC, en el que dicho gen asociado a NSCLC es KIF11.
2. Método de acuerdo con la reivindicación 1, en el que dicho aumento es al menos 10% mayor que dicho nivel de control normal.
3. Método de acuerdo con la reivindicación 1 ó 2, en el que dicho método comprende además determinar el nivel de expresión de una pluralidad de genes asociados a NSCLC.
4. Método según una cualquiera de las reivindicaciones 1 a 3, en el que dicho nivel de expresión se determina mediante un método cualquiera seleccionado del grupo que consiste en:
(1) detectar ARNm del gen asociado a NSCLC;
(2) detectar la proteína codificada por el gen asociado a NSCLC; y
(3) detectar la actividad biológica de la proteína codificada por el gen asociado a NSCLC.
5. Método según una cualquiera de las reivindicaciones 1 a 3, en el que dicho nivel de expresión se determina detectando la hibridación de una sonda de gen asociado a NSCLC con un transcrito génico de dicha muestra biológica derivada del paciente.
6. Método de acuerdo con la reivindicación 5, en el que dicha etapa de hibridación se lleva a cabo en un alineamiento de ADN.
7. Método según una cualquiera de las reivindicaciones 1 a 6, en el que dicha muestra biológica comprende esputo o sangre.
8. Método in vitro para identificar un compuesto para tratar o prevenir NSCLC, que comprende las etapas de:
(1) poner en contacto una célula de prueba que expresa dicho gen asociado a NSCLC con un compuesto de prueba;
(2) detectar el nivel de expresión de dicho gen asociado a NSCLC; y
(3) determinar el compuesto que suprime dicho nivel de expresión en comparación con un nivel de control normal de dicho gen,
en el que dicho gen asociado a NSCLC es KIF11 y dicho compuesto es para su uso para tratar o prevenir NSCLC.
9. Método de acuerdo con la reivindicación 8, en el que dicha célula de prueba es una célula de NSCLC.
10. Método in vitro para seleccionar un compuesto para tratar o prevenir NSCLC, comprendiendo dicho método las etapas de:
(1) poner en contacto un compuesto de prueba con un polipéptido de KIF11,
(2) detectar la actividad de unión entre el polipéptido y el compuesto de prueba; y
(3) seleccionar un compuesto que se une al polipéptido.
11. Método in vitro para seleccionar un compuesto para tratar o prevenir NSCLC, comprendiendo dicho método las etapas de:
(a) poner en contacto un compuesto de prueba con un polipéptido codificado por un polinucleótido de KIF11;
(b) detectar la actividad biológica del polipéptido de la etapa (a); y
(c) seleccionar un compuesto que suprime la actividad biológica del polipéptido de KIF11 en comparación con la actividad biológica detectada en ausencia del compuesto de prueba
en el que dicho compuesto es para su uso para tratar o prevenir NSCLC.
12. Método de acuerdo con la reivindicación 11, en el que dicha actividad biológica es la actividad de proliferación
celular.
13. Método in vitro para seleccionar un compuesto para tratar o prevenir NSCLC, comprendiendo dicho método las etapas de:
(1) poner en contacto un compuesto de prueba con un célula que expresa KIF11; y
(2) seleccionar un compuesto que reduce el nivel de expresión de KIF11, en el que dicho compuesto es para su uso para tratar o prevenir NSCLC.
14. Método de acuerdo con la reivindicación 13, en el que dicha célula es una célula de NSCLC.
15. Método in vitro para seleccionar un compuesto para tratar o prevenir NSCLC, comprendiendo dicho método las etapas de:
(1) poner en contacto un compuesto de prueba con una célula en la que se ha introducido un vector que comprende la región reguladora transcripcional de KIF11 y un gen indicador que se expresa bajo el control de la región reguladora transcripcional;
(2) medir la actividad de dicho gen indicador; y
(3) seleccionar un compuesto que reduce el nivel de expresión de dicho gen indicador, tal como se compara con un control,
en el que dicho compuesto es para su uso para tratar o prevenir NSCLC.
16. Método in vitro para seleccionar un compuesto para tratar o prevenir NSCLC, comprendiendo dicho método las etapas de:
(1) poner en contacto un polipéptido de KIF11 o equivalente funcional del mismo con polipéptido de KOC1 o equivalente funcional del mismo en presencia de un compuesto de prueba;
(2) detectar la unión entre los polipéptidos; y
(3) seleccionar el compuesto de prueba que inhibe la unión entre los polipéptidos.
17. Método de acuerdo con la reivindicación 16, en el que el equivalente funcional de polipéptido de KIF11 comprende una secuencia de aminoácidos del dominio de unión a KOC1.
18. Método de acuerdo con la reivindicación 16, en el que el equivalente funcional de polipéptido de KOC1 comprende una secuencia de aminoácidos del dominio de unión a KIF11.
19. Método in vitro para medir la actividad de transporte de ARN de un polipéptido, comprendiendo dicho método las etapas de:
(a) poner en contacto un polipéptido seleccionado del grupo que consiste en:
i. un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 2 (KIF11);
ii. un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 2 en la que uno o más aminoácidos se sustituyen, delecionan o insertan, y dicho polipéptido tiene una actividad biológica equivalente al polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 2;
iii. un polipéptido que comprende la secuencia de aminoácidos que tiene al menos aproximadamente 80% de homología con SEC ID Nº: 2; y
iv. un polipéptido codificado por un polinucleótido que hibrida en condiciones rigurosas con un polinucleótido que consiste en la secuencia de nucleótidos de SEC ID Nº: 1, en el que el polipéptido tiene una actividad biológica equivalente a un polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 2;
con un ARN que va a transportarse y en la condición que puede formar el transportador de ARN;
(b) detectar el nivel del ARN transportado; y
(c) medir la actividad de transporte de ARN correlacionando el nivel del ARN transportado de la etapa (b) con la actividad de transporte de ARN.
20. Método de acuerdo con la reivindicación 19, en el que la condición que puede formar el transportador de ARN se proporciona en la existencia del polipéptido de KOC1 o equivalente funcional del mismo.
21. Método de acuerdo con la reivindicación 20, en el que el equivalente funcional del polipéptido de KOC1 es un polipéptido seleccionado del grupo que consiste en:
(i) un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 105 (KOC1);
(ii) un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 105 en la que uno o más aminoácidos se sustituyen, delecionan o insertan, y dicho polipéptido tiene una actividad biológica equivalente al polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 105;
(iii) un polipéptido que comprende la secuencia de aminoácidos que tiene al menos aproximadamente 80% de homología con SEC ID Nº: 105; y
(iv) un polipéptido codificado por un polinucleótido que hibrida en condiciones rigurosas con un polinucleótido que consiste en la secuencia de nucleótidos de SEC ID Nº: 104, en el que el polipéptido tiene una actividad biológica equivalente a un polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 105;
22. Método in vitro para identificar un agente que modula la actividad de transporte de ARN, comprendiendo dicho método las etapas de:
(a) poner en contacto el agente con un polipéptido selecccionado del grupo que consiste en:
i. un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 2 (KIF11);
ii. un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 2 en la que uno o más aminoácidos se sustituyen, delecionan o insertan, y dicho polipéptido tiene una actividad biológica equivalente al polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 2;
iii. un polipéptido que comprende la secuencia de aminoácidos que tiene al menos aproximadamente 80% de homología con SEC ID Nº: 2; y
iv. un polipéptido codificado por un polinucleótido que hibrida en condiciones rigurosas con un polinucleótido que consiste en la secuencia de nucleótidos de SEC ID Nº: 1, en el que el polipéptido tiene una actividad biológica equivalente a un polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 2;
con un ARN que va a transportarse y en la condición que puede formar el transportador de ARN;
(b) detectar el nivel del ARN transportado; y
(c) comparar el nivel del ARN transportado con un nivel de control en ausencia del agente indicando un aumento o disminución del nivel del ARN transportado en comparación con el nivel de control que el compuesto de prueba modula la actividad de transporte de ARN.
23. Kit para detectar una actividad de un compuesto de prueba para regular la actividad de transporte de ARN, comprendiendo dicho kit una célula aislada que expresa los componentes de a a d, y un medio de cultivo que soporta el crecimiento celular,
(a) un polipéptido seleccionado del grupo que consiste en:
i. un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 2 (KIF11);
ii. un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 2 en la que uno o más aminoácidos se sustituyen, delecionan o insertan, y dicho polipéptido tiene una actividad biológica equivalente al polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 2;
iii. un polipéptido que comprende la secuencia de aminoácidos que tiene al menos aproximadamente 80% de homología con SEC ID Nº: 2; y
iv. un polipéptido codificado por un polinucleótido que hibrida en condiciones rigurosas con un polinucleótido que consiste en la secuencia de nucleótidos de SEC ID Nº: 1, en el que el polipéptido tiene una actividad biológica equivalente a un polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 2;
(b) un polipéptido seleccionado del grupo que consiste en:
i. un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 105 (KOC1);
ii. un polipéptido que comprende la secuencia de aminoácidos de SEC ID Nº: 105, en la que uno o más aminoácidos se sustituyen, delecionan o insertan, y dicho polipéptido tiene una actividad biológica equivalente al polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 105;
iii. un polipéptido que comprende la secuencia de aminoácidos que tiene al menos aproximadamente 80% de homología con SEC ID Nº: 105; y
iv. un polipéptido codificado por un polinucleótido que hibrida en condiciones rigurosas con un polinucleótido que consiste en la secuencia de nucleótidos de SEC ID Nº: 104, en el que el polipéptido tiene una actividad biológica equivalente a un polipéptido que consiste en la secuencia de aminoácidos de SEC ID Nº: 105; y
(c) un ARN que va a transportarse; y
(d) DCTN1.
24. Uso de
(i) una composición antisentido, comprendiendo dicha composición una secuencia de nucleótidos complementaria a una secuencia codificante de KIF11;
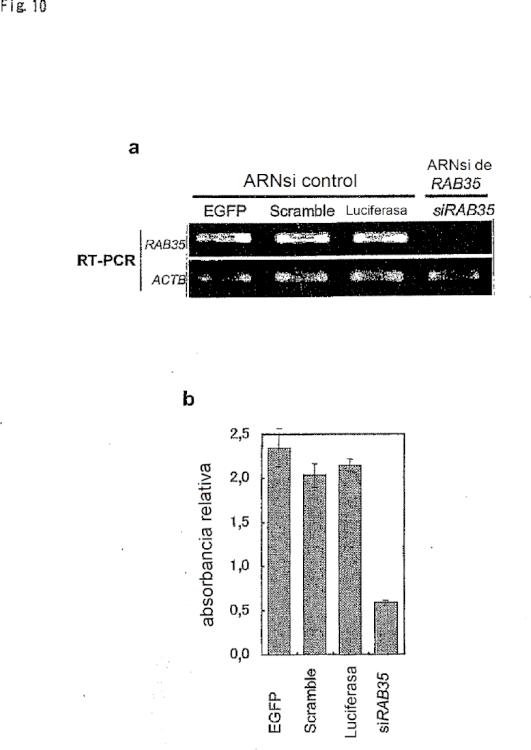
(ii) una composición de ARNsi que comprende un ARNsi, en el que dicha composición reduce la expresión de KIF11; o
(iii) un anticuerpo o fragmento del mismo que se une a un polipéptido codificado por KIF11;
para la preparación de una composición farmacéutica para tratar o prevenir NSCLC en un sujeto.
25. Uso de acuerdo con la reivindicación 24, en el que el ARNsi es una cadena sentido que comprende la secuencia de nucleótidos seleccionada del grupo de SEC ID Nº: 32, 33 y 34 como secuencia diana.
26. Uso de acuerdo con la reivindicación 25, en el que el ARNsi tiene la fórmula general 5'-[A]-[B]-[A']-3'
en la que [A] es una secuencia de ribonucleótidos que corresponde a una secuencia seleccionada del grupo que consiste en SEC ID Nº: 32, 33 y 34; [B] es una secuencia de ribonucleótidos que consiste en de 3 a 23 nucleótidos; y [A'] es una secuencia de ribonucleótidos complementaria a [A].
27. Uso de un polipéptido codificado por KIF11 o un fragmento inmunológicamente activo de dicho polipéptido, o un polinucleótido que codifica para el polipéptido para la preparación de una vacuna para tratar o prevenir NSCLC en un sujeto.
28. Molécula bicatenaria que comprende una cadena sentido y una cadena antisentido, en la que la cadena sentido comprende una secuencia de ribonucleótidos que corresponde a una secuencia diana de KIF11 seleccionada del grupo que consiste en SEC ID Nº: 32, 33 y 34, y en la que la cadena antisentido comprende una secuencia de ribonucleótidos que es complementaria a dicha cadena sentido, hibridando dicha cadena sentido y dicha cadena antisentido una con otra para formar dicha molécula bicatenaria, e inhibiendo dicha molécula bicatenaria, cuando se introduce en una célula que expresa un gen KIF11, la expresión de dicho gen, siendo además la molécula bicatenaria un oligonucleótido de entre aproximadamente 19 y aproximadamente 25 nucleótidos de longitud.
29. Molécula bicatenaria de acuerdo con la reivindicación 28, siendo dicha molécula bicatenaria un único transcrito de ribonucleótidos que comprende la cadena sentido y la cadena antisentido unidas por medio de una secuencia de ribonucleótidos monocatenaria.
30. Vector que codifica para la molécula bicatenaria según una cualquiera de las reivindicaciones 28 a 29.
31. Vector de acuerdo con la reivindicación 30, codificando el vector para un transcrito que tiene una estructura secundaria y comprende la cadena sentido y la cadena antisentido.
32. Vector de acuerdo con la reivindicación 31, en el que el transcrito comprende además una secuencia de ribonucleótidos monocatenaria que une dicha cadena sentido y dicha cadena antisentido.
33. Vector de acuerdo con la reivindicación 32, en el que dicho polinucleótido tiene la fórmula general
5'-[A]-[B]-[A']-3'
en la que [A] es una secuencia de nucleótidos seleccionada del grupo que consiste en SEC ID Nº: 32, 33 y 34; [B] es una secuencia de nucleótidos que consiste en de 3 a 23 nucleótidos; y [A'] es una secuencia de nucleótidos complementaria a [A].
34. Composición farmacéutica que comprende un ARNsi frente al gen KIF11, en la que el ARNsi comprende una cadena sentido que comprende la secuencia de nucleótidos de SEC ID Nº: 32, 33 y 34 como secuencia diana y que tiene de aproximadamente 19 a aproximadamente 25 nucleótidos de longitud.
35. Método para predecir un pronóstico de NSCLC, comprendiendo el método las etapas de:
5 (a) detectar el nivel de expresión de KIF11 en una muestra recogida de un sujeto cuyo pronóstico de NSCLC va a predecirse, e
(b) indicar un mal pronóstico cuando se detecta una elevación del nivel de expresión de KIF11.
36. Método de acuerdo con la reivindicación 35, en el que el nivel de expresión se detecta mediante uno cualquiera del método seleccionado del grupo que consiste en:
10 (a) detectar el ARNm que codifica para la secuencia de aminoácidos de SEC ID Nº: 2 (KIF11),
(b) detectar la proteína que comprende la secuencia de aminoácidos de SEC ID Nº: 2 (KIF11), y
(c) detectar la actividad biológica de la proteína que comprende la secuencia de aminoácidos de SEC ID Nº: 2 (KIF11).
Patentes similares o relacionadas:
Composición para el tratamiento de la cavidad de la garganta/orofaringe, del 25 de Marzo de 2020, de MARIA CLEMENTINE MARTIN KLOSTERFRAU VERTRIEBSGESELLSCHAFT MBH: Composición en forma de una dosificación líquida para su utilización en el tratamiento profiláctico o terapéutico de enfermedades inflamatorias de la cavidad oral y/o […]
Composición que comprende carragenina tipo iota contra conjuntivitis viral, del 24 de Abril de 2019, de VISUfarma B.V: Una composición farmacéutica que comprende carragenina tipo iota como un ingrediente antiviral activo en una cantidad eficaz antiviral, para su […]
Composición, en particular composición farmacéutica, en particular para el tratamiento de ronquera o dolor de garganta, del 8 de Junio de 2016, de MARIA CLEMENTINE MARTIN KLOSTERFRAU VERTRIEBSGESELLSCHAFT MBH: Una composición, en particular una composición farmacéutica, de manera preferida en forma de una dosificación sólida o líquida, en particular sólida, en particular para el […]
 Composiciones antimicrobianas acuosas que contienen carragenanos, del 12 de Noviembre de 2015, de THE POPULATION COUNCIL, INC.: Composición antimicrobiana acuosa que comprende una cantidad eficaz de un agente antimicrobiano que comprende carragenanos los cuales son […]
Composiciones antimicrobianas acuosas que contienen carragenanos, del 12 de Noviembre de 2015, de THE POPULATION COUNCIL, INC.: Composición antimicrobiana acuosa que comprende una cantidad eficaz de un agente antimicrobiano que comprende carragenanos los cuales son […]
 Carragenina modificada por un procedimiento de intercambio iónico, del 6 de Mayo de 2015, de CP KELCO U.S., INC.: Una composicióncaracterizadaporque comprende un iota carragenano que ha sido sometido a un proceso de intercambio iónico, en donde el iota […]
Carragenina modificada por un procedimiento de intercambio iónico, del 6 de Mayo de 2015, de CP KELCO U.S., INC.: Una composicióncaracterizadaporque comprende un iota carragenano que ha sido sometido a un proceso de intercambio iónico, en donde el iota […]
 Biopolímeros antialérgicos marinos, del 11 de Diciembre de 2013, de MARINOMED BIOTECHNOLOGIE GMBH: Iota-carragenina en una cantidad eficaz antialérgica para su uso como medicamento en el tratamiento profilácticoo terapéutico de la rinitis alérgica, la conjuntivitis […]
Biopolímeros antialérgicos marinos, del 11 de Diciembre de 2013, de MARINOMED BIOTECHNOLOGIE GMBH: Iota-carragenina en una cantidad eficaz antialérgica para su uso como medicamento en el tratamiento profilácticoo terapéutico de la rinitis alérgica, la conjuntivitis […]
EMPLEO DEL HIDROLIZADO DE COLÁGENO PARA DISMINUIR LA VISCOSIDAD DE UNA COMPOSICIÓN QUE CONTIENE GOMA GUAR, del 27 de Julio de 2011, de NESTEC S.A.: Empleo del hidrolizado de colágeno para disminuir la viscosidad de una composición que contiene una o más fibra(s) soluble(s) viscosa(s), en donde dicha fibra soluble […]
CORRECTOR DEL TRANSITO INTESTINAL, del 14 de Octubre de 2010, de LAINCO, S.A.: La presente invención se refiere a una composición correctora del tránsito intestinal que comprende al menos un hidrocoloide expandido sobre una carga inerte. La presente […]