Procedimiento de integración localizada de múltiples copias de un gen de interés en una cepa de Yarrowia.
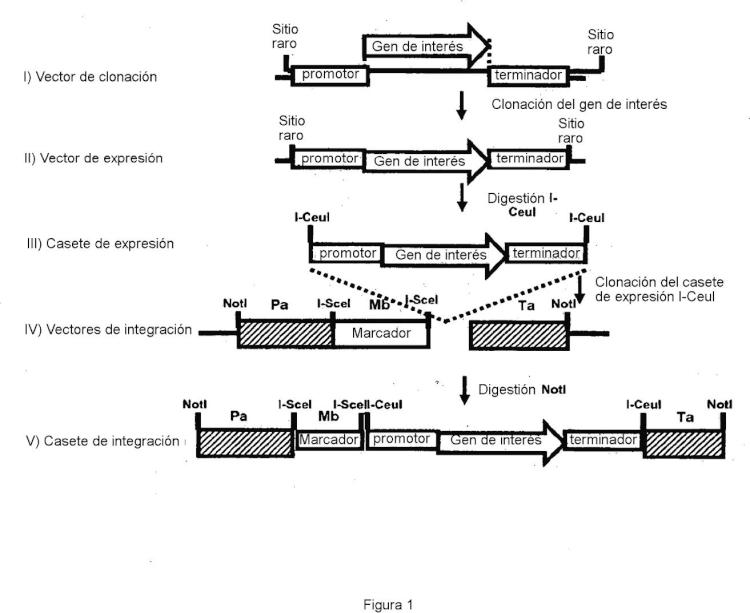
Procedimiento de integración localizada de por lo menos tres copias de un gen de interés en el genoma de una cepa de Yarrowia que comprende las etapas de:
a) puesta en cultivo de una cepa de Yarrowia que comprende por lo menos tres deleciones seleccionadas de entre Ura3-302, Leu2-270, Gut2-744 y Ade2-844, correspondiendo el fenotipo asociado a cada una de estas deleciones a una auxotrofia para dicha cepa, independientemente el uno del otro de estos por lo menos tres genes;
b) transformación de dicha cepa de Yarrowia auxótrofa con por lo menos tres vectores recombinantes que comprenden unos marcadores de selección que permiten, para dicha cepa de Yarrowia, la complementación de la auxotrofia resultante de cada una de dichas deleciones, comprendiendo dichos vectores recombinantes cada uno:
i) la secuencia de dicho gen de interés,
ii) un marcador de selección, y
iii) dos secuencias de ADN que enmarcan la secuencia de dicho gen de interés y dicho marcador de selección, siendo dichas secuencias de ADN homólogas a las secuencias que corresponden a los extremos del sitio de integración localizado en el genoma de dicha cepa de Yarrowia de manera que se permita la integración localizada de dicho vector recombinante por recombinación homóloga;
selección sobre un medio mínimo, de las levaduras que han integrado dichos por lo menos tres vectores recombinantes.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2009/051332.
Solicitante: INSTITUT NATIONAL DE LA RECHERCHE AGRONOMIQUE.
Nacionalidad solicitante: Francia.
Dirección: 147 Rue de l'Université 75007 Paris FRANCIA.
Inventor/es: NICAUD, JEAN-MARC, FUDALEJ,FRANCK, NEUVEGLISE,CÉCILE, BECKERICH,JEAN-MARIE.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/81 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › para levaduras.
PDF original: ES-2553383_T3.pdf

Patentes similares o relacionadas:
Sistema de expresión génica inducible por D-aminoácido para Rhodosporidium y Rhodotorula, del 6 de Mayo de 2020, de TEMASEK LIFE SCIENCES LABORATORY LIMITED: Un sistema de expresión génica inducible por D-aminoácido operable en una célula fúngica transgénica que comprende una construcción de ácido nucleico que comprende un […]
Secuencias de expresión, del 12 de Febrero de 2020, de LONZA LTD.: Un ácido nucleico aislado que codifica un líder unido de forma operable a una secuencia de ácidos nucleicos que codifica una proteína de interés (PDI), en donde el ácido nucleico […]
Cartucho de expresión para la trasformación de una célula eucariótica, método para transformar una célula eucariótica, organismo genéticamente modificado, y procedimiento para la producción de biocombustibles y/o compuestos bioquímicos y biocombustibles producidos de ese modo, del 13 de Noviembre de 2019, de Biocelere Agroindustrial Ltda: Un casete de expresión para transformar una célula eucariótica caracterizado porque comprende una combinación de los siguientes casetes de expresión: […]
Promotor regulable, del 8 de Noviembre de 2019, de LONZA LTD.: Un método para producir una proteína de interés (PDI) mediante el cultivo de una línea de células eucariotas recombinantes que comprenden una construcción […]
Métodos y composiciones para producir escualeno empleando levaduras, del 2 de Octubre de 2019, de Cibus Europe B.V: Una composición que comprende una levadura transformada genéticamente, en donde dicha levadura transformada genéticamente expresa una o más enzimas modificadas […]
Célula de levadura que consume acetato, del 25 de Septiembre de 2019, de DSM IP ASSETS B.V.: Una célula de levadura que está modificada genéticamente, que comprende: a) una alteración de una o más aldehído deshidrogenadas (E.C:1.2.1.4) nativas de la […]
Transformante y procedimiento para la producción del mismo, y procedimiento para la producción de ácido láctico, del 28 de Agosto de 2019, de JMTC Enzyme Corporation: Transformante de Schizosaccharomyces pombe que comprende 3 copias de un gen de lactato deshidrogenasa humano, en el que las 3 copias del gen de lactato deshidrogenasa humano […]
Anticuerpo de PD-1, fragmento de unión a antígeno del mismo y aplicación médica del mismo, del 21 de Agosto de 2019, de Shanghai Hengrui Pharmaceutical Co. Ltd: Un anticuerpo de PD-1, o un fragmento de unión a antígeno del mismo, que comprende: una región variable de cadena ligera que comprende […]