Procedimiento para la determinación de dosis de un antibiótico in vitro por medio de mecanismos de regulación genética y kit de diagnóstico correspondiente.
Kit de detección y/o de cuantificación de un antibiótico (C), perteneciente a la familia de las tetraciclinas o de lasvirginiamicinas, presente en una muestra de análisis, por medio de un receptor (A), estando dicho kit destinado auna puesta en práctica in vitro, es decir sin expresarse en ningún sistema biológico o celular, caracterizado porqueconsiste en los siguientes reactivos:
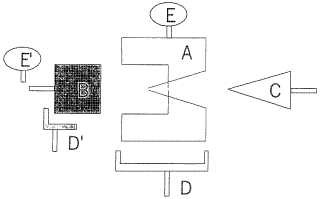
- a modo de receptor (A), un represor genético respectivamente de tetraciclina o de virginiamicina, que presentala propiedad de presentar al menos un primer sitio de reconocimiento específico para el antibiótico (C) y unsegundo sitio de reconocimiento específico para una secuencia nucleotídica operadora (B) correspondiente alrepresor, siendo el segundo sitio distinto del primer sitio, y que presenta la propiedad de que la fijación de dichasecuencia nucleotídica (B) en el receptor (A) en dicho segundo sitio de reconocimiento específico se modulanegativamente mediante la fijación del antibiótico (C) en el receptor (A) en dicho primer sitio de reconocimientoespecífico, es decir que la fijación de dicha secuencia nucleotídica (B) únicamente tiene lugar en ausencia defijación del antibiótico (C) en el receptor (A);
- dicha secuencia nucleotídica (B), monocatenaria o bicatenaria.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/BE2002/000183.
Solicitante: UNISENSOR S.A.
Nacionalidad solicitante: Bélgica.
Dirección: Route de Bonsgnée 145 4120 Rotheux-Rimiere BELGICA.
Inventor/es: GRANIER,BENOIT, LEPAGE,SOPHIE.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- G01N33/543 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › con un soporte insoluble para la inmovilización de compuestos inmunoquímicos.
PDF original: ES-2394200_T3.pdf
Fragmento de la descripción:
Procedimiento para la determinación de dosis de un antibiótico in vitro por medio de mecanismos de regulación genética y kit de diagnóstico correspondiente.
Objetivo de la invención La presente invención se refiere a un procedimiento que utiliza total o parcialmente in vitro elementos conocidos pertenecientes a los mecanismos de regulación de la expresión de los genes, que tendría principalmente como campo de aplicación el reconocimiento y la determinación de dosis de moléculas específicas tales como antibióticos.
La invención también se refiere a un kit de diagnóstico que pone en práctica el procedimiento.
Antecedentes científicos Los sistemas de regulación genética naturales se definen como mecanismos que pueden modular la expresión de los genes en función de la presencia o la ausencia de determinadas moléculas específicas en el entorno celular.
La expresión de los genes puede controlarse a lo largo de las etapas de transcripción, de elongación y de traducción de los ácidos nucleicos (figura 1) .
Se distingue el control negativo y el control positivo de la transcripción. En el primer caso, un receptor proteico, denominado represor, impide que el gen se exprese fijándose sobre el operador del mismo e impide así que la ARN polimerasa inicie la transcripción en el promotor. Este sistema es muy frecuente en las bacterias (TetR, VarR, LacI) . En el segundo caso, se necesita un activador o factor de transcripción que se fija en el ADN para iniciar la transcripción. Este sistema es frecuente en las eucariotas (por ejemplo receptores hormonales) pero también está presente en las bacterias, como es el caso del activador AmpR que regula la transcripción de la º-lactamasa AmpC en las bacterias Gram negativas tales como Citrobacter freundei (Jacobs et al., Cell, vol. 88 (1997) , págs. 823-832) o de PurR (Schumacher et al., Cell, vol. 83 (1995) , págs. 147-155) . De manera análoga, la traducción también puede regularse mediante la fijación de un represor en el ARNm que impide que el ribosoma inicie la traducción en proteínas. En las eucariotas, la elongación del ARNm puede regularse además durante las etapas de modificación, “corte y empalme”, transporte o estabilidad.
Los reguladores son entidades proteicas que pueden encontrarse en 2 estados: o bien en un estado activado (RTA) en el que pueden, fijándose en el ADN o el ARNm, regular de manera positiva (+) o negativa (-) la expresión de los genes, y por tanto la síntesis de las proteínas, o bien en un estado inactivado (RTI) en el que ya no pueden unirse a una secuencia nucleotídica particular. La activación se debe generalmente a una transición alostérica del represor en función de la presencia o la ausencia de un ligando de alta afinidad en un sitio específico del regulador. Es el caso de los reguladores bacterianos pertenecientes a la familia LacI (Weickert and Adhya, J. Biol. Chem., vol. 267 (1992) , págs. 15869-74) o de los receptores hormonales en las eucariotas. Otros fenómenos bioquímicos tales como la oxidación, la fosforilación o la glicosilación también pueden “iniciar” estos reguladores.
En este caso del mecanismo asociado a la fijación de un ligando, los reguladores presentan dos sitios de fijación:
-por un lado un sitio de fijación al ADN con frecuencia caracterizado por un motivo expuesto de hélice (-giro º hélice ( (HTH) , característico de las proteínas que se unen al ADN;
-por otro lado un sitio de fijación al ligando.
La presencia del ligando, denominado efector, en el regulador induce generalmente una modificación de conformación del mismo que va a permitir (o por el contrario impedir) la fijación de este regulador sobre una secuencia nucleotídica determinada. Estos efectores actúan por tanto como inductores o co-represores. Estos casos más frecuentes se ilustran en las figuras 2A y 2B.
Es el caso de la purina, co-represor necesario para la fijación del represor dimérico PurR, perteneciente a la familia de los reguladores transcripcionales bacterianos LacI, sobre el operador PurF (Schumacher et al., Cell, vol. 83 (1995) , págs. 147-155; Schumacher et al., Science, vol. 266 (1994) , págs. 763-770) o del L-triptófano, co-represor del dímero Trp (Otwinowski et al., Nature, vol. 335 (1988) , págs. 321-329) .
La mayor parte de los otros reguladores bacterianos de la familia de LacI, con la excepción de PurR, reprimen una ruta catabólica y no biosintética, siendo la afinidad de los reguladores por el operador superior en ausencia de ligando en el represor. Por tanto, los reguladores TetR y VarR son dímeros que también pertenecen a la familia LacI que intervienen en los mecanismos de resistencia bacteriana frente a los antibióticos tetraciclinas y virginiamicinas respectivamente. Estos represores, que presentan por otro lado una gran homología de secuencia, pueden fijarse a los operadores “tet” y “var” únicamente en ausencia de sus ligandos respectivos, tetraciclina o derivados y virginiamicina S considerados efectores inductores (Hillen et al., Ann. Rev. Microbiol., vol. 48 (1994) , pág. 345; Namwat et al., J. Bac., vol. 183 (mar. de 2001) , págs. 2025-2031) .
En presencia de un inductor fijado, los reguladores “sueltan” el operador e inmediatamente se eleva la represión. Se observa una síntesis de TetA y de VarS, transportadores de membrana que catalizan el transporte de los antibióticos fuera de la célula. Este sistema necesita una sensibilidad muy controlada ya que regula la toxicidad del transportador que a la vez va a impedir que el antibiótico alcance su diana (el ribosoma) pero también puede soltar otros iones no específicos fuera de la célula, lo que también es mortal para la célula. Por tanto, TetR debe mantener una gran represión sobre TetA hasta que esté presente un bajo nivel de tetraciclina (Tc) .
En cuanto a TetR, se conoce la estructura tridimensional en presencia de Tc así como en presencia del operador (Hinrichs et al., Science, vol. 264 (1994) , págs. 418-420; Kisker et al., J. Mol. Biol., vol. 247 (1995) , págs. 260-280; Orth et al., Nature Structural Biol., vol. 7 (2000) , págs. 215-219) . Permite comprender mejor la maquinaria molecular. Se requiere una orientación precisa de una cadena lateral de los aminoácidos terminales para permitir un contacto de alta afinidad entre la región operadora del ADN y el regulador. Cuando se fija la tetraciclina, la distancia de centroa centro entre los dos motivos de HTH del dímero de TetR aumenta 5 Å y esta ligera modificación de estructura es suficiente para interrumpir el contacto de alta afinidad con el operador. Los estudios in vitro muestran que la afinidad de TetR por el operador en ausencia de Tc es de 1012 a 1013 M-1 y disminuye en un factor de ~109 en presencia de Tc. TetR constituye el sistema de regulación transcripcional inducible más eficaz conocido en la actualidad (Orth et al., Nature Structural Biol., vol. 7 (2000) , págs. 215-219) .
Por otro lado, debe observarse que en función de las homologías de secuencia, se conocen seis clases (A, B, C, D, E y G) de TetR en las bacterias Gram negativas. Los genes que codifican para estos receptores son plasmídicos o se portan por transposones y todos son inducibles a concentraciones nanomolares en tetraciclina. Las proteínas presentan el 29% de aminoácidos idénticos entre las diferentes clases lo que supone una estructura 3D probablemente similar (Hillen et al., Annu. Rev. Microbiol., vol. 48 (1994) , págs. 345-332; Klock et al., J. Bac., vol. 161 (1985) , págs. 326-332) .
En cuanto a VarR, aún no se conocen los parámetros de afinidad en presencia y en ausencia de inductor pero es probable que sean del mismo orden de magnitud. Dado que la afinidad del antibiótico por su diana, el ribosoma 50S, es similar, un alto delta entre la represión y la inducción del transportador de membrana también es vital para la célula.
Otros represores menos vitales tales como LacI sólo muestran una pérdida de actividad de 105 en presencia del inductor (Matthews et al., Nature Structure Biol., vol. 7 (2000) , págs. 184-187) .
Una vez más, debe observarse que existen excepciones ya que AmpR fijado al operador puede activar la transcripción in vitro en ausencia de ligando o en presencia del ligando tripéptido anhidro-Mur-Nac. Por el contrario, en presencia de otro ligando de AmpR, el pentapéptido UDP-Mur-Nac, se inhibe la transcripción (Jacobs et al., Cell, vol. 88 (1997) , págs. 823-832) . AraC se fija en el ADN en presencia y en ausencia de ligando (L-arabinosa) pero la transcripción sólo se inicia en presencia de ligando tras una “formación de bucle” de ADN (Soisson et al., Science, vol. 276 (1997) ,... [Seguir leyendo]
Reivindicaciones:
1. Kit de detección y/o de cuantificación de un antibiótico (C) , perteneciente a la familia de las tetraciclinas o de las virginiamicinas, presente en una muestra de análisis, por medio de un receptor (A) , estando dicho kit destinado a una puesta en práctica in vitro, es decir sin expresarse en ningún sistema biológico o celular, caracterizado porque consiste en los siguientes reactivos:
-a modo de receptor (A) , un represor genético respectivamente de tetraciclina o de virginiamicina, que presenta la propiedad de presentar al menos un primer sitio de reconocimiento específico para el antibiótico (C) y un segundo sitio de reconocimiento específico para una secuencia nucleotídica operadora (B) correspondiente al represor, siendo el segundo sitio distinto del primer sitio, y que presenta la propiedad de que la fijación de dicha secuencia nucleotídica (B) en el receptor (A) en dicho segundo sitio de reconocimiento específico se modula negativamente mediante la fijación del antibiótico (C) en el receptor (A) en dicho primer sitio de reconocimiento específico, es decir que la fijación de dicha secuencia nucleotídica (B) únicamente tiene lugar en ausencia de fijación del antibiótico (C) en el receptor (A) ;
-dicha secuencia nucleotídica (B) , monocatenaria o bicatenaria.
2. Kit según la reivindicación 1, caracterizado porque dicho represor genético de tetraciclina, respectivamente de virginiamicina, es el represor natural TetR de una de las clases A a G conocidas en las bacterias Gram, o un mutante del mismo, respectivamente el represor natural VarR o un mutante del mismo.
3. Kit según la reivindicación 1 ó 2, caracterizado porque la secuencia nucleotídica operadora (B) es un fragmento bicatenario de secuencia específica de las regiones operadoras de los genes tetA y tetR para el reconocimiento de las tetraciclinas o varA y varR para el reconocimiento de las virginiamicinas.
4. Kit según la reivindicación 3, caracterizado porque dicho fragmento está situado en el extremo 3’ de un elemento constituido por una biotina y por una cadena monocatenaria poli-T.
5. Kit según cualquiera de las reivindicaciones 1 a 4, caracterizado porque la secuencia nucleotídica operadora (B) está inmovilizada, directa o indirectamente, sobre un soporte sólido, estando el receptor (A) eventualmente marcado por al menos una molécula (E, D) .
6. Kit según cualquiera de las reivindicaciones 1 a 4, caracterizado porque el receptor (A) está inmovilizado, directa
o indirectamente, sobre un soporte sólido, con ayuda de un anticuerpo específico y de una proteína A, estando la secuencia nucleotídica (B) opcionalmente marcada por al menos una molécula (E’, D’ ) .
7. Kit según la reivindicación 5, caracterizado porque el receptor (A) está marcado, directa o indirectamente, por medio de un anticuerpo (D) o una biotina (E) , pudiendo, dicho anticuerpo (D) , respectivamente dicha biotina (E) , estar, a su vez, marcado/marcada por medio de una proteína A, respectivamente por una molécula que fija la biotina, realizándose dicho marcaje del receptor (A) , del anticuerpo (D) o de la biotina (E) con ayuda de partículas coloreadas, preferentemente partículas de oro coloidales, o con ayuda de una enzima, preferentemente la peroxidasa.
8. Kit según la reivindicación 6, caracterizado porque la secuencia nucleotídica (B) está marcada, directa o indirectamente, por medio de un anticuerpo (D’) o una biotina (E’) , pudiendo, a su vez, dicho anticuerpo (D’) , respectivamente dicha biotina (E’) , estar marcado/marcada por medio de una proteína A, respectivamente por una molécula que fija la biotina, realizándose dicho marcaje de la secuencia nucleotídica (B) , del anticuerpo (D’) o de la biotina (E’) con ayuda de partículas coloreadas, preferentemente partículas de oro coloidales, o con ayuda de una enzima, preferentemente la peroxidasa.
9. Kit según la reivindicación 5 ó 6, caracterizado porque no se necesita ningún elemento marcado para una detección, pudiendo detectarse la simple presencia o la ausencia del complejo receptor (A) -fragmento nucleotídico
(B) mediante un procedimiento fisicoquímico, preferentemente un procedimiento de resonancia de plasmón superficial (SPR) o de espectrometría de masas.
10. Kit según la reivindicación 5, caracterizado porque el fragmento nucleotídico (B) , acoplado a una biotina (E) , está asociado a una proteína que fija la biotina, preferentemente la avidina, la estreptavidina, la neutravidina o un anticuerpo anti-biotina, para formar un complejo que se deposita sobre una membrana de nitrocelulosa, definiendo un primer punto de captura para el receptor (A) .
11. Kit según la reivindicación 10, caracterizado porque la membrana de nitrocelulosa comprende un segundo punto de captura que puede recuperar, total o parcialmente, el exceso de reactivos que no se fija al primer punto de captura, comprendiendo dicho segundo punto de captura preferentemente gammaglobulina, proteína A o anticuerpo anti-proteína A.
12. Kit según la reivindicación 11, caracterizado porque, tras la detección, comprende unos medios para cuantificar las señales obtenidas en dichos dos puntos de captura, preferentemente mediante interpretación visual, mediante medición óptica, e incluso preferentemente mediante reflectividad, absorbancia, transmisión, fluorescencia, cámara digital, quimioluminiscencia, mediante resonancia de plasmón superficial o mediante espectrometría de masas.
13. Kit según la reivindicación 1, caracterizado porque dichos reactivos se acondicionan en un matraz liofilizado y sobre un elemento de tipo tira inmunocromatográfica.
14. Kit según la reivindicación 13, caracterizado porque está desprovisto de matraz y porque dichos reactivos se asocian directamente en el interior de dicho elemento de tira sobre una membrana próxima a su extremo que puede entrar en contacto con un líquido de análisis.
15. Kit según la reivindicación 13 ó 14, caracterizado porque dicho elemento de tipo tira inmunocromatográfica está formado por un soporte lineal que comprende una membrana de nitrocelulosa acoplada a un papel absorbente que presenta dos puntos de captura, un primer punto de captura para recuperar total o parcialmente el complejo secuencia nucleotídica (B) -receptor (A) -ligando (C) y un segundo punto de captura para recuperar todo el exceso de reactivos que no se han fijado al primer punto de captura.
16. Procedimiento de detección y/o de cuantificación de un antibiótico (C) , perteneciente a la familia de las tetraciclinas o de las virginiamicinas, presente en una muestra biológica, caracterizado porque:
-se pone en contacto dicha muestra con los reactivos presentes en el kit según cualquiera de las reivindicaciones 1 a 15 y porque
-se detecta la presencia o se mide la concentración de antibiótico (C) presente en dicha muestra mediante inmunocromatografía, mediante determinación enzimática de dosis, preferentemente de tipo ELISA, mediante SPR o mediante espectrometría de masas.
17. Procedimiento según la reivindicación 16, caracterizado porque la muestra biológica se pone directamente en presencia de los reactivos sin tener que purificarse previamente.
18. Procedimiento según la reivindicación 16 ó 17, caracterizado porque la muestra biológica comprende carne, pescado, sangre, suero, líquido fisiológico, orina, lágrimas, saliva, leche, miel, agua, alimentos para ganado u otras preparaciones alimenticias o líquidos de cultivo y de fermentación, o incluso preparaciones o derivados de estas sustancias.
Patentes similares o relacionadas:
Método de determinación de la presencia y/o cantidad de moléculas diana, del 22 de Julio de 2020, de Canopy Biosciences, LLC: Método para el análisis de células individuales en una muestra de sangre mediante la determinación de la presencia y/o cantidad de una o más moléculas […]
Kit de reactivos utilizado para detectar gastrina-17 y método de preparación y aplicación para el kit de reactivos, del 15 de Julio de 2020, de Shenzhen New Industries Biomedical Engineering Co., Ltd: Un kit para detectar gastrina-17, caracterizado porque comprende un componente A y un componente B, en donde el componente A es un primer […]
Un sustrato para inmovilizar sustancias funcionales y un método para preparar las mismas, del 15 de Julio de 2020, de Temasek Polytechnic: Un sustrato sólido que tiene compuestos dispuestos sobre el mismo, en donde se inmoviliza una molécula funcional sobre los compuestos, teniendo cada compuesto una cadena que […]
Análisis biológico autónomo de alta densidad, del 1 de Julio de 2020, de BioFire Diagnostics, LLC: Recipiente para realizar reacciones de amplificación en un sistema cerrado que comprende una porción flexible que tiene una serie de blísteres […]
Chip de análisis y aparato de análisis de muestras, del 1 de Julio de 2020, de Takano Co., Ltd: Un chip de análisis que comprende: un sustrato conformado sustancialmente en forma de disco; un puerto de inyección formado […]
Dispositivo para la detección de analitos, del 1 de Julio de 2020, de TECHLAB, INC.: Un dispositivo para detectar por lo menos una sustancia de interés en una muestra líquida, comprendiendo el dispositivo: (a) una unidad que […]
Estructuras para controlar la interacción de luz con dispositivos microfluídicos, del 3 de Junio de 2020, de Opko Diagnostics, LLC: Un sistema de ensayo múltiplex que comprende: un artículo que soporta ensayos en fase sólida, comprendiendo dicho artículo un sustrato plano rígido y comprendiendo […]
Uso de un estándar para la detección de agregados de proteínas de una enfermedad por plegamiento incorrecto de proteínas, del 27 de Mayo de 2020, de FORSCHUNGSZENTRUM JULICH GMBH: Uso de un estándar, que comprende una nanopartícula inorgánica con un tamaño de 2 a 200 nm, en cuya superficie ya sea mediante i) ácido carboxílico-espaciador-maleimida […]