Métodos para alterar la reactividad de las paredes celulares vegetales.
Un método para la producción de oligosacáridos cargados positivamente en la pared celular vegetal,
particularmente la pared celular secundaria, comprendiendo dicho método
i. introducir o proporcionar un gen quimérico en la célula vegetal,
comprendiendo dicho gen quimérico:
1. un promotor expresable en plantas;
2. una región de ADN que codifica una proteína de nodulación C fusionada a una secuencia de anclaje señal heteróloga para fijar como objetivo las membranas del aparato de Golgi; y
3. una región de terminación de la transcripción y de poliadenilación.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2011/004929.
Solicitante: Bayer CropScience NV .
Nacionalidad solicitante: Bélgica.
Dirección: J.E. Mommaertslaan 14 1831 Diegem BELGICA.
Inventor/es: MEULEWAETER,FRANK, VANHOLME,BARTEL.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- A01H5/00 NECESIDADES CORRIENTES DE LA VIDA. › A01 AGRICULTURA; SILVICULTURA; CRIA; CAZA; CAPTURA; PESCA. › A01H NOVEDADES VEGETALES O PROCEDIMIENTOS PARA SU OBTENCION; REPRODUCCION DE PLANTAS POR TECNICAS DE CULTIVO DE TEJIDOS. › Angiospermas,es decir, plantas con flores, caracterizadas por sus partes vegetales; Angiospermas caracterizadas de forma distinta que por su taxonomía botánica.
- A01H5/10 A01H […] › A01H 5/00 Angiospermas,es decir, plantas con flores, caracterizadas por sus partes vegetales; Angiospermas caracterizadas de forma distinta que por su taxonomía botánica. › Semillas.
- C12N15/54 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Transferasas (2).
- C12N15/62 C12N 15/00 […] › Secuencias de ADN que codifican proteínas de fusión.
- C12N15/82 C12N 15/00 […] › para células vegetales.
- C12N5/10 C12N […] › C12N 5/00 Células no diferenciadas humanas, animales o vegetales, p. ej. líneas celulares; Tejidos; Su cultivo o conservación; Medios de cultivo para este fin (reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00). › Células modificadas por introducción de material genético extraño, p. ej. células transformadas por virus.
PDF original: ES-2548166_T3.pdf
Fragmento de la descripción:
Métodos para alterar la reactividad de las paredes celulares vegetales La presente invención se refiere a la modificación de la reactividad de las paredes celulares vegetales, incluyendo las paredes celulares de plantas secundarias, particularmente como se pueden encontrar en fibras naturales de plantas productoras de fibra. En particular, la presente invención se refiere a fibras de algodón con reactividad alterada. La reactividad modificada se podría aplicar en métodos para la tinción de la pared celular que contiene material derivado de plantas tal como fibras naturales, utilizando colorantes reactivos con las fibras, para mejorar, p. ej., la solidez del color, o para disminuir los volúmenes de aguas residuales utilizadas durante el proceso de tinción. La reactividad modificada se podría aplicar también para mejorar la reactividad de fibras naturales con reaccionantes tales como ignífugos, agua, aceite y repelentes de la suciedad, agentes antiarrugas, suavizantes, agentes antiestáticos, agentes blanqueadores fluorescentes, etc. La presente invención proporciona métodos para aumentar la eficiencia de la producción de oligómeros de Nacetilglucosamina en las paredes celulares vegetales, con la ventaja adicional de que las plantas producidas por métodos de la invención no muestran un retraso en el crecimiento de las raíces.
Antecedentes de la técnica Las fibras naturales, que incluyen celulosa que contiene fibras naturales de plantas tales como algodón y lino, han sido utilizadas por la humanidad durante más de 5000 años. Sin embargo, fibras que contienen celulosa natural, no poseen la versatilidad química de las fibras sintéticas, debido a la naturaleza inerte relativa de la celulosa que consiste en monómeros de glucosa β-1-4-enlazados.
Esta naturaleza relativamente inerte es, p. ej., evidente durante el proceso de tinción de fibras y tejidos de algodón. Se utilizan varios tipos de colorantes para el algodón de color tales como colorantes directos y, sobre todo, colorantes reactivos con las fibras, que son ambos moléculas aniónicas. El propio algodón desarrolla una carga aniónica en agua, por lo que sin un tratamiento especial, la absorción de colorante por la fibra o el tejido es bastante elaborada.
Los colorantes directos crean un enlace hidrógeno relativamente débil con el polímero de celulosa formando una unión semi-permanente. Los colorantes directos son más fáciles de utilizar y menos costosos que los tintes reactivos con fibras, pero no resisten bien el lavado. Colorantes reactivos con las fibras son moléculas que combinan cromóforos con un grupo reactivo que forma enlaces covalentes fuertes con la fibra a través de reacción con grupos hidroxilo. Los enlaces covalentes proporcionan una buena resistencia de la fibra teñida frente al lavado.
Durante el proceso de tinción, se necesitan grandes cantidades de electrolitos para proteger los colorantes aniónicos de las cargas de fibra aniónicas. Colorantes hidrolizados sin tratar (hasta 40%) necesitan ser separados mediante múltiples etapas de lavado, generando grandes volúmenes de aguas residuales, que también contienen los electrolitos antes mencionados.
La provisión de fibras de celulosa con una carga eléctrica positiva, p. ej., mediante la incorporación de compuestos químicos cargados positivamente, podría, por lo tanto, mejorar la capacidad de tinción de fibras de celulosa naturales, así como mejorar cualquier reacción química de la fibra de celulosa modificada con compuestos químicos cargados negativamente. También haría posible el uso de colorantes de carácter ácido.
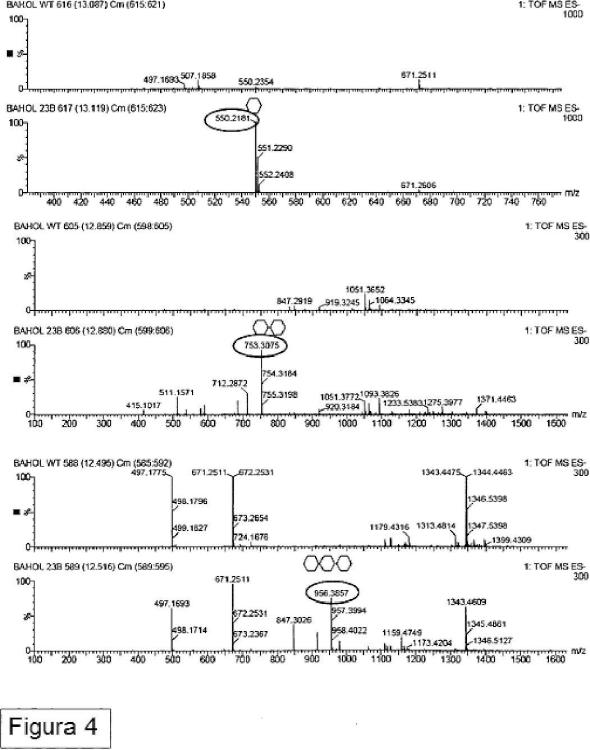
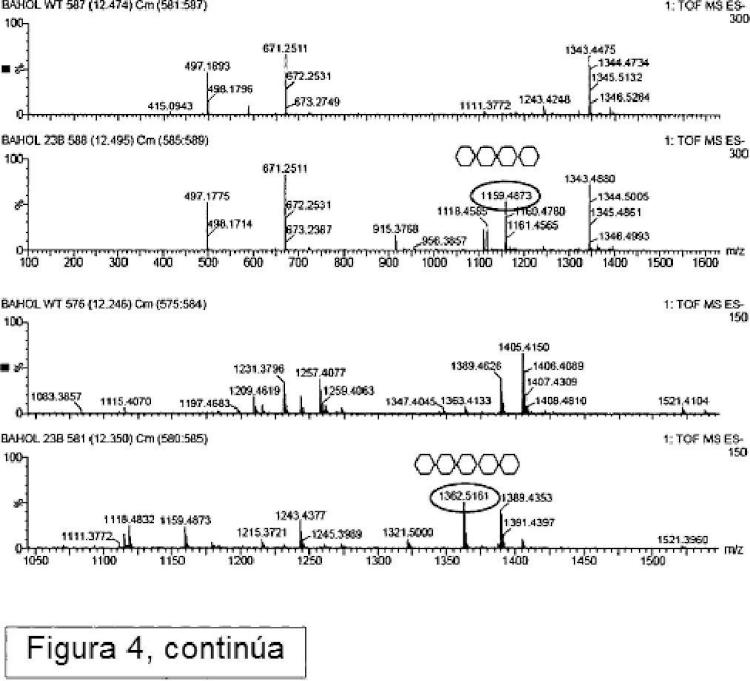
Varias publicaciones han descrito la incorporación en o el revestimiento de oligómeros de quitosano en fibras de celulosa para preparar mezclas de quitosano/celulosa, hilos o tejidos. El quitosano es un polímero de glucosamina cargado positivamente, que se puede obtener por desacetilación de quitina, p. ej., mediante tratamientos alcalinos. La quitina en sí es un polímero de N-acetilglucosamina β-1-4 enlazado (GlcNAc) .
La solicitud de patente US2003/0134120 describe el revestimiento de fibras naturales con quitosano.
Liu et al. (Carbohydrate Polymers 44 (2003) 233-238) describen un método para el revestimiento de fibras de algodón con quitosano, por oxidación del hilo de algodón con per y odato de potasio a 60°C en agua y subsiguiente tratamiento con una disolución de quitosano en ácido acético acuoso. Con el revestimiento de quitosano, la superficie de la fibra de algodón se volvió fisiológica y biológicamente activa. Puesto que la reactividad química del grupo amino es mayor que el grupo hidroxilo de monómeros de celulosa, la fibra tiene más potencial para una modificación química adicional. Además de ello, la superficie lisa de la fibra de algodón se volvió tosca, sugiriendo un mayor potencial para la absorción del fármaco y la liberación controlada del mismo.
Sobre la base de la función fisiológica de quitosano en inhibir, p. ej., dermatofitos, mucha prendas, tejidos y fibras funcionales emplean fibras de mezcla de celulosa quitosano, conjugados de fibras de celulosa-quitosano y tejidos revestidos con resinas que contienen quitosano.
El documento WO 00/09729 describe la expresión de genes de quitina sintasa y quitina desacetilasa en vegetales para alterar la pared celular para usos industriales y una resistencia mejorada a enfermedades. Específicamente usos citados son: proporcionar una única fuente vegetal de celulosa, quitina y quitosano, para aumentar la resistencia a la tracción y aumentar el chasquido quebradizo. Genes de quitina sintasa sugeridos específicamente se derivan de organismos fúngicos. No se proporcionan datos experimentales sobre la producción de quitina o quitosano en plantas, ni sobre la incorporación de los mismos en las paredes celulares vegetales.
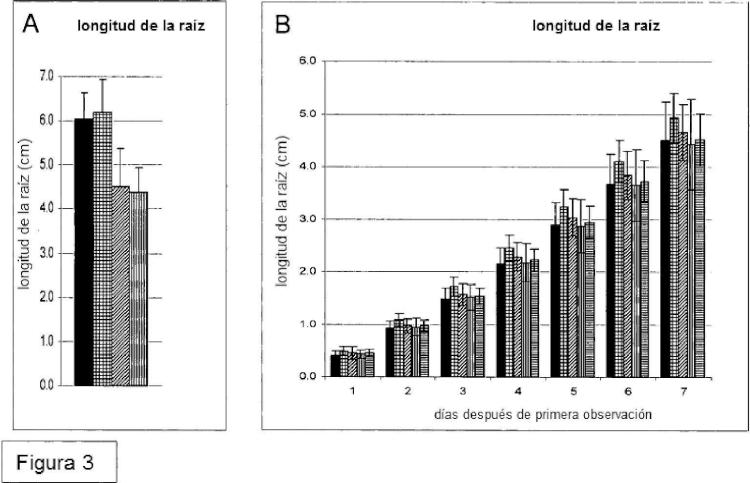
El documento WO2006/136351 demostró que la estrategia tal como se propone en el documento WO00/09729 no conduce a la incorporación funcional de quitina en la pared celular vegetal. En su lugar, el documento WO 2006/136351 describe que la quitina se produce eficazmente en la pared celular secundaria de fibras de algodón sólo cuando la N-acetilglucosamina transferasa se reubica en el aparato de Golgi. Para la quitina sintasa fúngica de Neurospora crassa la reubicación en el aparato de Golgi se consigue mediante la fusión operable de esta quitina sintasa fúngica con una secuencia de anclaje de señal heteróloga específica para el aparato de Golgi, y expresando el gen quimérico resultante en vegetales. Sin embargo, para el tipo NODC de N-acetil glucosamina transferasa, no se requiere la adición de una secuencia de anclaje señal para la localización de la proteína NodC en el aparato de Golgi ni para la incorporación de quito-oligosacáridos en la pared celular vegetal sin alimentación externa de GlcNAc. Aunque la quitina se podría producir de manera eficiente en las paredes celulares vegetales, también se observó que las plantas transgénicas que comprenden NODC tenían raíces más cortas en comparación con las plantas de tipo salvaje. Por lo tanto sigue existiendo una necesidad de métodos alternativos para producir paredes celulares vegetales tales como paredes celulares secundarias que comprenden polisacáridos cargados positivamente. En particular existe una necesidad de proporcionar métodos para producir plantas con oligosacáridos cargados positivamente en sus paredes celulares, pero sin retraso en el crecimiento de la raíz. Estos y otros problemas se resuelven como se describe en lo que sigue en las diferentes realizaciones, ejemplos y reivindicaciones.
Sumario de la invención En una realización, la invención proporciona un método para la producción de una célula vegetal, o una planta tal como una planta de algodón, que comprende oligosacáridos cargados positivamente en la pared celular, particularmente la pared celular secundaria de una célula vegetal, que comprende introducir un gen quimérico en la célula vegetal, en que el gen quimérico comprende un promotor expresable en vegetales operativamente enlazado a una región que codifica el ADN para una N-acetilglucosamina transferasa de tipo NODC fusionada a una secuencia de anclaje señal de Golgi; y una región de terminación de la transcripción y de poliadenilación. En otra realización, se proporciona un método para la producción de una planta tal como una planta de algodón que comprende oligosacáridos cargados positivamente en la célula utilizando métodos de acuerdo con la invención, caracterizado por que la longitud de la raíz de dicha planta es esencialmente la misma que la de una planta de tipo salvaje que no comprende el gen NODC.
La invención proporciona, además, un método para producir plantas que... [Seguir leyendo]
Reivindicaciones:
1. Un método para la producción de oligosacáridos cargados positivamente en la pared celular vegetal, particularmente la pared celular secundaria, comprendiendo dicho método i. introducir o proporcionar un gen quimérico en la célula vegetal, comprendiendo dicho gen quimérico:
1. un promotor expresable en plantas;
2. una región de ADN que codifica una proteína de nodulación C fusionada a una secuencia de anclaje señal heteróloga para fijar como objetivo las membranas del aparato de Golgi; y
3. una región de terminación de la transcripción y de poliadenilación.
2. Un método de acuerdo con la reivindicación 1, que comprende, además, la etapa de regeneración de dicha célula vegetal en una planta.
3. Un método de acuerdo con la reivindicación 2, caracterizado por que la longitud de la raíz de dicha planta es esencialmente la misma que la de una planta de tipo salvaje que comprende la proteína de nodulación C.
4. Un método de acuerdo con una cualquiera de las reivindicaciones 1 a 3, en el que dichos oligosacáridos cargados positivamente consisten en N-acetilglucaminasas β1-4-enlazadas con un grado de polimerización de 2 a 10.
5. Un método de acuerdo con una cualquiera de las reivindicaciones 1 a 4, en el que dicha secuencia de anclaje señal se selecciona se selecciona de la secuencia de anclaje señal de una sialil transferasa de rata, la secuencia de anclaje señal de una galactosiltransferasa humana, la secuencia de anclaje señal del homólogo de Arabidopsis del receptor de levadura HDEL (AtERD2) , la secuencia de anclaje señal de la α-2, 6-sialiltransferasa, la secuencia de anclaje señal de β1, 2-xilosiltransferasa de Arabidopsis thaliana, la secuencia de anclaje señal de Nmetilglucosaminil-transferasa I del tabaco o la secuencia de aminoácidos YYHDL o LKLEI.
6. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 5, en el que dicha región de ADN codifica una proteína de nodulación C obtenible de una especie de Rhizobium, una especie de Azorhizobium, una especie de Bradyrhizobium, una especie de Mesorhizobium, una especie de Ralstonia, una especie de Streptomyces, una especie de Burkholderia, una especie de Cupriavidus, una especie de Sinorhizobium, una especie de Desulfobacterium, una especie de Dokdonia, una especie de Methylobacterium, una especie de Phyllobacterium o una especie de Psychroflexus.
7. El método de la reivindicación 6, en el que dicha proteína de nodulación C comprende la secuencia de aminoácidos SEQ ID NO 1, SEQ ID NO 2. SEQ ID NO 3, SEQ ID NO 4, SEQ ID NO 5, SEQ ID NO 6, SEQ ID NO 7, SEQ ID NO 8 SEQ ID NO 9, 1615305C, 1615305D, 1615305E, AAA26226, AAA63602, AAB16897, AAB24745, AAB34509, AAB47353, AAB51164, AAB71694, AAB91695, AAB95329, AAC80567, AAD11313 , AAD11315, AAD11317, AAD11319, AAD11321, AAD11323, AAD11325, AAD11327, AAD11329, AAD11331, AAD11333, AAD11335, AAD11337, AAD11339, AAD11341, AAD11343, AAD11345, AAD11347, AAD11349, AAD11351, AAD11353, AAD11355, AAD11357, AAD11359, AAD11361, AAD11363 , AAD11365, AAD11367, AAD11369, AAD11371, AAD11373, AAD11375, AAD11377, AAD11379, AAD11381, AAD11383, AAD11385, AAD11387, AAD11389, AAD11391, AAD11393, AAD11395, AAD11397, AAD11399, AAD11401, AAD11403, AAD11405, AAG60998, AAK00157, AAK39956, AAK39957 , AAK39958, AAK39959, AAK39960, AAK39961, AAK39962, AAK39963, AAK39964, AAK39965, AAK39966, AAK39967, AAK50872, AAK65131, AAL88670, AAN62903, AAS91748, AAU11338, AAU11339, AAU11340, AAU11341, AAU11342, AAU11343, AAU11344, AAU11345, AAU11346, AAU11347 , AAU11348, AAU11349, AAU11350, AAU11351, AAU11352, AAU11353, AAU11354, AAU11355, AAU11356, AAU11357, AAU11358, AAU11359, AAU11360, AAU11361, AAU11362, AAU11363, AAU11364, AAU11365, AAX30049, AAX30050, AAY44091, AAY44092, AAY44093, AAY89044, AAZ81541 , ABC40958, ABC67303, ABD39006, ABD39007, ABD39008, ABD39009, ABD39010, ABD3901 1, ABD39012, ABD39013, ABD39014, ABD39015, ABD39016, ABD39017, ABD39018, ABD39019, ABD39020, ABD39021, ABD39022, ABD39023, ABD39024, ABD39025, ABD39026, ABD39027, ABD39028, ABD39029, ABD39030, ABD39031, ABD39032, ABD39033, ABD39034, ABD39035, ABD39036, ABD39037, ABD39038, ABD67413, ABD67416, ABD67419, ABD67422, ABD67425, ABD67428, ABD67431, ABD67434, ABD73319, ABD73320, ABD73321, ABD73322, ABD73323, ABD73324, ABD73325, ABD73326, ABD73327, ABD73328, ABD73329, ABD73330, ABD94161, ABD94162, ABD94163, ABD94164, ABD94165, ABF93198, ABF93199, ABF93200, ABF93201, ABF93202, ABM69186, ABM69187, ABM69188, ABM69189, ABM69190, ABN09217, ABN09218, ABN09219, ABN11 177, ABN1 1 178, ABN11179, ABP93834, ABS85176, ABS85177, ABS85178, ABS85179, ABS85180, ABS85181, ABS85182, ABU69044, ABU69045, ABU69046, ABU69047, ABU69048, ABU69049, ABU69050, ABU69051, ABU69052, ABU69053, ABU69054, ABU69055, ABU69056, ABU69057, ABU69058, ABU69059, ABU69060, ABU69061, ABU89879, ABV25689, ABV25690, ABV25691, ABV25692, ABV25693,
ABV25694, ABW96196, ABW96197, ABW96198, ABW96199, ABW96200, ABW96201, ABW96202, ABW96203, ABW96204, ABW96205, ABW96206, ABW96207, ABW96208, ABW96209, ABW96210, ABW96211, ABY59633, ABY59634, ABY59635, ABY59636, ABY59637, ACA80309, ACA80310, ACA80311, ACA80312, ACA80313, ACC77565, ACD39337, ACD39338, ACD39339, ACD39340, ACD39341, ACD39342, ACD39343, ACD39344, ACD39345, ACD39346, ACD39347, ACD62595, ACD63093, ACD63094, ACD63095, ACD63096, ACD63097, ACD63098, ACD63099, ACD63100, ACD63101, ACD63102, ACD63103, ACD63104, ACF19762, ACF 19763, 19764 ACF, ACF 19765, 19766 ACF, ACF 19767, 19768 ACF, ACF 19769, ACF19770, ACH91221, ACH91222, ACH91223, ACH91224, ACH91225, ACH91226, ACH91227, ACH91228, ACH91229, ACH91230, ACH91231, ACH91232, ACH91233, ACH91242, ACH91243, ACH91244, ACH91245, ACH91246, ACH91247, ACH91248, ACH91249, ACI47333, ACI47334, ACI47335, ACI47336, ACI47337, ACI47338, ACI47339, ACI47340, ACI47341, ACI47342, ACI47343, ACI47344, ACI47345, ACL12058, ACL12059, ACL50517, ACL50518, ACL50519, ACL50520, ACL50521, ACL50522, ACL50523, ACM69382, ACM79634, ACM79635, ACM79636, ACM79637, ACM79638, ACM79639, ACM79640, ACM79641, ACM79642, ACM79643, ACM79644, ACM79645, ACM79646, ACN17701, ACN69201, ACN69202, ACN69203, ACN69204, ACN69205, ACN69206, ACN69207, ACN69208, ACN69209, ACN69210, ACN69211, ACN69212, ACN69213, AC058664, AC058665, AC058666, AC058667, AC058668, AC058669, ACO58670, AC058671, AC058672, AC058673, AC058674, AC058675, ACP40990, ACS35430, ACS35434, ACT34091, ACT34094, ACT34097, ACT34100, ACT34101, ACT34104, ACT34107, ACT34110, ACT34113, ACT34116, ACT34119, ACT34122, ACT34125, ACT34128, ACT34131, ACT34134, ACT34137, ACT34140, ACT34143, ACV52950, ACV52951, ACV52952, ACV52953, ACV52954, ACV52955, ACX47326, ACX47327, ACX47328, ACX47329, ACX47330, ACX47331, ACX47332, ACX47333, ACX47334, ACY02884, ACY78518, ACZ52692, ACZ52693, ACZ52694, ACZ52695, ACZ52696, ACZ52697, ADD20957, ADD20958, ADD20959, ADD20960, ADD20961, ADD20962, ADD20963, ADD20964, ADD20965, ADD20966, ADD20967, ADD20968, ADD20969, ADD20970, ADD20971, ADG63645, ADG63646, ADG63647, ADG63648, ADG63649, ADG63650, ADG63651, ADG63652, ADJ18191, ADJ18192, BAA06082, BAA06083, BAA06084, BAA06085, BAA06086, BAA06087, BAA06088, BAA06089, BAA06090, BAA24092, BAB52500, C26813, CAA25810, CAA25811, CAA25814, CAA26310, CAA26311, CAA51773, CAA51774, CAA608779, CAA67139, CAB56055, CAC42489, CAD29949, CAD29950, CAD29951, CAD29952, CAD29953, CAD29954, CAD29955, CAD29956, CAD29957, CAD31533, CAD43933, CAD90257, CAD90583, CAD90584, CAD90585, CAD90586, CAD90587, CAD90588, CAH04369, CAN84684, CAP64017, EAQ38847, EAS72439, NP_106714, NP_435719, NP_443883, P04340, P04341, P04677, P04678, P04679, P06234, P06235, P17862, P24151, P26024, P50357, P53417, P72334, Q07755, Q53513, YP 001.796.208, YP 002605865, ZP_01050448 o ZP_01252.570.
8. El método de una cualquiera de las reivindicaciones 1 a 7, en el que dicha NODC fusionada a la secuencia anclaje de señal de Golgi comprende la secuencia de aminoácidos de SEQ ID NO 11.
9. El método de una cualquiera de las reivindicaciones 1 a 8, en el que dicho promotor expresable en plantas es un promotor específico para fibras.
10. El método de una cualquiera de las reivindicaciones 1 a 9, en el que dicho promotor expresable en plantas es un promotor específico para fibras seleccionado del promotor específico para fibras de un gen beta-tubulina del algodón, un promotor específico para fibras de un gen actina del algodón, un promotor específico para fibras de un gen de proteína de transferencia de lípidos del algodón, un promotor de la envuelta de semillas y proteasa específica para fibras del algodón, un promotor del gen R2R3 MYB específico para fibras del algodón, un promotor de un gen expansina del algodón o un promotor de un gen quitinasa en algodón.
11. El método de una cualquiera de las reivindicaciones 1 a 10, en el que dicho se selecciona de algodón, cáñamo o lino.
12. El método de la reivindicación 11, en el que la pared celular vegetal en dicha planta de algodón comprende fibras.
13. El método de una cualquiera de las reivindicaciones 1 a 12, que comprende, además, la etapa de desacetilar dichos oligosacáridos que consisten en monómeros de N-acetilglucosamina tratando la pared celular de dicha planta con una disolución alcalina durante un tiempo suficiente para desacetilar dicho oligosacárido que consiste en monómeros de N-acetilglucosamina, o a través de la acción enzimática de quitina desacetilasas.
14. El método de una cualquiera de las reivindicaciones 2 a 13, que comprende, además, la etapa de aislar la pared celular vegetal o fibras de dicha planta.
15. El método de una cualquiera de las reivindicaciones 2 a 14, en el que la pared celular de dicha planta es una pared celular vegetal secundaria.
16. Un gen quimérico según se describe en las reivindicaciones 1 a 10.
17. Una célula vegetal que comprende el gen quimérico de la reivindicación 16.
18. Una planta que consiste esencialmente en las células vegetales de la reivindicación 17.
19. La planta de la reivindicación 18, caracterizada por que la longitud de la raíz de dicha planta es esencialmente la misma que la de plantas de tipo salvaje que no comprenden NODC.
20. La planta de la reivindicación 18 ó 19, que es algodón.
21. Uso del gen quimérico de la reivindicación 16 para incrementar la cantidad de oligosacáridos cargados positivamente en la pared celular de una célula vegetal o para incrementar la reactividad de las paredes celulares vegetales para modificaciones químicas de estas paredes celulares vegetales.
22. Uso del gen quimérico de la reivindicación 16, para producir plantas con oligosacáridos cargados positivamente
en la pared celular, caracterizado por que la longitud de la raíz de dicha planta es esencialmente la misma que la de una planta isogénica que no comprende la proteína de nodulación C.
23. Uso del gen quimérico de la reivindicación 16, para producir plantas de algodón con oligosacáridos cargados positivamente en las fibras.
24. Uso del gen quimérico de la reivindicación 16. para producir fibras de algodón con reactividad mejorada tal como 15 una capacidad de tinción mejorada.
25. Uso del gen quimérico de la reivindicación 16, para producir hilos y tejidos con reactividad mejorada tal como una capacidad de tinción mejorada.
79 80 81 82 83 84
Patentes similares o relacionadas:
Cadena ligera de enteroquinasa modificada, del 22 de Julio de 2020, de NOVO NORDISK A/S: Un análogo de la cadena ligera de la enteroquinasa bovina que comprende una secuencia de aminoácidos establecida en la SEQ ID NO: 1, en donde dicho análogo comprende […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos y composiciones para ingeniería genómica, del 3 de Junio de 2020, de Sangamo Therapeutics, Inc: Una pareja de nucleasas de dedo de zinc (ZFN) que comprende una ZFN izquierda y una ZFN derecha, comprendiendo cada ZFN un dominio de escisión […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Antígenos de coagulasa estafilocócica y métodos para su uso, del 13 de Mayo de 2020, de UNIVERSITY OF CHICAGO: Una composición inmunógena que comprende al menos dos dominios 1-2 de coagulasa estafilocócica diferentes, en donde cada uno de los al menos dos dominios […]
Reconocimiento de unión a diana celular mediante un agente bioactivo usando transferencia de energía de resonancia de bioluminiscencia intracelular, del 6 de Mayo de 2020, de PROMEGA CORPORATION: Un sistema de ensayo que comprende: (a) una biblioteca de agentes bioactivos, cada uno de los cuales está fijado a un fluoróforo; (b) una diana celular fusionada a […]
Etiqueta de epítopo y método de detección, captura y/o purificación de polipéptidos etiquetados, del 15 de Abril de 2020, de ChromoTek GmbH: Péptido epítopo aislado que tiene de 12 a 25 aminoácidos, en donde la secuencia de aminoácidos comprende una secuencia según se define en SEQ ID NO: 32 (X1X2RX4X5AX7SX9WX11X12), […]
Utilización diagnóstica de un polipéptido de fusión que comprende una proteína vírica y un enzima MGMT, del 15 de Abril de 2020, de INSTITUT PASTEUR: Utilización in vitro de un polipéptido de fusión que comprende una proteína vírica y i) el enzima 6-metilguanina-ADN-metiltransferasa (MGMT, EC 2.1.1.63) o un homólogo […]