Método para dectectar inserciones de espaciadores en estructuras CRISPR.
La presente invención se refiere a un método para detectar inserciones de espaciadores mediante selección independiente de la interferencia a partir de estructuras artificiales basadas en repeticiones palindrómicas cortas agrupadas regularmente espaciadas (CRISPR) que comprende la inserción de al menos una unidad CRISPR-espaciador,
en la estructura artificial dentro de la secuencia que codifica un gen testigo, que restaura la pauta de lectura de traducción, donde la expresión del gen testigo es indicativo de la inserción del espaciador. También se refiera a la estructura artificial utilizada.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201300203.
Solicitante: UNIVERSIDAD DE ALICANTE.
Nacionalidad solicitante: España.
Inventor/es: GARCIA MARTINEZ,JESUS, DÍEZ VILLASEÑOR,César, MARCO GUZMÁN,Noemí, MARTÍNEZ MÓJICA,Francisco J, ALMENDROS ROMERO,Cristóbal.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/74 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Vectores o sistemas de expresión especialmente adaptados a huéspedes procariotas distintos a E. coli, p. ej. Lactobacillus, Micromonospora.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2522765_A1.pdf
Fragmento de la descripción:
MÉTODO PARA DETECTAR INSERCIONES DE ESPACIADORES EN ESTRUCTURAS CRISPR
Campo de la invención
La presente invención se encuadra en general en el campo de la genética y en 5 particular se refiere a un método para detectar inserciones de espaciadores mediante selección independiente de la interferencia a partir de estructuras artificiales basadas en CRISPR.
Estado de la técnica
Los sistemas CRISPR-Cas se han identificado en la mayoría de las arqueas y 10 aproximadamente la mitad de los genomas de bacterias. Cada sistema está constituido por una o varias agrupaciones de repeticiones de ADN denominadas CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) y un conjunto de genes que codifican las proteínas Cas (CRISPR associated) funcionalmente relacionados con la actividad de las CRISPR. Dichas repeticiones se encuentran 15 regularmente espaciadas dentro de cada agrupación por secuencias no reiteradas denominadas espaciadores, al menos algunas de las cuales derivan de fragmentos localizados en elementos genéticos móviles, tales como plásmidos y virus. Adyacente a cada agrupación de repeticiones-espaciadores hay una secuencia denominada líder donde se localiza el promotor responsable de la transcripción de dicha agrupación en 20 un RNA primario (pre-crRNA) que abarca la totalidad de la misma.
La identidad de los genes cas asociados a una agrupación CRISPR puede variar de manera considerable entre sistemas, en base a la cual los CRISPR-Cas se clasifican en 3 tipos principales, cada uno de los cuales incluye varios subtipos. Tan solo casi y cas2 están presentes en todos los subtipos identificados hasta la fecha.
Los sistemas CRISPR-Cas producen la degradación (interferencia) de ADN exógeno conteniendo secuencias complementarias a los espaciadores. Un sistema CRISPR- Cas activo puede insertar nuevos espaciadores (proceso conocido como adquisición y catalizado por Casi y Cas2) entre el líder y la repetición contigua. La inserción conlleva la duplicación de la repetición adyacente al sitio de inserción. La adquisición 30 de un nuevo espaciador proporciona inmunidad frente a moléculas conteniendo la secuencia de la cual deriva. Para ello, en un primer paso del proceso de interferencia los pre-crRNAs son procesados dando lugar a moléculas crRNA conteniendo un único
espaciador. Cada uno de estos crRNAs aparea con su secuencia complementaria en una molécula de ADN reclutando a continuación proteínas Cas específicas que producen su degradación. Además de una función como sistema inmune, se ha propuesto una implicación de los CRISPR-Cas en regulación génica y reparación de ácidos nucleicos.
La inserción de nuevas unidades CRISPR-espaciador es un evento poco frecuente y cuya detección requiere realizar un escrutinio al azar de las agrupaciones CRISPR de un gran número de clones (por ejemplo mediante amplificación por PCR). Tan solo cuando dicha adquisición produzca un nuevo patrón de interferencia se podrían seleccionar las células adaptadas, requiriendo para ello condiciones experimentales que permitan dicha interferencia y excluyendo la detección de inserciones silenciosas.
La disponibilidad de una herramienta para detectar inserciones de espaciadores mediante una selección independiente de la interferencia resulta por tanto ventajosa respecto a sistemas no seleccionables.
Descripción de la invención
Así pues la presente invención en un primer aspecto se refiere a un método para detectar inserciones de espaciadores, de ahora en adelante método de la presente invención, mediante selección independiente de la interferencia a partir de estructuras artificiales basadas en repeticiones palindrómicas cortas agrupadas regularmente espaciadas (CRISPR) que comprende la inserción de al menos una unidad CRISPR- espaciador, en la estructura artificial dentro de la secuencia que codifica un gen testigo, que restaura la pauta de lectura de traducción, donde la expresión del gen testigo es indicativo de la inserción del espaciador.
En una realización más en particular la unidad CRISPR-espaciador insertada comprende un número de nucleótidos no múltiplo de 3.
En una realización más en particular, el espaciador de la unidad CRISPR-espaciador del método de la presente invención comprende un codón de terminación. Más en particular el codón de terminación es críptico.
En una realización más en particular, la estructura artificial del método de la presente invención comprende la secuencia identificada como SEQ ID NO: 103, en una realización más en particular, la estructura artificial del método de la presente
invención se corresponde con la fusión traduccional de un cassette CRISPR- espaciador-CRISPR-líder y la secuencia codificante del gen de la cloranfenicol acetil-transferasa (caí), donde los nucleótidos del 96-124 y 156-165 se corresponden con las unidades CRISPR y la región líder (nucleótidos 165-234) del cassette insertado 5 de la región codificante de la proteína CAT (nucleótidos 275-953) que se encuentra fuera de fase respecto al inicio de la traducción.
En una realización más en particular de la presente invención, el espaciador es seleccionado de entre las secuencias identificadas como SEQ ID NO: 1- SEQ ID NO: 89.
En un segundo aspecto, la presente invención se refiere a una estructura artificial, de ahora en adelante, estructura artificial de la presente invención) identificada caracterizada porque comprende la secuencia identificada como SEQ ID NO: 103.
En un tercer aspecto, la presente invención se refiere al uso de la estructura artificial de la presente invención para la detección de inserciones de espaciadores.
Descripción de las figuras
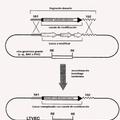
Figura 1: Representación esquemática de la integración en los cassettes testigo clonados en los plásmidos pCSIR y la estrategia usada para detección específica de integración. A. Cassette de integración en pCSIR-A o pCSIR-T consta de un líder y dos duplicones CRISPR (CR) interespaciados por un espaciador (Sp-1) fusionado aguas arriba a un fragmento del gen of lacZ-a (lacZa) y, aguas abajo la secuencia completa del gen cat fuera de fase (cat +2). Se indican el punto de unión al ribosoma (rbs) y el codón de inicio de la traducción lacZ-a (punta de flecha blanca). La traducción de los transcritos generados desde el promotor tac (P,ac, representado con una flecha) termina en el líder (punta de flecha negra). Aguas abajo del gen cat se localiza un promotor de T7 (P-17), que inicia la transcripción en sentido opuesto (la flecha apunta en el sentido de la transcripción). Se muestran las secuencias de los fragmentos en la región líder-caí de pCSIR-T y pCSIR-A. Los nucleótidos idénticos a la variante de líder T o A correspondiente están sombreados, los codones de terminación y la caja -10 subrayados y etiquetados (el punto de inicio se indica con una flecha que apunta en la dirección de transcripción). Para evitar codones de terminación, se reemplazaron las timinas en las posiciones 42 y 70 del líder de pCSIR-T por citosina y adenina respectivamente (ver el cebador TLR en la Tabla 2). La traducción del
transcrito generado de Ptac acaba en el líder en caso de deleclón (caí seguiría fuera de fase, caf+1) (B), en el espaciador duplicado (Sp-1) en caso de duplicación CRISPR-espaciador (C), o en el codón de terminación de la secuencia codificante de cat en el caso de la inserción (D) de un espaciador nuevo (Sp-2).
Figura 2: Distribución, en pCas1-2[K], de las secuencias correspondientes a espaciadores muestreados en los ensayos de adquisición. Los espaciadores se muestran como flechas negras apuntando al líder tal y como se insertaron en el cassette de integración. Se muestra el origen de replicación (CDF ori), los genes codificando el represor del operón lac (Lacl), la proteína de resistencia a estreptomicina (SmR), y el promotor T7-lac (Pj7) que promueve la transcripción de la variante K de los genes cas1-2 (casi-K y cas2-K).
Figura 3: WebLogos generados por el alineamiento de las regiones de protoespaciadores de CRISPR2 de E. coli. Las secuencias conteniendo protoespaciadores (posiciones -1 a -33) y los dos nucleótidos adyacentes de la región PAM (posiciones 0 y 1) estaban orientados del mismo modo que los espaciadores correspondientes en la agrupación CRISPR (el extremo 3 hacia el líder). Los logos se obtuvieron a partir de secuencias no CRISPR con una identidad mayor al 90% a espaciadores de genomas de E. coli, asociados con el líder-T (A) o el líder-A (B), así como protoespaciadores correspondientes a espaciadores encontrados en los experimentos de adquisición realizados en cepas alojando los genes Cas-EK (C) o Cas-EO (D).
Figura 4: Mecanismo hipotético de... [Seguir leyendo]
Reivindicaciones:
1. Método para detectar inserciones de espaciadores mediante selección independiente de la interferencia a partir de estructuras artificiales basadas en repeticiones palindrómicas cortas agrupadas regularmente espaciadas (CRISPR) que
comprende la inserción de al menos una unidad CRISPR-espaciador, en la estructura artificial dentro de la secuencia que codifica un gen testigo, que restaura la pauta de lectura de traducción, donde la expresión del gen testigo es indicativo de la inserción del espaciador.
2. Método según la reivindicación 1, caracterizado por que la unidad CRISPR- 10 espaciador insertada comprende un número de nucleótidos no múltiplo de 3.
3. Método según cualquiera de las reivindicaciones anteriores, caracterizado por que el espaciador comprende un codón de terminación críptico.
4. Método según cualquiera de las reivindicaciones anteriores, caracterizado por que la estructura artificial comprende la secuencia identificada como SEQ ID NO: 103.
5. Método según cualquiera de las reivindicaciones anteriores, caracterizado por que
el espaciador es seleccionado de entre las secuencias identificadas como SEQ ID NO: 1-SEQ ID NO: 89.
6. Estructura artificial identificada caracterizada por que comprende la secuencia identificada como SEQ ID NO: 103.
7. Uso de la estructura artificial según la reivindicación 6 para la detección de
inserciones de espaciadores.
Patentes similares o relacionadas:
Sistema de expresión bacteriano cistrónico doble, del 25 de Marzo de 2020, de LUPIN LIMITED: Un procedimiento para la producción de un anticuerpo o un fragmento del mismo que comprende las etapas de: (i) transformar una célula hospedadora bacteriana […]
Partículas de transducción no replicativas y sistemas indicadores basados en partículas de transducción, del 15 de Enero de 2020, de Geneweave Biosciences Inc: Un sistema de empaquetamiento de células bacterianas para empaquetar una molécula de ácido nucleico indicadora en una partícula de transducción no replicativa (NRTP) para su introducción […]
Método de construcción de biblioteca de péptidos y vectores relacionados, del 1 de Enero de 2020, de Hunan Zonsen Peplib Biotech Co., Ltd: Método integrado de construcción de una biblioteca de péptidos completa que contiene todos los péptidos posibles para el tamaño específico, comprendiendo el método: (i) […]
 Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Transporte de proteínas basado en bacterias, del 14 de Agosto de 2019, de Universität Basel: Un vector que comprende, en la dirección 5' a 3': un promotor; una primera secuencia de ADN que codifica una señal de transporte procedente […]
Sistema de expresión y secreción, del 26 de Junio de 2019, de F. HOFFMANN-LA ROCHE AG: Una molécula de ácido nucleico que codifica un primer polipéptido y un segundo polipéptido, enlazada de forma funcional a una secuencia señal que codifica […]
 Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Síntesis microbiana de aldehídos y alcoholes correspondientes, del 17 de Junio de 2019, de Easel Biotechnologies, LLC: Un método para producir un alcohol, que comprende: cultivar una pluralidad de células microbianas en un medio de fermentación, en donde las […]