Translocación de proteínas en células de plantas.
Método para llevar a cabo una modificación en una célula no humana,
en el que un sistema de transferenciabacteriano se pone en contacto con la célula a ser modificada, dicho sistema de transferencia tiene un sistema detransporte de proteínas que comprende un poro, el canal de secreción de tipo IV, que incluye un complejo VirB yproteína VirD4, en el que el sistema de transferencia comprende una proteína de fusión o es capaz de crear unaproteína de fusión que se introduce en la célula utilizando el sistema de transporte de proteínas, en el que seintroduce una proteína de fusión BA en la célula a ser modificada, cuya proteína de fusión BA comprende.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/NL2001/000388.
Solicitante: STICHTING VOOR DE TECHNISCHE WETENSCHAPPEN.
Nacionalidad solicitante: Países Bajos.
Dirección: Van Vollenhovenlaan 661 3527 JP Utrecht PAISES BAJOS.
Inventor/es: HOOYKAAS, PAUL, JAN, JACOB, VERGUNST,ANNETTE CAROLINE, SCHRAMMEIJER,BARBARA.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07K14/195 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 14/00 Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados. › de origen bacteriano.
- C12N15/62 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Secuencias de ADN que codifican proteínas de fusión.
- C12N15/82 C12N 15/00 […] › para células vegetales.
- C12N9/00 C12N […] › Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas.
PDF original: ES-2427928_T3.pdf
Fragmento de la descripción:
Translocación de proteínas en células de plantas La presente invención se refiere a un método para llevar a cabo una modificación en una célula no humana, en el que un sistema de transferencia se pone en contacto con la célula a ser modificada, dicho sistema de transferencia comprende una membrana con un sistema de transporte de proteínas en ella que comprende un poro que comprende un complejo VirB y la proteína VirD4, en el que el sistema de transferencia comprende una proteína de fusión o es capaz de crear una proteína de fusión que se introduce en la célula por medio del sistema de transporte de proteínas.
Dicho método se sugirió por Zhou, XR y otros (Journal of Bacteriology, 181 (14) , p. 4342-4352 (1999) ) . Esta publicación da a conocer que la proteína VirE2, una proteína de unión a ADN de hebra única de Agrobacterium tumefaciens, se puede transferir a una célula huésped por un Agrobacterium tumorigénico. Zhou y otros dan a conocer que eran sólo fueron capaces de introducir una secuencia de aminoácidos heteróloga en el aminoácido 39 (a partir del N-terminal, como es habitual en la técnica) que fue transferida a la célula huésped. Se llegó a la conclusión de que no es viable en la práctica basar un sistema de administración de proteínas en fusiones en uno de los dos extremos de VirE2. Como mínimo, las proteínas pequeñas son toleradas como una inserción en el aminoácido 39.
El objeto de la presente invención es dar a conocer un método, que es simple de realizar y, si se desea, se lleva a cabo sin actividad nativa de VirE2 en la célula a ser modificada.
En consecuencia, el método según la presente invención, es el método que se describe en la reivindicación 1.
Sorprendentemente, se ha descubierto que, siempre que se cumplan las condiciones mencionadas anteriormente, una proteína puede ser introducida desde el exterior en una célula a ser modificada, cuya proteína se forma mediante el acoplamiento del polipéptido (segunda parte B) a un aminoácido N-terminal (interno) de VirF, VirD2, VirE2, VirE3 o MobA (primera parte A) , como resultado de lo cual la actividad de la segunda parte de B introducida se puede expresar en la célula a ser modificada, como resultado de lo cual se modifica la célula. Esta modificación puede ser temporal (reversible) o permanente (irreversible) . Las secuencias de aminoácidos de VirF, VirD2, VirE2, VirE3 y MobA están representadas por las respectivas secuencias número 1-5. Una cepa que comprende un plásmido (LBA8250, una cepa de Agrobacterium tumefaciens, que contiene el plásmido pTi15955. PTi15955 se describe por Sciaky y otros, Plasmid 1, p. 238-253 (1978) ) que codifica todas las proteínas Vir, está en la colección Phabagen del Centraalbureau voor Schimmelcultures (CBS) , Baarn, Países Bajos con el número de acceso PC2692. La proteína MobA es conocida a partir del plásmido IncQ RSF1010, que está presente en la cepa de E. coli K12 C600 con el número de acceso PC-V3110 de la colección NCCB del CBS, y se describió por Scholz, P. y otros en Gene 75, p. 271-288 (1989) .
En la presente invención, un aminoácido correspondiente es un aminoácido según la tabla siguiente:
A, G;
S, T;
D, E;
N, Q;
R, K;
I, L, M, V; y
F, Y, W.
No es necesario mencionar que la primera parte A puede comprender más aminoácidos adyacentes o no adyacentes, idénticos o correspondientes a las secuencias definidas anteriormente.
La primera parte A, si se desea, comprende más aminoácidos desde el aminoácido 41 (a partir del C-terminal) de VirF, o VirE2 de VirE3 o VirD2 o MobA. Esto bajo la condición mencionada para VirE2.
En la presente invención, un vector se refiere a cualquier secuencia de ADN o ARN que, directa o indirectamente, conduce a la formación de la proteína de fusión en el sistema de transferencia.
El sistema de transferencia puede ser una célula, tal como de forma muy adecuada una célula bacteriana, tal como, en particular, Agrobacterium tumefaciens, pero también puede ser un sistema artificial, tales como una minicélula o un sistema artificial de vesículas.
La proteína de fusión a ser transferida o bien se puede formar en el propio sistema de transferencia, por ejemplo, mediante la expresión de un vector que contiene un sistema de genes funcionales que se puede expresar produciendo la proteína de fusión, o la propia proteína de fusión se puede introducir en el sistema de transporte. Generalmente, este último será el caso en los sistemas de transporte artificiales. Una ventaja importante de dicho sistema de transporte artificial es que se puede introducir en el medio ambiente, por ejemplo, para el tratamiento de un cultivo, sin riesgo de propagación del material genético.
Preferentemente, la proteína de fusión se introduce en la célula sin la introducción de una secuencia de ADN o ARN. En el caso de la utilización de Agrobacterium tumefaciens como sistema de transferencia, esto implica la ausencia de ADN-T. Esto significa que es posible modificar una célula sin introducir material genético en la célula a ser modificada.
Según una realización importante, la proteína de fusión introducida tiene actividad recombinasa.
En dicho caso, es de hecho posible llevar a cabo un cambio en el ADN cromosómico de la célula a ser modificada, sin la introducción de material genético adicional (un vector que codifica el ADN-T o péptido B) en la célula. Una aplicación importante, por ejemplo, es la eliminación de un gen marcador, cuya admisión en el medio ambiente no es deseable. En particular, esto puede incluir la eliminación de los genes de resistencia a antibióticos presentes entre secuencias de ADN en repetición directa a ser reconocidos por la recombinasa.
Convenientemente, una bacteria de la clase Rhizobiaceae se puede utilizar como sistema de transferencia.
Dichas bacterias, entre las que se encuentran Agrobacterium, Rhizobium y Phyllobacterium, son muy adecuadas para la modificación de plantas, levaduras u hongos. Otras bacterias que pertenecen a esta familia, tales como Brucella, son conocidas por su interacción con células animales y se pueden utilizar para la modificación de las mismas.
Según una realización preferente, se utiliza Agrobacterium, que se conoce que es muy adecuado para la modificación tanto de procariotas (bacterias) como de eucariotas (plantas, levaduras, hongos y células de animales) .
Según una realización preferente, una célula seleccionada del grupo que comprende i) una célula de plantas; ii) una célula de levaduras, y iii) una célula de hongos, se utiliza como la célula a ser modificada.
La presente invención también se refiere a un vector, estando dicho vector caracterizado porque codifica un sistema de transporte de proteínas que comprende un poro que contiene un complejo VirB y proteína VirD4, así como una proteína de fusión BA tal como se define en la reivindicación 7.
Dicho vector se puede introducir en un sistema de transferencia, tal como una bacteria. Entonces, el vector posee toda la información que necesita ser expresada para la transferencia de la proteína de fusión, por lo que es posible que la proteína de fusión sea transferida a la célula a ser modificada.
Por último, la presente invención se refiere a un conjunto de vectores, caracterizado porque el conjunto de vectores comprende uno o más vectores que codifican un sistema de transporte de proteínas que comprende un poro que contiene un complejo VirB y proteína VirD4, así como un vector adicional que codifica un péptido de fusión BA, tal como se define en la reivindicación 7.
La ventaja de dicho conjunto de vectores es que el vector o vectores que codifican el sistema de transporte de proteínas, se pueden introducir en un sistema de transferencia por separado a partir del vector adicional que codifica la proteína de fusión. Esto hace que sea posible la utilización de un sistema de transferencia, en particular una bacteria, como vehículo estándar para la modificación de una célula, en el que el sistema de transporte está provisto de un vector adicional que se expresa en el sistema de transporte para llevar a cabo la modificación.
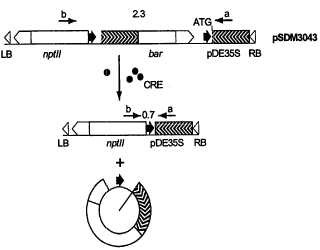
La presente invención se ilustrará con referencia a los siguientes ejemplos y al dibujo, en el que la única figura muestra de forma esquemática un sistema de ensayo para demostrar la actividad Cre en plantas.
Cepas bacterianas La cepa de Agrobacterium LBA1010 (Koekman y otros, Plasmid 7 (1982) ; 119-132; Centraal Bureau voor Schimmelcultures; Baarn, Países Bajos, número de acceso: PC2805) posee el plásmido Ti no modificado pTiB6 en un fondo cromosómico C58. LBA 1100 (Beijersbergen... [Seguir leyendo]
Reivindicaciones:
1. Método para llevar a cabo una modificación en una célula no humana, en el que un sistema de transferencia bacteriano se pone en contacto con la célula a ser modificada, dicho sistema de transferencia tiene un sistema de transporte de proteínas que comprende un poro, el canal de secreción de tipo IV, que incluye un complejo VirB y proteína VirD4, en el que el sistema de transferencia comprende una proteína de fusión o es capaz de crear una proteína de fusión que se introduce en la célula utilizando el sistema de transporte de proteínas, en el que se introduce una proteína de fusión BA en la célula a ser modificada, cuya proteína de fusión BA comprende
i) como parte A un oligopéptido que comprende los 37 aminoácidos más hacia el extremo C-terminal de VirF, cuando se fusiona con cre, o los 40 aminoácidos más hacia el extremo C-terminal de una proteína bacteriana transportada por un canal de secresión de tipo IV, en el que dicha proteína bacteriana transportada por un canal de secresión de tipo IV se selecciona del grupo que comprende VirF, VirD2, VirE2, VirE3 y MobA; y
ii) como parte B un polipéptido capaz de ejercer una actividad de modificación celular en la célula a ser modificada;
en el que el extremo C-terminal del polipéptido de la parte B está unido al extremo N-terminal de la parte A, con la condición de que si la proteína de fusión comprende una parte A derivada de VirE2, la proteína de fusión no comprende los 84 aminoácidos del extremo N-terminal de VirE2.
2. Método, según la reivindicación 1, caracterizado porque la proteína de fusión se forma mediante la expresión en el sistema de transferencia.
3. Método, según la reivindicación 1 ó 2, caracterizado porque la proteína de fusión se introduce en la célula a ser modificada sin la introducción de una secuencia de ADN o ARN.
4. Método, según cualquiera de las reivindicaciones 1-3, caracterizado porque la proteína de fusión introducida posee una actividad recombinasa.
5. Método, según cualquiera de las reivindicaciones 1-4, caracterizado porque se utiliza una bacteria de la clase Rhizobiaceae como sistema de transferencia.
6. Método, según cualquiera de las reivindicaciones 1-5, caracterizado porque la célula a ser modificada se selecciona del grupo que comprende i) una célula de plantas, ii) una célula de levaduras, y iii) una célula de hongos.
7. Vector, que codifica un sistema de transporte de proteínas bacterianas que tiene un poro, el canal de secresión de tipo IV, que incluye un complejo VirB y proteína VirD4, así como una proteína de fusión BA que comprende:
i) como parte A un oligopéptido que comprende los 37 aminoácidos más hacia el extremo C-terminal de VirF, cuando se fusiona con cre, o los 40 aminoácidos más hacia el extremo C-terminal de una proteína bacteriana transportada por un canal de secresión de tipo IV, en el que dicha proteína bacteriana transportada por un canal de secresión de tipo IV se selecciona del grupo que comprende VirF, VirD2, VirE2, VirE3 y MobA; y
ii) como parte B un polipéptido capaz de ejercer una actividad de modificación celular en la célula a ser modificada;
en el que el extremo C-terminal del polipéptido de la parte B está unido al extremo N-terminal de la parte A, con la condición de que si la proteína de fusión comprende una parte A derivada de VirE2, la proteína de fusión no comprende los 84 aminoácidos del extremo N-terminal de VirE2.
8. Conjunto de vectores, caracterizado porque el conjunto de vectores comprende uno o más vectores que codifican un sistema de transporte de proteínas bacterianas que tiene un poro, el canal de secresión de tipo IV, que incluye un complejo VirB y proteína VirD4, así como una proteína de fusión BA que comprende:
i) como parte A un oligopéptido que comprende los 37 aminoácidos más hacia el extremo C-terminal de VirF, cuando se fusiona con cre, o los 40 aminoácidos más hacia el extremo C-terminal de una proteína bacteriana transportada por un canal de secresión de tipo IV, en el que dicha proteína bacteriana transportada por un canal de secresión de tipo IV se selecciona del grupo que comprende VirF, VirD2, VirE2, VirE3 y MobA; y
ii) como parte B un polipéptido capaz de ejercer una actividad de modificación celular en la célula a ser modificada;
en el que el extremo C-terminal del polipéptido de la parte B está unido al extremo N-terminal de la parte A, con la condición de que si la proteína de fusión comprende una parte A derivada de VirE2, la proteína de fusión no comprende los 84 aminoácidos del extremo N-terminal de VirE2.
Patentes similares o relacionadas:
Cadena ligera de enteroquinasa modificada, del 22 de Julio de 2020, de NOVO NORDISK A/S: Un análogo de la cadena ligera de la enteroquinasa bovina que comprende una secuencia de aminoácidos establecida en la SEQ ID NO: 1, en donde dicho análogo comprende […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos y composiciones para ingeniería genómica, del 3 de Junio de 2020, de Sangamo Therapeutics, Inc: Una pareja de nucleasas de dedo de zinc (ZFN) que comprende una ZFN izquierda y una ZFN derecha, comprendiendo cada ZFN un dominio de escisión […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Antígenos de coagulasa estafilocócica y métodos para su uso, del 13 de Mayo de 2020, de UNIVERSITY OF CHICAGO: Una composición inmunógena que comprende al menos dos dominios 1-2 de coagulasa estafilocócica diferentes, en donde cada uno de los al menos dos dominios […]
Reconocimiento de unión a diana celular mediante un agente bioactivo usando transferencia de energía de resonancia de bioluminiscencia intracelular, del 6 de Mayo de 2020, de PROMEGA CORPORATION: Un sistema de ensayo que comprende: (a) una biblioteca de agentes bioactivos, cada uno de los cuales está fijado a un fluoróforo; (b) una diana celular fusionada a […]
Etiqueta de epítopo y método de detección, captura y/o purificación de polipéptidos etiquetados, del 15 de Abril de 2020, de ChromoTek GmbH: Péptido epítopo aislado que tiene de 12 a 25 aminoácidos, en donde la secuencia de aminoácidos comprende una secuencia según se define en SEQ ID NO: 32 (X1X2RX4X5AX7SX9WX11X12), […]
Utilización diagnóstica de un polipéptido de fusión que comprende una proteína vírica y un enzima MGMT, del 15 de Abril de 2020, de INSTITUT PASTEUR: Utilización in vitro de un polipéptido de fusión que comprende una proteína vírica y i) el enzima 6-metilguanina-ADN-metiltransferasa (MGMT, EC 2.1.1.63) o un homólogo […]