MÉTODO PARA LA DESAMBIGUACIÓN DEL METABOLISMO ACETILANTE DE UN INDIVIDUO.
La presente invención se encuadra en el campo de la medicina personalizada.
En particular, la presente invención se refiere a un método para la desambiguación del metabolismo acetilante del gen NAT2 de un individuo. Este método es de utilidad para la predicción del metabolismo de fármacos por parte de un individuo concreto con la finalidad de minimizar los riesgos de posibles efectos secundarios provocados por un determinado fármaco.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201030214.
Solicitante: UNIVERSIDAD DE EXTREMADURA.
Nacionalidad solicitante: España.
Inventor/es: GARCIA-AGUNDEZ PEREZ-COCA,JOSE AUGUSTO, GARCIA MARTIN,MARIA ELENA.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
Método para la desambiguación del metabolismo acetilante de un individuo.
Campo de la invención
La presente invención se encuadra en el campo de la medicina personalizada. En particular, la presente invención se refiere a un método para la desambiguación del metabolismo acetilante del gen NAT2 de un individuo. Este método es de utilidad para la predicción del metabolismo de fármacos por parte de un individuo concreto con la finalidad de minimizar los riesgos de posibles efectos secundarios provocados por un determinado fármaco.
Antecedentes de la invención
El metabolismo de los fármacos comprende un conjunto de reacciones bioquímicas que resultan en modificaciones de su estructura. Estas modificaciones pueden producir metabolitos inactivos, metabolitos activos o productos metabólicos con actividad farmacológica distinta a la del fármaco original. En la fase I del metabolismo de los fármacos, suelen darse oxidaciones, reducciones o hidrólisis. Estas reacciones suelen introducir en la estructura del fármaco un grupo reactivo que lo convierte en más activo químicamente. En la Fase II, suelen darse reacciones de conjugación, que por regla general, inactivan el fármaco y favorecen su eliminación renal o biliar. La mayoría, aunque no todas las reacciones de la fase 2 tienen lugar en el hígado. La enzima N-acetiltransferasa tipo 2 (NAT2), participa en la fase II o metabolismo hepático de los medicamentos. Según sus polimorfismos genéticos, y de acuerdo con su capacidad acetilante, los pacientes se clasifican en acetiladores lentos, medios o rápidos.
Entre los fármacos que se acetilan de forma polimórfica por la enzima NAT2 se encuentran fármacos antituberculosos como la isoniacida, o la rifampicina, así como metabolitos de otros fármacos como el metamizol o el clonacepam. Tradicionalmente la clasificación se hacia en base a ensayos fenotípicos llevados a cabo con sustratos como la cafeína o la isoniazida. Más recientemente, análisis en el gen que codifica la enzima NAT2, que se encuentra en el cromosoma 8p22 han permitido la identificación de varias decenas de alelos NAT2. Los individuos que son acetiladores rápidos, llevan uno o dos alelos que codifican para enzimas de alta actividad (NAT2*4 y NAT2*12) mientras que los acetiladores lentos tienen dos alelos de baja actividad. La actividad enzimática resultante de la combinación particular de alelos de cada individuo determina, no solo la concentración el fármaco, si no también su eficacia y su toxicidad. Así, la capacidad acetilante de los individuos condiciona mucho el espectro de antibióticos que toleran. Por ejemplo, en individuos sometidos a tratamientos contra la tuberculosis, los acetiladores lentos presentan una mayor probabilidad a desarrollar polineuropatías y hepatotoxicidad. De hecho, a dosis terapéuticas, existe una elevada asociación entre el genotipo NAT2 y el daño hepático severo, incluyendo el cáncer de hígado y vesícula. Por lo tanto, el genotipado de NAT2, previo a la terapia servirla para evitar daños adversos en los pacientes y para personalizar la dosis terapéutica más apropiada de este tipo de fármacos (Ohno et al., 2000. Int. J. Tuberc. Lung. Dis. 4:256-261). La determinación del genotipo NAT2 en la práctica clínica está dificultada por la existencia de combinaciones ambiguas de haplotipos que lleva a equivocaciones en la clasificación de los pacientes. Métodos para el genotipado de NAT2 en la práctica clínica van desde el análisis por amplificación-restricción, PCR, la secuenciación, a los microarrays. Sin embargo, estos métodos no pueden resolver el genotipo cuando existen varias posibles ordenaciones de mutaciones que conducen a diferentes fenotipos acetiladores. Como consecuencia los esfuerzos para evitar daños hepáticos irreversibles debido a la sobredosis en pacientes con fenotipo acetilante lento, no encuentran una solución efectiva. Para solucionar este problema se han diseñado diversas estrategias basadas en estadísticas para reconstruir los haplotipos NAT2 (Stephens et al., 2001; Stephens and Donnelly 2003; Golka et al., Arch Toxicol 2008, 82:265-70)
No obstante, estos métodos se basan exclusivamente en algoritmos matemáticos asumiendo que los diferentes haplotipos se asocian entre ellos de acuerdo con Leyes matemáticas de probabilidad. En la naturaleza esto no ocurre pues la presión evolutiva disminuye la esperanza de vida de los individuos portadores de aquellas combinaciones peor adaptadas, impidiendo en ocasiones incluso que lleguen a producir descendientes por lo que, en la naturaleza, la probabilidad de que una combinación determinada se de no obedece a leyes únicamente probabilísticas si no también biológicas.
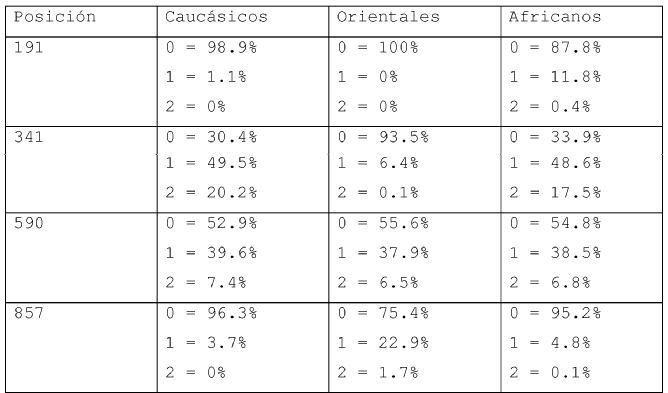
Un estudio comparativo entre los resultados que predicen el método matemático y lo que ocurre en la naturaleza, demuestra que la determinación del genotipo NAT2 no puede ser resuelta claramente mediante técnicas de predicción matemáticas. En este estudio, un 0.8% de los individuos fueron clasificados erróneamente cuando se utilizaron únicamente los algoritmos matemáticos para la predicción (Agundez et al., Clinical Chemistry 2008 54:8). El método de desambiguación descrito en dicho estudio se basaba en el análisis de haplotipos para 7 polimorfismos comunes en el gen NAT2 (191, 282, 341, 481, 590, 803 y 857), sin embargo, tenía el problema de que estaba limitado a datos recogidos solo para la raza caucásica y por tanto solo es aplicable a la desambiguación del fenotipo acetilador en individuos de esta raza. Además este método no proporciona la indicación de la probabilidad de predicción correcta.
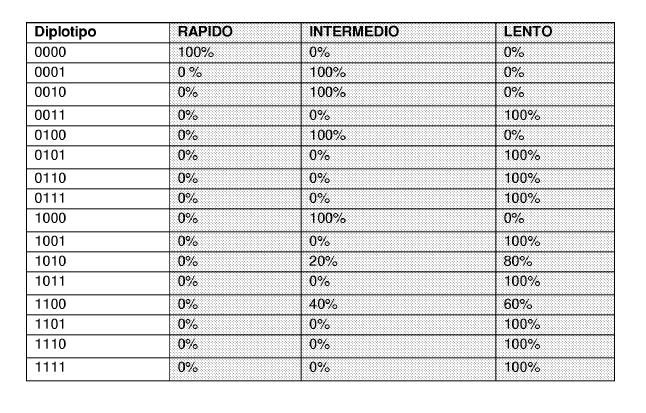
La presente invención proporciona un método para la desambiguación del fenotipo acetilador que no solo soluciona estos problemas sino que además tiene la ventaja de que se basa en el estudio de 4 polimorfismos en vez de 7 lo cual representa una disminución sustancial del trabajo analítico. El método, además, permite directamente inferir el fenotipo acetilador a partir de diplotipos en vez de haplotipos lo que evita el paso de tener que deconstruir los genotipos para luego reconstruirlos por predicción de haplotipos. Por último, el método de la presente invención es aplicable para la desambiguación del fenotipo acetilador de cualquier individuo independientemente de su raza.
Breve descripción de las figuras
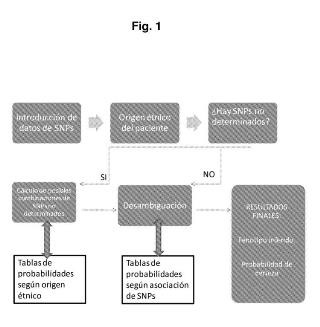
Figura 1: esta figura representa esquemáticamente los pasos del método para la desambiguación del fenotipo acetilador del gen NAT2 de la presente invención.
Descripción de la invención
La presente invención tiene por objeto un método para la desambiguación del fenotipo acetilante del gen NAT2 de un individuo que comprende las etapas de:
Reivindicaciones:
1. Método para la desambiguación del fenotipo acetilante del gen NAT2 de un individuo que comprende las etapas de:
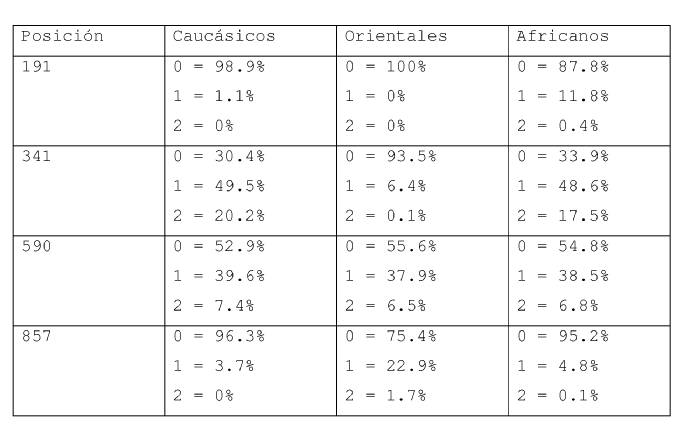
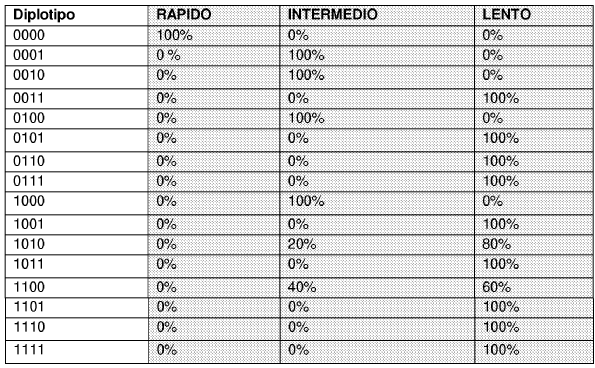
2. Método de acuerdo con la reivindicación 1 donde en el punto i) se determinan todos los puntos polimórficos 191, 341, 590 y 857 del gen NAT2.
3. Un programa informático que comprende medios de código de programa informático adaptados para realizar las etapas del método según cualquiera de las reivindicaciones 1 a 2, cuando dicho programa se ejecuta en un ordenador, un procesador de señal digital, una disposición de puertas de campo programable, un circuito integrado de aplicación específica, un microprocesador, un microcontrolador, y cualquier otra forma de hardware programable.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]