Estructura de los ribosomas e inhibidores de la síntesis de proteínas.
Un método para identificar una molécula que se une a una gran subunidad ribosómica,
en donde el método comprende las etapas siguientes:
(a) proveer un modelo molecular que comprende una o más regiones objetivo seleccionadas del grupo que consiste en al menos una porción de: (i) un sitio de unión de anisomicina, (ii) un sitio de unión de azitromicina, (iii) un sitio de unión de blasticidina, (iv) un sitio de unión de carbomicina, (v) un sitio de unión de eritromicina, (vi) un sitio de unión de linezólido, (vii) un sitio de unión de esparsomicina, (viii) un sitio de unión de espiramicina, (ix) un sitio de unión de tilosina, y (x) un sitio de unión de virginiamicina M, en donde el modelo molecular se hace
(1) a partir de las coordenadas atómicas correspondientes a la gran subunidad ribosómica de Haloarcula marismortui que se hallan en el Disco 1 bajo los nombres de archivo ANISOMYCIN.PDB, azithromycin.pdb, BLASTICIDIN.PDB,
CARBOMYCIN.PDB, erythromycin. pdb, linezolid.pdb, SPARSOMYCIN.PDB, SPIRAMYCIN.PDB, TYLOSIN.PDB, o VIRGINAMYCIN. PDB, o que se hallan en el Disco 1, bajo FOLDERB, bajo los nombres de archivo ANISOMYC.PDB, BLASTICI.PDB, CARBOMYC. PDB, SPARSOMY.PDB, SPIRAMYC.PDB, TYLOSIN.PDB, VIRGINIA.PDB, o que se hallan en el Disco 1, bajo FOLDERC, bajo los nombres de archivo AZITHROM.PDB, o LINEZOLI.PDB, o
(2) a partir de las coordenadas atómicas derivadas mediante modelación molecular mediante la utilización de coordenadas correspondientes a la gran subunidad ribosómica de Haloarcula marismortui que se hallan en el Disco 1 bajo los nombres de archivo ANISOMYCIN.PDB, azithromycin. pdb, BLASTICIDIN.PDB, CARBOMYCIN.PDB, erythromycin.pdb, linezolid.pdb, SPARSOMYCIN.PDB, SPIRAMYCIN.PDB, TYLOSIN.PDB, o VIRGINAMYCIN.PDB, o que se hallan en el Disco 1, bajo FOLDERB, bajo los nombres de archivo ANISOMYC.PDB, BLASTICI.PDB, 20 CARBOMYC.PDB, SPARSOMY.PDB, SPIRAMYC.PDB, TYLOSIN.PDB, VIRGINIA.PDB, o que se hallan en el Disco 1, bajo FOLDERC, bajo los nombres de archivo AZITHROM.PDB, o LINEZOLI.PDB; y
(b) el uso del modelo molecular para identificar una molécula candidata que pueda unirse al modelo molecular
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E02255442.
Solicitante: YALE UNIVERSITY.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 451 COLLEGE STREET NEW HAVEN, CT 06520 ESTADOS UNIDOS DE AMERICA.
Inventor/es: STEITZ,THOMAS A, MOORE,PETER B, BAN,NENAD, NISSEN,POUL, HANSEN,JEFFREY, IPPOLITO,JOSEPH A.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07K14/215 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 14/00 Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados. › de Halobacteriaceae (F).
- C12Q1/18 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › Investigación o análisis de la actividad antimicrobiana de un material.
- G01N33/68 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › en los que intervienen proteínas, péptidos o aminoácidos.
- G06F17/50
PDF original: ES-2448815_T3.pdf
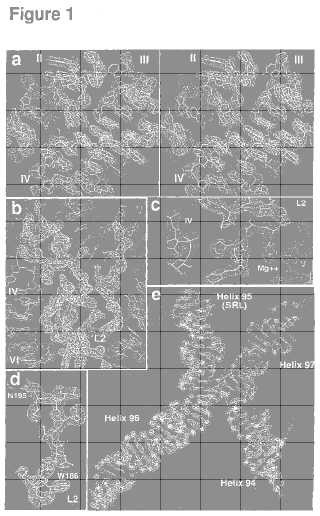
Fragmento de la descripción:
Estructura de los ribosomas e inhibidores de la sintesis de proteinas La presente invencion se refiere en general al campo de la biosintesis de proteinas y a moduladores, por ejemplo, inhibidores, de la biosintesis de proteinas. Mas particularmente, la invencion se refiere a: metodos y composiciones para elucidar la estructura tridimensional de la unidad ribosomica grande, ya sea sola o en combinacion con un inhibidor de la sintesis de proteinas; a la estructura tridimensional de la subunidad ribosomica grande, ya sea sola o en combinacion con un inhibidor de la sintesis de proteinas; al uso de tales estructuras en el disefo y el ensayo de nuevos inhibidores de la sintesis de proteinas; y a los nuevos inhibidores de la sintesis de proteinas.
ANTECEDENTES
I. Ribosomas: estructura, funcion y composicion Los ribosomas son ribonucleoproteinas que estan presentes en los procariotas y en los eucariotas. Comprenden alrededor de dos tercios de ARN y un tercio de proteina. Los ribosomas son los organulos celulares responsables de la sintesis de proteinas. Durante la expresion genica los ribosomas traducen la informacion genetica codificada en un ARN mensajero a la proteina (Garrett et al. (2000) "The Ribosome: Structure, Function, Antibiotics and Cellular Interactions", American Society for Microbiology, Washington, D.C.) .
Los ribosomas comprenden dos subunidades de ribonucleoproteinas no equivalentes. La subunidad mas grande (conocida tambien como la "subunidad ribosomica grande") es alrededor de dos veces mas grande en tamafo que la subunidad mas pequefa (tambien conocida como la "subunidad ribosomica pequefa") . La subunidad ribosomica pequefa se une al ARN mensajero (ARNm) y media en las interacciones entre el ARNm y los anticodones del ARN de transferencia (ARNt) de los cuales depende la fidelidad de la traduccion. La subunidad ribosomica grande cataliza la formacion de uniones peptidicas -la reaccion de la peptidiltransferasa de la sintesis de proteinas- e incluye (al menos) dos diferentes sitios de union del ARNt: el sitio A que aloja al aminoacil-ARNt entrante, que contribuira su aminoacido a la cadena peptidica creciente, y el sitio P que aloja el complejo peptidil-ARNt, es decir, el ARNt unido a todos los aminoacidos que han sido agregados hasta ese momento a la cadena peptidica. La subunidad ribosomica grande tambien incluye uno o mas sitios de union para los factores de la proteina G que ayudan a las fases de iniciacion, elongacion y terminacion de la sintesis de proteinas. Las subunidades ribosomicas grande y pequefa se comportan en forma independiente durante la fase de iniciacion de la sintesis de proteinas; sin embargo, se ensamblan como ribosomas completos cuando esta por comenzar la elongacion.
El peso molecular del ribosoma procariotico es de aproximadamente 2, 6 x 106 daltons. En los procariotas, la subunidad ribosomica pequefa contiene un ARN ribosomico (ARNr) 16S (unidades Svedberg) que tiene un peso molecular de aproximadamente 5, 0 x 105 daltons. La subunidad ribosomica grande contiene un ARNr 23S que tiene un peso molecular de aproximadamente 1, 0 x 106 daltons y un ARNr 5S que tiene un peso molecular de aproximadamente 4, 0 x 105 daltons. La subunidad pequefa procariotica contiene alrededor de 20 proteinas diferentes y su subunidad grande contiene alrededor de 35 proteinas. Las subunidades ribosomicas grande y pequefa juntas constituyen un ribosoma 70S en los procariotas.
Los ribosomas eucarioticos son en general mas grandes que sus contrapartes procarioticas. En los eucariotas, las subunidades grandes y pequefas juntas forman un ribosoma 80S. La subunidad pequefa de un ribosoma eucariotico incluye un solo ARNr 18S, mientas que la subunidad grande incluye un ARNr 5S, un ARNr 5.8S y un ARNr 28S. El ARNr 5.8S esta relacionado estructuralmente con el extremo 5' del ARNr 23S procariotico, y el ARNr 28S esta relacionado estructuralmente con el resto del ARNr 23S procariotico 3S (Moore (1998) Annu. Rev. Biophys.
27: 35-58) . Las proteinas ribosomicas eucarioticas son cualitativamente similares a las proteinas ribosomicas procarioticas; sin embargo, las proteinas eucarioticas son mas grandes y mas numerosas (Moore (1998) supra) .
II. Conservacion estructural de la subunidad ribosomica grande Si bien la composicion quimica de las subunidades ribosomicas grandes varian de especie a especie, las secuencias de sus componentes proveen evidencia inequivoca de que son similares en estructura tridimensional, funcionan de una manera similar y estan relacionadas evolutivamente. Las implicancias evolutivas de los datos de secuencias de ARNr disponibles se sintetizan en los articulos de Woese y otros en la parte II de "Ribosomal RNA. Structure, Evolution, Processing and Function in Protein Biosynthesis", (Zimmermann y Dahlberg, eds., CRC Press, Boca Raton, FL, 1996) . El articulo de Garret y Rodriguez-Fonseca en la parte IV del mismo volumen comenta el nivel inusualmente alto de la conservacion de secuencias observado en la region de la peptidiltransferasa de la subunidad ribosomica grande. Los ribosomas de especies de archeas tales como Haloarcula marismortu se asemejan a los obtenidos de las especies eubacterianas tales como E. coli en tamafo y complejidad. Sin embargo, las proteinas en los ribosomas de H. marismortui esta relacionadas mas estrechamente con las proteinas ribosomicas halladas en los eucariotas (Wool et al. (1995) Biochem. Cell Biol. 73: 933-947) .
III. Determinacion de la estructura de los ribosomas Mucho de lo que se conoce con respecto a la estructura de los ribosomas se deriva de los metodos fisicos y quimicos que producen informacion de resolucion relativamente baja. La microscopia electronica (EM, por sus siglas en ingles) ha contribuido a una comprension de la estructura de los ribosomas desde que se descubrio el ribosoma. En los afos 70, la EM de baja resolucion revelo la forma y la organizacion cuaternaria del ribosoma. A fines de los afos 80, las posiciones de los epitopes superficiales de todas las proteinas en la subunidad pequefa de E. coli, asi como tambien muchos en la subunidad grande habian sido mapeados usando tecnicas de microscopia inmunoelectronica (Oakes et al. (1986) , Structure. Function and Genetics of Ribosomes, (Hardesty, B. y Kramer, G., eds.) Springer-Verlag, Nueva York, NY, pp. 47-67; Stoeffler et al. (1986) , Structure, Function and Genetics of Ribosomes, (Hardesty, B. y Kramer, G., eds.) Springer-Verlag, Nueva York, NY, pp. 28-46) . En los ultimos afos, los progresos realizados en la crio-EM de particulas simples y la reconstruccion de imagenes han llevado a las reconstrucciones tridimensionales del ribosoma de E. coli 70S y sus complejos con ARNts y factores de elongacion a resoluciones de entre 15 A y 25 A (Stark et al. (1995) Structure 3: 815-821; Stark et al. (1997) Nature 3898: 403-406; Agrawal et al. (1996) Science 271: 1000-1002; Stark et al. (1997) Cell 28: 19-28) . Adicionalmente, las imagenes de EM tridimensionales del ribosoma han sido producidas a resoluciones suficientemente altas de modo que muchas de las proteinas y acidos nucleicos que ayudan a la sintesis de proteinas pueden ser visualizadas unidas al ribosoma. Un modelo aproximado de la estructura de ARN en la subunidad grande ha sido construido para ajustarse a unmapa de microscopia electronica de 7, 5 A de resolucion de la subunidad 50S de E. coli y los datos bioquimicos disponibles (Mueller et al. (2000) J. Mol. Biol. 298: 35-59) .
Si bien las imagenes provistas por la EM han sido utiles, se ha reconocido desde hace tiempo que una comprension completa de la estructura de los ribosomas se derivaria solo de la cristalografia de rayos X. En 1979, Yonath y Wittman obtuvieron los primeros cristales potencialmente utiles de ribosomas y subunidades ribosomicas (Yonath et al. (1980) Biochem. Internat. 1: 428-435) . A mediados de los afos 1980, los cientificos prepararon cristales de ribosomas para la cristalografia de rayos X (Maskowski et al. (1987) J. Mol. Biol. 193: 818-822) . Los primeros cristales de la subunidad ribosomica 50S de H. marismortui se obtuvieron en 1987. En 1991, se informaron mejoras en la resolucion de los datos de difraccion que se podian obtener de los cristales de la subunidad ribosomica 50S de H. marismortui (van Bohlen, K. (1991) J. Mol. Biol. 222: 11) .
En 1995, se informaron mapas de densidad electronica de baja resolucion para las subunidades ribosomicas grandes y pequefas de fuentes halofilicas y termofilicas (Schlunzen et al. (1995) Biochem. Cell Biol. 73: 739-749) . Sin embargo, estos mapas de densidad electronica de baja resolucion demostraron ser falsos (Ban et al. (1998) Cell
93: 1105-1115) .
El primer mapa de densidad electronica del ribosoma que mostro caracteristicas reconocibles como ARN doble fueun mapa cristalografico de rayos X de 9 A de resolucion de la subunidad... [Seguir leyendo]
Reivindicaciones:
1. Un metodo para identificar una molecula que se une a una gran subunidad ribosomica, en donde el metodo comprende las etapas siguientes:
(a) proveer un modelo molecular que comprende una o mas regiones objetivo seleccionadas del grupo que consiste en al menos una porcion de: (i) un sitio de union de anisomicina, (ii) un sitio de union de azitromicina, (iii) un sitio de union de blasticidina, (iv) un sitio de union de carbomicina, (v) un sitio de union de eritromicina, (vi) un sitio de union de linezolido, (vii) un sitio de union de esparsomicina, (viii) un sitio de union de espiramicina, (ix) un sitio de union de tilosina, y (x) un sitio de union de virginiamicina M, en donde el modelo molecular se hace
(1) a partir de las coordenadas atomicas correspondientes a la gran subunidad ribosomica de Haloarcula marismortui que se hallan en el Disco 1 bajo los nombres de archivo ANISOMYCIN.PDB, azithromycin.pdb, BLASTICIDIN.PDB, CARBOMYCIN.PDB, er y thromycin. pdb, linezolid.pdb, SPARSOMYCIN.PDB, SPIRAMYCIN.PDB, TYLOSIN.PDB, o VIRGINAMYCIN. PDB, o que se hallan en el Disco 1, bajo FOLDERB, bajo los nombres de archivo ANISOMYC.PDB, BLASTICI.PDB, CARBOMYC. PDB, SPARSOMY.PDB, SPIRAMYC.PDB, TYLOSIN.PDB, VIRGINIA.PDB, o que se hallan en el Disco 1, bajo FOLDERC, bajo los nombres de archivo AZITHROM.PDB, o LINEZOLI.PDB, o
(2) a partir de las coordenadas atomicas derivadas mediante modelacion molecular mediante la utilizacion de coordenadas correspondientes a la gran subunidad ribosomica de Haloarcula marismortui que se hallan en el Disco 1 bajo los nombres de archivo ANISOMYCIN.PDB, azithromycin. pdb, BLASTICIDIN.PDB, CARBOMYCIN.PDB, er y thromycin.pdb, linezolid.pdb, SPARSOMYCIN.PDB, SPIRAMYCIN.PDB, TYLOSIN.PDB, o VIRGINAMYCIN.PDB,
(b) el uso del modelo molecular para identificar una molecula candidata que pueda unirse al modelo molecular.
2. El metodo de acuerdo con la reivindicacion 1, en donde en la etapa (a) (2) la modelacion molecular consiste en modelacion por homologia.
3. El metodo de acuerdo con la reivindicacion 1, en donde en la etapa (a) (2) la modelacion molecular consiste en el reemplazo molecular.
4. El metodo de acuerdo con la reivindicacion 1, que comprende la etapa adicional de determinar si la molecula candidata modula actividad ribosomica.
5. El metodo de acuerdo con la reivindicacion 4, que comprende la etapa adicional de repetir una o mas de las etapas (a) y (b) para identificar una molecula candidata modificada.
6. El metodo de acuerdo con la reivindicacion 5, que comprende la etapa adicional de determinar si la molecula candidata modificada modula actividad ribosomica.
7. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo ANISOMYCIN.PDB, o en el Disco 1, bajo FOLDERB, bajo el nombre de archivo ANISOMYC.PDB.
8. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo azithromycin.pdb o en el Disco 1, bajo FOLDERC, bajo el nombre de archivo AZITHROM.PDB.
9. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo BLASTICIDIN.PDB o en el Disco 1, bajo FOLDERB, bajo el nombre de archivo BLASTICI.PDB.
10. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo CARBOMYCIN.PDB o en el Disco 1, bajo FOLDERB, bajo el nombre de archivo CARBOMYC.PDB.
11. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo er y thromycin.pdb.
12. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo linezolid.pdb o en el Disco 1, bajo FOLDERC, bajo el nombre de archivo LINEZOLI.PDB.
13. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo SPARSOMYCIN.PDB, o en el Disco 1, bajo FOLDERB, bajo el nombre de archivo SPARSOMY.PDB.
14. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo SPIRAMYCIN.PDB o en el Disco 1, bajo FOLDERB, bajo el nombre de archivo SPIRAMYC.PDB.
15. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo TYLOSIN.PDB o en el Disco 1, bajo FOLDERB, bajo el nombre de archivo TYLOSIN.PDB,
16. El metodo de acuerdo con la reivindicacion 1, en donde las coordenadas atomicas se encuentran en el Disco 1 bajo el nombre de archivo VIRGINIAMYCIN.PDB o en el Disco 1, bajo FOLDERB, bajo el nombre de archivo VIRGINIA.PDB.
Se buscan diversos componentes de PTC
Patentes similares o relacionadas:
Inmunomoduladores, del 29 de Julio de 2020, de BRISTOL-MYERS SQUIBB COMPANY: Un compuesto de la fórmula (I) **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo, en donde: A se selecciona de **(Ver fórmula)** en donde: […]
Métodos y composiciones para el diagnóstico y pronóstico de lesión renal e insuficiencia renal, del 29 de Julio de 2020, de Astute Medical, Inc: Un método para evaluar el estado renal en un sujeto, que comprende: realizar una pluralidad de ensayos configurados para detectar una […]
Neuregulina para tratar la insuficiencia cardíaca, del 29 de Julio de 2020, de Zensun (Shanghai) Science & Technology, Co., Ltd: Neuregulina para usar en un método para tratar la insuficiencia cardíaca crónica en un paciente, donde el paciente tiene un nivel plasmático de NT-proBNP […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Método para llevar a cabo el seguimiento de la enfermedad de Gaucher, del 15 de Julio de 2020, de Centogene GmbH: Un método para determinar la evolución de la enfermedad de Gaucher en un sujeto, que comprende la etapa de determinar en varios puntos en el […]
Procedimiento para evaluación de la función hepática y el flujo sanguíneo portal, del 15 de Julio de 2020, de The Regents of the University of Colorado, a body corporate: Procedimiento in vitro para la estimación del flujo sanguíneo portal en un individuo a partir de una única muestra de sangre o suero, comprendiendo el procedimiento: […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos de monitorización terapéutica de profármacos de ácido fenilacético, del 24 de Junio de 2020, de Immedica Pharma AB: Glicerilo tri-[4-fenilbutirato] (HPN-100) para su uso en un método para tratar un trastorno del ciclo de la urea en un sujeto que tiene discapacidad […]