Oligonucleótidos.
Un oligonucleótido monocatenario, que no tiene una secuencia que contiene partes hechas de cuatro o más restos nucleotídicos consecutivos que son el complemento inverso de cada uno de los otros que permite el apareamiento intramolecular de bases,
y que está formado de restos nucleotídicos donde las bases de dos o más restos nucleotídicos internos están marcadas con un fluoróforo sin un inactivador asociado, donde al menos dos de los restos nucleotídicos marcados con un fluoróforo están separados por al menos dos restos nucleotídicos no marcados, donde el resto nucleotídico 3' no contiene un hidroxilo 3' y donde cada fluoróforo es el mismo fluoróforo.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2006/002719.
Solicitante: LGC LIMITED.
Nacionalidad solicitante: Reino Unido.
Dirección: QUEENS ROAD TEDDINGTON, MIDDLESEX TW11 0LY REINO UNIDO.
Inventor/es: FRENCH, DAVID, JOHN, MCDOWELL, DAVID, GORDON.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2445624_T3.pdf
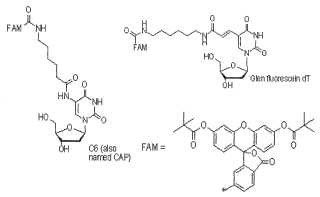
Fragmento de la descripción:
Oligonucleótidos La presente invención se refiere a oligonucleótidos, en particular a oligonucleótidos marcados de forma fluorescente para su uso como sondas de hibridación.
Se conoce una multitud de tecnologías para detectar secuencias específicas de ADN y valorar los polimorfismos conocidos de un único nucleótido. Varios de estos métodos de detección utilizan hibridación de sondas fluorescentes y dependen de la transferencia de energía entre los restos donante y aceptor.
La reacción en cadena de la polimerasa (PCR) homogénea es un método que acopla los procesos de amplificación y detección. Los beneficios de la PCR homogénea sobre los métodos tradicionales, tales como amplificación de diana seguido por electroforesis en gel, son una reducción en la duración del ensayo, las manipulaciones y el potencial de contaminación cruzada. Se emplean sondas fluorescentes con instrumentos de PCR en tiempo real, tales como el LightCycler (Roche) , ABI PRISM 7700 y 7900 (Applied Biosystems) , Smartcycler (Cepheid) , Rotogene (Corbett Research) , MX4000 (Stratagene) , SynChron (Biogene) e iCycler (BioRad) , para detectar e identificar productos amplificados durante (a tiempo real) o después (punto final) de la amplificación. Ejemplos de tecnologías basadas en sondas incluyen, balizas moleculares (Tyagi et al, 1996) , el ensayo de 5'-exonucleasa (documento US 5.691.146) , sondas de hibridación (documento US 6.174.670) , cebadores Scorpion (Whitcombe et al, 1999) , y ResonSense (Lee et al, 2002) . Las sondas fluorescentes normalmente están en un estado inactivado o no excitado hasta que su hibridación con una secuencias diana provoca la desactivación, tal como por cambios conformacionales o degradación de la sonda, o excitación como resultado de interacciones de la sonda. Estos sistemas de sonda dependen de la transferencia de energía entre los restos donante (por ejemplo fluoróforo) y aceptor (por ejemplo, inactivador o fluoróforo secundario) . La energía absorbida por un fluoróforo puede transferirse a un inactivador o un fluoróforo aceptor y liberarse como calor o emitirse como luz de una diferente longitud de onda. La inactivación de la señal fluorescente puede suceder por mecanismos de Transferencia de Energía por Resonancia de Fluorescencia (FRET) o no FRET. La inactivación FRET requiere un solapamiento espectral entre el donante y el aceptor, donde la eficacia de la inactivación está relacionada con la distancia entre los dos restos. La inactivación no FRET sucede a través de "contactos" de corto intervalo entre el fluoróforo y el inactivador, lo que no requiere solapamiento espectral entre los restos.
El ensayo de 5'-exonucleasa (TaqManT) usa inactivación FRET para detectar el ADN diana amplificado por PCR. Las sondas TaqMan son oligonucleótidos que contienen restos de fluoróforo e inactivador preferiblemente localizados en los extremos 5' y 3'. Se emite muy poca fluorescencia desde la sonda intacta debido a la inactivación intramolecular eficaz. Sin embargo, durante la amplificación por PCR, la sonda hibrida específicamente con su secuencia diana y la actividad 5'-3'-exonucleasa de la Taq polimerasa escinde la sonda entre los restos de fluoróforo e inactivador. La escisión enzimática de las sondas TaqMan separa espacialmente los componentes de fluoróforo e inactivador, causando aumentos significativos en la emisión de fluorescencia correlacionados con la amplificación de la diana. Un diseño cuidadoso de las sondas TaqMan permite la discriminación de dianas polimórficas, donde solamente se degradan las sondas perfectamente apareadas para generar aumentos en la señal de fluorescencia. Como las sondas TaqMan se digieren durante la amplificación por PCR en tiempo real, las sondas no están disponibles para análisis de secuencias post-amplificación.
Las sondas Eclipse son oligonucleótidos lineales cortos que tienen un agente de unión al surco menor (MGB) y un resto inactivador en el extremo 5' y un fluoróforo unido al extremo 3'. Se cree que el MGB evita la actividad exonucleasa de Taq que escinde la sonda. El enrollamiento aleatorio de la sonda sitúa a los componentes de fluoróforo e inactivador en cercana proximidad causando inactivación de fluorescencia. La hibridación con las secuencias diana causa que la sonda se enderece de modo que los restos de fluoróforo e inactivador queden separados espacialmente.
Las Balizas Moleculares son sondas oligonucleotídicas monocatenarias que son no fluorescentes en aislamiento, pero que llegan a ser fluorescentes tras la hibridación con las secuencias diana. Las Balizas Moleculares no hibridadas forman estructuras de tallo-bucle, que tienen un fluoróforo unido covalentemente a un extremo de la molécula y un inactivador unido al otro, de modo que la horquilla de la baliza sitúa al resto de fluoróforo en cercana proximidad del inactivador. Cuando las Balizas Moleculares hibridan con las secuencias diana, los restos de fluoróforo e inactivador quedan espacialmente separados, de modo que el fluoróforo ya no está inactivado y la Baliza Molecular emite fluorescencia. Pueden usarse Balizas Moleculares en ensayos en tiempo real y de punto final para la detección de secuencia y discriminación de SNP. La estructura secundaria de la Baliza Molecular transmite alta especificidad a la sonda, permitiendo la identificación de dianas que difieren en un único nucleótido. Sin embargo, la interacción intramolecular de la Baliza Molecular produce potencialmente una fuente de competición para la hibridación de la diana intermolecular y, como la Baliza Molecular es una sonda interna, debe competir con la hebra opuesta del amplicón por la unión con la secuencia diana. La combinación de ambas formas de competición puede reducir la eficacia de hibridación de la Baliza Molecular con algunas moléculas diana.
Los Scorpion son cebadores de PCR con una cola de tallo-bucle que contiene un fluoróforo y un inactivador (Whitcombe et al, 1999) . La estructura de tallo-bucle sitúa los componentes de fluoróforo e inactivador en cercana proximidad. El componente de bucle del Scorpion contiene la secuencia de sonda y la detección de la diana sucede por un mecanismo intramolecular altamente eficaz. Durante la PCR, los Scorpion se extienden para formar productos que incluyen la secuencia diana sonda. La cola del Scorpion se pliega sobre y posibilita la hibridación de la sonda y las secuencias diana. La hibridación de la secuencia de sonda con la diana amplificada está termodinámicamente favorecida sobre la estructura de tallo-bucle y un diseño cuidadoso de la sonda posibilita la discriminación de dianas que difieren en tan solo un único nucleótido. La hibridación de la sonda causa que la estructura de tallo-bucle se disocie de modo que los componentes de fluoróforo e inactivador queden espacialmente separados y se generan grandes aumentos en la emisión de fluorescencia. La cola de tallo-bucle se separa de la secuencia cebadora de PCR por un tapón de PCR (por ejemplo, HEG) que evita que la ADN polimerasa copie la secuencia de tallo-bucle del Scorpion. El Scorpion dúplex (Solinas et al, 2001) utiliza dos oligonucleótidos marcados, uno que comprende el cebador, el fluoróforo y la secuencia de sonda, y el otro que contiene el resto inactivador. De nuevo, la extensión del Scorpion dúplex posibilita que la secuencia de sonda se pliegue sobre e hibride la diana amplificada, disociando de este modo los oligonucleótidos fluoróforo e inactivador y aumentando la cantidad de señal fluorescente emitida.
Las sondas de hibridación son oligonucleótidos que están marcadas individualmente con restos de fluoróforo. Se requieren dos de dichos oligonucleótidos para cada ensayo de sonda de hibridación, uno marcado con un fluoróforo donante y el otro con un fluoróforo aceptor. La fluoresceína se emplea habitualmente como el donante y Cy5, LC-RED 640 y LC-RED 705 se usan habitualmente como aceptores. La excitación del fluoróforo donante produce un espectro de emisión que solapa con el espectro de absorción del fluoróforo aceptor. Se diseñan pares de sondas de hibridación para reconocer secuencias de nucleótidos adyacentes dentro de moléculas diana. En aislamiento, el oligonucleótido aceptor no se excita y no genera una señal fluorescente. Sin embargo, durante la hibridación con las secuencias diana polinucleotídicas, las sondas donante y aceptora se sitúan en cercana proximidad, permitiendo la transferencia de energía por resonancia de fluorescencia desde el donante hasta el aceptor. La señal fluorescente desde el fluoróforo aceptor se emite solamente cuando ambas sondas se hibridan con la molécula diana. Cuando se incorporan en reacciones de PCR, la fluorescencia desde la sonda aceptora se controla una vez por ciclo de amplificación, para facilitar la... [Seguir leyendo]
Reivindicaciones:
1. Un oligonucleótido monocatenario, que no tiene una secuencia que contiene partes hechas de cuatro o más restos nucleotídicos consecutivos que son el complemento inverso de cada uno de los otros que permite el apareamiento intramolecular de bases, y que está formado de restos nucleotídicos donde las bases de dos o más restos nucleotídicos internos están marcadas con un fluoróforo sin un inactivador asociado, donde al menos dos de los restos nucleotídicos marcados con un fluoróforo están separados por al menos dos restos nucleotídicos no marcados, donde el resto nucleotídico 3' no contiene un hidroxilo 3' y donde cada fluoróforo es el mismo fluoróforo.
2. Un oligonucleótido de acuerdo con la reivindicación 1, donde hasta aproximadamente un tercio de los restos internos están marcados con un fluoróforo.
3. Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones anteriores, donde todos los restos nucleotídicos marcados con un fluoróforo están separados por al menos dos restos nucleotídicos no marcados.
4. Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones anteriores y que contiene al menos un resto G, donde al menos uno de los restos nucleotídicos marcados con fluoróforo está posicionado adyacente a al menos uno de los restos G.
5. Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones anteriores, donde el resto nucleotídico 3' contiene un fosfato 3' o es un resto 3' desoxi.
6. Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones anteriores que contiene de 10 a 50 restos nucleotídicos.
7. Un oligonucleótido de acuerdo con la reivindicación 6 que contiene de 15 a 30 restos nucleotídicos.
8. Un oligonucleótido de acuerdo con la reivindicación 7 que contiene de 2 a 10 restos nucleotídicos internos marcados con fluoróforo.
9. Un oligonucleótido de acuerdo con la reivindicación 8 que contiene de 2 a 5 restos internos marcados con fluoróforo.
10. Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones anteriores, donde el fluoróforo se selecciona entre el grupo que consiste en colorantes basados en fluoresceína tales como 6-carboxifluoresceína (FAM) , tetraclorofluoresceína (TET) , hexaclorofluoresceína (HEX) , colorantes basados en rodamina tales como 6carboxi-X-rodamina (ROX) , 6-carboxitetrametilrodamina (TAMRA) ; un colorante de la familia Cy tal como Cy3 o Cy5; otros fluoróforos tales como NED o JOE, y colorantes Alexa, colorantes Atto, colorantes Dyomic, colorantes Dyomic megastoke y colorantes Thilyte.
11. Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones 1 a 10, donde el fluoróforo no se intercala en ADN estándar bicatenario.
12. Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones anteriores que tiene una secuencia complementaria a una secuencia polinucleotídica diana.
13. Un oligonucleótido de acuerdo con la reivindicación 12, donde la secuencia polinucleotídica diana se selecciona entre el grupo que consiste en el genoma humano incluyendo cualquier gen en el mismo o alelo del mismo, el genoma de ratón incluyendo cualquier gen en el mismo o alelo del mismo, y el genoma de cualquier agente infeccioso incluyendo cualquier gen en el mismo o alelo del mismo; o donde el oligonucleótido tiene una secuencia complementaria al gen PrP de oveja.
14. Un oligonucleótido de acuerdo con la reivindicación 13 que tiene una secuencia complementaria a uno cualquiera de los siguientes genes humanos o un alelo de los mismos:
N-acetiltransferasa 2 Factor V Leiden Factor II MTHFR Anemia falciforme
15. Un oligonucleótido de acuerdo con la reivindicación 13 que tiene una secuencia complementaria al genoma o a cualquier gen en el mismo o alelo del mismo de los siguientes agentes infecciosos:
Chlamydia trachomatis Adenovirus Influenza A
Streptococcus pneumoniae 16 Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones 13 a 15 que tiene una secuencia completamente complementaria a un alelo de un gen.
17. Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones 12 a 15 que tiene una identidad de secuencia de al menos el 70 % con el complemento de la secuencia polinucleotídica diana.
18. Un oligonucleótido de acuerdo con la reivindicación 17 que tiene una identidad de secuencia de al menos el 80 % con el complemento de la secuencia polinucleotídica diana.
19. Un oligonucleótido de acuerdo con la reivindicación 16, donde la secuencia complementaria a la diferencia de la secuencia nucleotídica del alelo dado en comparación con otro alelo del gen está localizada hacia el centro del oligonucleótido.
20. Un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones anteriores, inmovilizado sobre o dentro de un soporte.
21. Una serie de oligonucléotidos inmovilizados en localizaciones espaciadas sobre o dentro de un soporte, donde al menos uno de los oligonucléotidos es un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones 1 a
19.
22. Una serie de oligonucléotidos de acuerdo con la reivindicación 21, donde cada oligonucleótido es un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones 1 a 19.
23. Un método para investigar una secuencia polinucleotídica diana en una muestra que contiene secuencias polinucleotídicas, comprendiendo el método poner en contacto la muestra con un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones 1 a 20, que es capaz de hibridar con la secuencia polinucleotídica diana, y determinar si sucede hibridación.
24. Un método de acuerdo con la reivindicación 23, donde la detección de un aumento de la fluorescencia se usa para determinar si ha sucedido hibridación entre el oligonucleótido y la secuencia polinucleotídica diana.
25. Un método de acuerdo con la reivindicación 23 o la reivindicación 24, donde el método se realiza a una temperatura predeterminada próxima al, o en un intervalo de temperaturas que abarca el punto de fusión del híbrido
o de los híbridos formados entre la secuencia polinucleotídica diana y dicho oligonucleótido.
26. Un método de acuerdo con una cualquiera de las reivindicaciones 23 a 25, donde el oligonucleótido es completamente complementario a la secuencia polinucleotídica diana.
27. Un método de acuerdo con una cualquiera de las reivindicaciones 23 a 25, donde el oligonucleótido no es completamente complementario a la secuencia polinucleotídica diana.
28. Un método de acuerdo con la reivindicación 27, donde el oligonucleótido tiene una identidad de secuencia de al menos el 70 % con el complemento de la secuencia polinucleotídica diana.
29. Un método de acuerdo con la reivindicación 28, donde el oligonucleótido tiene una identidad de secuencia de al menos el 80 % con el complemento de la secuencia polinucleotídica diana.
30. Un método de acuerdo con una cualquiera de las reivindicaciones 23 a 28, donde la secuencia polinucleotídica diana investigada son uno o más alelos de un gen.
31. Un método de acuerdo con la reivindicación 30, donde el oligonucleótido es capaz de hibridar con más de un alelo del gen.
32. Un método de acuerdo con una cualquiera de las reivindicaciones 23 a 29, donde el método se usa para detectar, identificar o cuantificar la presencia o la cantidad de la secuencia polinucleotídica diana en la muestra.
33. Un método de acuerdo con una cualquiera de las reivindicaciones 23 a 30, donde la muestra contiene secuencias polinucleotídicas producidas por amplificación.
34. Un método de acuerdo con la reivindicación 33, donde las secuencias polinucleotídicas se producen mediante una cualquiera o más de reacción en cadena de la polimerasa (PCR) , PCR de transcripción inversa (RT-PCR) , reacción en cadena de la ligasa (LCR) , amplificación por desplazamiento de hebra (SDA) , amplificación mediada por transcripción (TMA) , amplificación por círculo rodante (RCA) o amplificación basada en secuencias de ácido nucleico
(NASBA) .
35. Un método de acuerdo con la reivindicación 33 ó 34, donde la amplificación de las secuencias polinucleotídicas en la muestra se realiza en presencia del oligonucleótido que es capaz de hibridar con la secuencia polinucleotídica diana.
36. Un método de acuerdo con la reivindicación 35, donde la observación de la fluorescencia se hace durante la amplificación de las secuencias polinucleotídicas en la muestra.
37. Un método de acuerdo con una cualquiera de las reivindicaciones 33 a 36, donde las secuencias polinucleotídicas de la muestra se producen por amplificación de una fuente biológica sin extracción previa del ácido nucleico.
38. Un método de acuerdo con la reivindicación 34, donde la PCR es una amplificación asimétrica.
39. Un método de acuerdo con una cualquiera de las reivindicaciones 25 a 38, donde el oligonucleótido que es capaz de hibridar con la secuencia polinucleotídica diana, la muestra que puede contener la secuencia polinucleotídica diana o ambos están inmovilizados sobre o dentro de un soporte.
40. Un método de acuerdo con una cualquiera de las reivindicaciones 25 a 39, donde la secuencia polinucleotídica diana se selecciona entre el grupo que consiste en el genoma humano incluyendo cualquier gen en el mismo o alelo del mismo, el genoma de ratón o cualquier gen en el mismo o alelo del mismo, y el genoma de cualquier agente infeccioso incluyendo cualquier gen en el mismo o alelo del mismo.
41. Un proceso para preparar un oligonucleótido de acuerdo con una cualquiera de las reivindicaciones 12 a 19, comprendiendo el proceso seleccionar una secuencia polinucleotídica diana y sintetizar un oligonucleótido que tiene una secuencia complementaria al polinucleótido diana y que tiene las propiedades expuestas en cualquiera de las reivindicaciones 1 a 20.
42. Un proceso de acuerdo con la reivindicación 41, donde la secuencia polinucleotídica diana se selecciona entre el grupo que consiste en el genoma humano incluyendo cualquier gen en el mismo o alelo del mismo, el genoma de ratón incluyendo cualquier gen en el mismo o alelo del mismo, y el genoma de cualquier agente infeccioso incluyendo cualquier gen en el mismo o alelo del mismo.
43. Un método para investigar una secuencia polinucleotídica diana en una muestra que contiene secuencias polinucleotídicas producidas por amplificación, comprendiendo el método poner en contacto la muestra con un oligonucleótido que es capaz de hibridar con la secuencia polinucleotídica diana y determinar si sucede hibridación, donde el oligonucleótido es un oligonucleótido monocatenario que no tiene una secuencia que contiene partes hechas de cuatro o más restos nucleotídicos consecutivos que son el complemento inverso de cada uno de los otros que permiten el apareamiento intramolecular de bases y que está formado de restos nucleotídicos, donde las bases de dos o más restos nucleotídicos internos están marcadas con un fluoróforo sin un inactivador asociado, donde al menos dos de los restos nucleotídicos marcados con un fluoróforo están separados por al menos dos restos nucleotídicos no marcados, y donde cada fluoróforo es el mismo fluoróforo.
Fluorescencia-d (F1) /dT
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]