METODO PARA EXPLORAR LA PRESENCIA DE UN DEFECTO GENETICO ASOCIADO CON TROMBOSIS VENOSA PROFUNDA.
Un método de exploración de un individuo para determinar un riesgo aumentando de trombosis venosa,
en particular de trombosis venosa profunda, que comprende detectar la presencia en el genoma del individuo de un marcador genético indicativo de un riesgo aumentado de trombosis venosa, en particular de trombosis venosa profunda, en el que el marcador genético es parte del haplotipo 2 del gen del fibrinógeno ? (FGG-H2) según indica la Figura 5A (SEQ ID NO:21) y en el que la presencia de FGG-H2 está asociada con la presencia de un conjunto de una, dos, tres o cuatro mutaciones en el material de ácido nucleico que codifica el fibrinógeno ?, estando seleccionadas las mutaciones entre el grupo que consiste en 129A/T (rs2066854), 7874G/A (rs2066861), 9615C/T (rs2066864) y 10034C/T (rs2066865)
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2006/003195.
Solicitante: LEIDEN UNIVERSITY MEDICAL CENTRE (LUMC) ACTING ON BEHALF OF THE ACADEMIC HOSPITAL LEIDEN.
Nacionalidad solicitante: Países Bajos.
Dirección: ALBINUSDREEF 2,2333 ZA LEIDEN.
Inventor/es: BERTINA, ROGIER MARIA, ROSENDAAL,FRITS,R, DE WILLIGE,SHIRLEY,UITTE, DE VISSER- VAN SOEST,MARIA,CATHARINA,HENRICA, VOS,HANS,LUUK.
Fecha de Publicación: .
Fecha Concesión Europea: 2 de Junio de 2010.
Clasificación Internacional de Patentes:
- C12Q1/68M6
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
Método para explorar la presencia de un defecto genético asociado con trombosis venosa profunda.
La presente invención se refiere a un método para explorar la presencia de un defecto genético asociado con las trombosis venosas, en particular la trombosis venosa profunda. La invención se refiere, además, a un kit de diagnóstico para usar en el método.
La trombosis venosa (VT) consiste en la obstrucción de la circulación por coágulos que se han formado localmente en las venas o que se han desprendido desde un trombo en otra parte (embolización). Los sitos habituales de formación de trombos son las venas superficiales y profundas de las piernas, pero también pueden ocurrir en venas del cerebro, la retina, el hígado y el mesenterio. Son complicaciones principales el síndrome post-trombótico y la muerte procedentes de embolismo pulmonar, que ocurren en 20-40% y 1-2% de los pacientes, respectivamente. En países desarrollados la incidencia anual de VT es 1/1000, aproximadamente.
La trombosis venosa es un enfermedad multicausal. Además de factores de riesgo adquiridos, bien conocidos, tales como inmovilización, cirugía reciente o traumas, el embarazo y el puerperio, y el uso actual de contraceptivos orales, existen también diversos factores de riesgo genéticos para la incidencia de VT, tales como el factor V Leiden y la mutación 20210A de la protrombina.
Es el objeto de la presente invención proporcionar un método de exploración que permite determinar la presencia de un factor de riesgo genético de la trombosis venosa, en particular, la trombosis venosa profunda, en un indivi- duo.
Este objeto ha sido resuelto mediante la identificación de un haplotipo del fibrinógeno ? (en el presente contexto definido como una serie de polimorfismos de un solo nucleótido (SNPs) que se heredan en conjunto) que está asociado con un nivel reducido de fibrinógeno ?' plasmático, una relación reducida de fibrinógeno ?'/fibrinógeno total (?'/?) y un riesgo incrementado de trombosis venosa profunda (DVT).
El fibrinógeno es un componente esencial del sistema hemostático, siendo el precursor de la fibrina, el producto final de la cascada de la coagulación. El fibrinógeno es convertido en fibrina a través de proteólisis limitada por trombina, que expone sitios de polimerización situados sobre los monómeros de fibrina. Estos monómeros se asocian espontáneamente formando fibrina insoluble. El factor XIII activado forma enlaces covalentes entre monómeros de fibrina adyacentes. Las reticulaciones realizadas refuerzan el coágulo de fibrina y aumentan su resistencia a la degradación por el sistema fibrinolítico.
El fibrinógeno, representado en la Figura 1 por Fiona Green (
El gen del FGG contiene 10 exones y está orientado en tándem con el gen del FGA, que contiene 6 exones. Ellos están transcritos en sentido opuesto al gen del FGB, que está colocado aguas abajo del gen del FGA y que contiene 8 exones.
Un corte y empalme alternativo puede tener lugar en los genes del FGA y el FGG. La cadena Aa predominante del fibrinógeno que circula contiene 610 restos de aminoácidos, mientras que la cadena Aa alternativa contiene 846 restos de aminoácidos. La cadena Bß consiste en 461 aminoácidos. La forma más abundante de la cadena ?, ?A, consiste en 411 restos de aminoácidos. La cadena ? variante (?B) contiene 427 restos de aminoácidos.
Se conocen anomalías del fibrinógeno que afectan al riesgo de trombosis venosa profunda (DVT). Koster et al., (Thromb. Haemost. 71: 719-722 (1994)) han descrito que niveles elevados de fibrinógeno plasmático (> 5 g/l) aumentan el riesgo de DVT. El mecanismo de este efecto no es conocido. La concentración de fibrinógeno ejerce un efecto profundo sobre la estructura del coágulo de fibrina en experimentos in vitro. La velocidad de liberación del fibrinopéptido A aumenta con los niveles crecientes de fibrinógeno, y este hecho está asociado con la formación de un retículo de fibrina más resistente a la lisis, y más denso y apretado (Blombäck, Thromb. Res. 75. 327-328 (1994); Siebenlist and Mosesson, J. Biol. Chem. 68:315-320 (1994)).
Otro mecanismo mediante el cual niveles elevados de fibrinógeno pueden contribuir al riesgo de trombosis ocurre mediante el aumento de la viscosidad de la sangre. Además, variantes genéticas de fibrinógeno (disfibrinogenemías) han sido descubiertas en pacientes con trombosis y un tiempo de trombina prolongado (revisado por Mosesson (Semin. Thromb. Hemost. 25:311-319 (1999)); Hanss and Biot (Ann. N.Y. Acad. Sci., 936: 89-90 (2001))). La mayoría de estos pacientes poseen una mutación en el gen del FGA o el FGG, aun cuando la correlación precisa existente entre el medio de transporte de estas mutaciones y la trombosis venosa está mal documentado (Haverkate et al., Thromb. Haemost. 73:151-161 (1995)).
Los presentes inventores, por tanto, formularon la hipótesis de que pudieran existir variaciones relativamente comunes de los genes del fibrinógeno que influyeran en el riesgo de trombosis, en particular de la trombosis venosa profunda. Estas variaciones pueden afectar a los niveles de fibrinógeno, a la formación de la estructura reticular de la fibrina o a la sensibilidad del coágulo de fibrina para el sistema fibrinolítico.
En la investigación que condujo a la invención, los inventores tipificaron 15 polimorfismos de un solo nucleótido que marcaban haplotipos (htSNPs, que son SNPs específicos de un haplotipo) en los tres genes de la agrupación del fibrinógeno en un estudio de caso-testigo, basado en una población grande, sobre factores de riesgo de trombosis venosa, el Estudio de Trombofilia de Leiden (Leiden Thrombophilia Study) (LETS). Además, fueron medidos en todos los sujetos los niveles combinados de las isoformas de fibrinógeno ?A/?' y ?'/?' que contienen una variante alternativa de corte y empalme de la cadena del fibrinógeno ? (?').
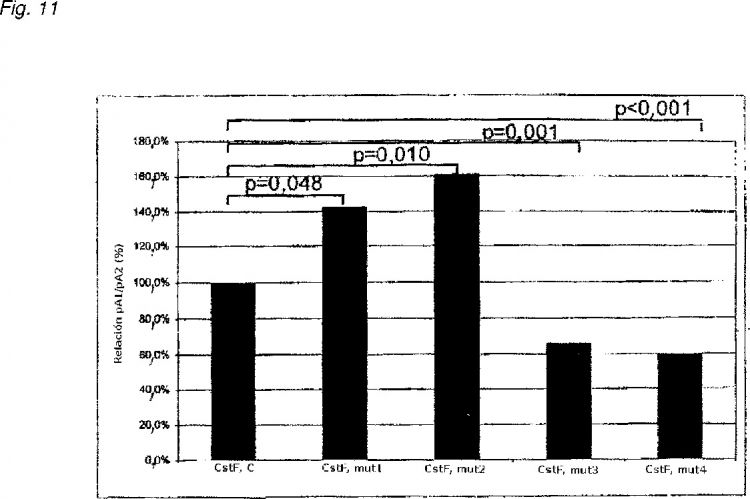
Se descubrió que los homozigotos individuales de FGB-H2, FGA-H2 ó FGG-H2, tenían todos un riesgo incrementado de trombosis venosa (FGB-H2: OR = 1,9, IC95% (intervalo de confianza 95%): 1,1-3,4; FGA-H2: OR = 2,0, IC95%: 1,3-3,2; FGG-H2: OR=2,4, IC95%: 1,5-3,9). Dado que los tres genes de fibrinógeno están situados sobre una única extensión del DNA de 50 kb, se usó regresión logística múltiple para ajustar el desequilibrio de enlace existente entre los genes. Después del ajuste, el riesgo elevado solamente permanecía para homozigotos individuales del FGG-H2. Ninguno de los haplotipos del fibrinógeno estaba asociado con los niveles de fibrinógeno total medidos con el método Clauss. El FGG-H2 estaba asociado también con niveles reducidos de fibrinógeno ?' y con una relación reducida de fibrinógeno ?'/?. La regresión logística puso de manifiesto que ambos, niveles reducidos de fibrinógeno ?' y niveles aumentados de fibrinógeno total estaban asociados con un riesgo aumentado tres veces de DVT, incluso después de ajuste del FGG-H2.
Sobre la base de este descubrimiento, se llegó a la conclusión de que el FGG-H2 está asociado con niveles reducidos de fibrinógeno ?' y de que en análisis multivariados los niveles reducidos de fibrinógeno ?' están asociados con un riesgo aumentado tres veces de desarrollo de DVT, lo que demuestra que el haplotipo FGG-H2 actúa sobre el riesgo trombótico a través del fenotipo de un nivel reducido de fibrinógeno ?'.
Debido a que la concentración plasmática de fibrinógeno ?' y de fibrinógeno total influyen, ambas, en el riesgo trombótico y puesto que el nivel de fibrinógeno ?' depende siempre del nivel de fibrinógeno total, los inventores analizaron también el efecto de la relación fibrinógeno ?'/fibrinógeno total (relación ?'/?) sobre el riesgo de trombosis venosa. Encontraron que individuos...
Reivindicaciones:
1. Un método de exploración de un individuo para determinar un riesgo aumentando de trombosis venosa, en particular de trombosis venosa profunda, que comprende detectar la presencia en el genoma del individuo de un marcador genético indicativo de un riesgo aumentado de trombosis venosa, en particular de trombosis venosa profunda, en el que el marcador genético es parte del haplotipo 2 del gen del fibrinógeno ? (FGG-H2) según indica la Figura 5A (SEQ ID NO:21) y en el que la presencia de FGG-H2 está asociada con la presencia de un conjunto de una, dos, tres o cuatro mutaciones en el material de ácido nucleico que codifica el fibrinógeno ?, estando seleccionadas las mutaciones entre el grupo que consiste en 129A/T (rs2066854), 7874G/A (rs2066861), 9615C/T (rs2066864) y 10034C/T (rs2066865).
2. Un método según la reivindicación 1, en el que el conjunto de mutaciones está seleccionado entre los conjuntos indicados en la tabla que sigue:

3. Un método según la reivindicación 2, en el que el conjunto de mutaciones está seleccionado entre los conjuntos 5, 6, 7, 8, 11, 12, 14 y 15 indicados en la tabla de la reivindicación 2.
4. Un método según una cualquiera de las reivindicaciones 1-3, en el que la presencia de FGG-H2 está asociada con la presencia de la mutación 10034C/T (rs2066865) en el material de ácido nucleico que codifica fibrinógeno ?.
5. Un método según una cualquiera de las reivindicaciones 1-4, en el que dicho marcador es detectado llevando a cabo una reacción de amplificación del ácido nucleico diana de una extensión de un material de ácido nucleico que comprende dicho conjunto de mutaciones, y analizando el ácido nucleico diana amplificado para determinar la presencia del conjunto de mutaciones.
6. Un método según la reivindicación 4, en el que la reacción de amplificación está seleccionada entre NASBA, PCR, LCR, RCR, 3SR y TMA.
7. Un método según cualquiera de las reivindicaciones 1-6, en el que cada ácido nucleico amplificado forma un complejo detectable con un oligonucleótido complementario.
8. Un método según la reivindicación 7, en el que el oligonucleótido complementario está inmovilizado sobre una fase sólida, preferentemente una partícula magnética, y cada uno de los complejos detectables puede reaccionar con un reactante marcado.
9. Un método según la reivindicación 7, en el que el oligonucleótido complementario está inmovilizado sobre una fase sólida y cada uno de los complejos detectables puede reaccionar con reactantes marcados de modo diferente.
10. Un método según cualquiera de las reivindicaciones 5-6 en el que los ácidos nucleicos amplificados, diferentes, forman un complejo detectable con un oligonucleótido complementario diferente.
11. Un método según la reivindicación 10, en el que dichos oligonucleótidos complementarios diferentes están inmovilizados, cada uno, en un lugar diferente sobre una fase sólida.
12. Un kit para llevar a cabo el método según una cualquiera de las reivindicaciones 1-11, para explorar a un individuo y determinar un riesgo aumentado de trombosis venosa, en particular de trombosis venosa profunda, cuyo kit comprende, por lo menos, un par de cebadores de amplificación que reconocen y se hibridan a extensiones de ácido nucleico que rodean al menos una extensión de un ácido nucleico diana que comprende, al menos, una mutación, siendo la mutación un marcador genético de haplotipo 2 del gen del fibrinógeno ? (FGG-H2) estando seleccionada la mutación entre el grupo que consiste en 129A/T, 7874G/A, 9615C/T y 10034C/T, y una sonda para detectar el ácido nucleico diana amplificado para determinar la presencia de dicha mutación.
13. Un kit según la reivindicación 12, que comprende un par de cebadores de amplificación que reconocen y se hibridan a extensiones de ácido nucleico que rodean una extensión de un ácido nucleico diana que ha de ser amplificado, cuyo ácido nucleico diana corresponde a parte al menos del gen del fibrinógeno ? que comprende al menos una mutación, siendo la mutación un marcador genético del haplotipo 2 del gen del fibrinógeno ? (FGG-H2), y al menos una sonda para detectar el ácido nucleico diana amplificado para determinar la presencia de dicha mutación.
14. Un kit según cualquiera de las reivindicaciones 12 ó 13, que comprende un par de cebadores de amplificación que reconocen y se hibridan a extensiones de ácido nucleico que rodean una extensión de un ácido nucleico diana que ha de ser amplificado, cuyo ácido nucleico diana corresponde a parte al menos del gen del fibrinógeno ? que comprende al menos una mutación seleccionada entre el grupo que consiste en 129A/T, 7874G/A, 9615C/T y 10034C/T, y al menos una sonda para detectar cada mutación de interés en el ácido nucleico diana amplificado.
15. Un kit según cualquiera de las reivindicaciones 12-14, que comprende:

o sus secuencias complementarias,

o sus secuencias complementarias.
16. Un kit según cualquiera de las reivindicaciones 12-15, para detectar la mutación 129A/T que comprende:
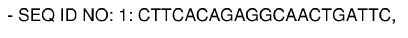
o su secuencia complementaria,


o sus secuencias complementarias.
17. Un kit según la reivindicación 16, en el que el primer oligonucleótido tiene una longitud de 10-26 nucleótidos, y el segundo oligonucleótido tiene una longitud de 10-26 nucleótidos y comprende la SEQ ID NO:10: GTGTCAAC CATGTTCATAGGC, o al menos un fragmento de 10 de sus nucleótidos.
18. Un kit según cualquiera de las reivindicaciones 12-15, para detectar la mutación 7874G/A, que comprende:

o sus secuencias complementarias,
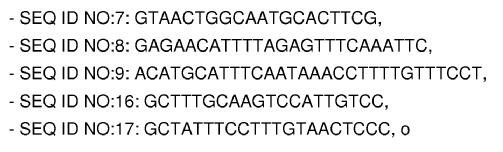

o sus secuencias complementarias.
19. Un kit según la reivindicación 18, en el que el primer oligonucleótido tiene una longitud de 10-26 nucleótidos y comprende: SEQ ID NO:6: TTCCAAGGAAGCATCCTACG o al menos un fragmento de 10 de sus nucleótidos, y el segundo oligonucleótido tiene una longitud de 10-26 nucleótidos y comprende la SEQ ID NO:16: GCTTTG CAAGTCCATTGTCC o al menos un fragmento de 10 de sus nucleótidos.
20. Un kit según cualquiera de las reivindicaciones 12-15, para detectar la mutación 9615C/T, que comprende.

o sus secuencias complementarias.

o sus secuencias complementarias.
21. Un kit según la reivindicación 20, en el que el primer oligonucleótido tiene una longitud de 10-26 nucleótidos y comprende la SEQ ID NO:7: GTAACTGGCAATGCACTTCG o al menos un fragmento de 10 de sus nucleótidos, y el segundo oligonucleótido tiene una longitud de 10-26 nucleótidos y comprende la SEQ ID NO:16: GCTTTGCAAGTC CATTGTCC o al menos un fragmento de 10 de sus nucleótidos.
22. Un kit según cualquiera de las reivindicaciones 12-15, para detectar la mutación 10034C/T, que comprende:

o sus secuencias complementarias.

o sus secuencias complementarias.
23. Un kit según la reivindicación 22, en el que el primer oligonucleótido tiene una longitud de 10-26 nucleótidos y comprende la SEQ ID NO: 8: GAGAACATTTTAGAGTTTCAAATTC ó la SEQ ID NO: 9: ACATGCATTTCAA TAAACCTTTTGTTTCCT, o al menos un fragmento de 10 de sus nucleótidos, y el segundo oligonucleótido tiene una longitud de 10-26 nucleótidos.
24. Un kit según cualquiera de las reivindicaciones 13 ó 14, en el que hay una sonda para detectar cada mutación (129A/T, 7874G/A, 9615C/T ó 10034C/T) de interés en el ácido nucleico diana amplificado, comprendiendo dicha sonda una secuencia de ácido nucleico que corresponde al ácido nucleico analito que ha sido mutado para discriminarle de un ácido nucleico analito sin mutar.
25. Un kit según la reivindicación 24, en el que la sonda es un indicador molecular.
26. Un kit según cualquiera de las reivindicaciones 24 ó 25, en el que la sonda para detectar el haplotipo FGG-H2 por unión al alelo 10034T (mutación 10034C/T) es la SEQ ID NO: 19: ATGGTCAATAAAGATACCA o contiene un oligonucleótido que tiene una longitud de 10-50 nucleótidos, preferentemente 10-26, y que comprende la SEQ ID NO: 19 ATGGTCAATAAAGATACCA o al menos un fragmento de 10 de sus nucleótidos.
27. Un kit según cualquiera de las reivindicaciones 24-26, que contiene, además, una sonda para detectar el haplotipo FGG-H2 por unión al alelo 10034C, que es la SEQ ID NO:20: TTTTAATGGTCAATAAAGGTACCA o contiene un oligonucleótido que tiene una longitud de 10-50 nucleótidos, preferentemente 10-26, y que comprende la SEQ ID NO:20: TTTTAATGGTCAATAAAGGTACCA o al menos un fragmento de 10 de sus nucleótidos.
28. Un kit según cualquiera de las reivindicaciones 12-27, en el que el kit contiene, además, reactivos de amplificación adecuados.
29. Un método para explorar a un individuo y determinar un riesgo aumentado de trombosis venosa, en especial trombosis de venosa profunda, en el que el método según cualquiera de las reivindicaciones 1-11 determina, además, la relación de fibrinógeno ?'/fibrinógeno total (relación ?'/?), y en el que la relación ?'/? debe ser inferior a 0,69 para tener un riesgo aumentado de trombosis venosa profunda.
Patentes similares o relacionadas:
MODULADORES DEL PESO CORPORAL, ÁCIDOS NUCLEICOS Y PROTEÍNAS CORRESPONDIENTES, Y USOS DIAGNÓSTICOS Y TERAPÉUTICOS DE LOS MISMOS, del 17 de Febrero de 2012, de THE ROCKEFELLER UNIVERSITY: LA PRESENTE INVENCION SE REFIERE GENERALMENTE AL CONTROL DEL PESO CORPORAL DE ANIMALES INCLUIDOS MAMIFEROS Y HUMANOS, Y EN CONCRETO A MATERIALES […]
POLIMORFISMOS DE VEGF Y TERAPIA ANTI-ANGIOGÉNESIS, del 17 de Enero de 2012, de GENENTECH, INC.: Método para predecir si un paciente se encuentra con un mayor riesgo de hipertensión asociada con el tratamiento con un antagonista de VEGF, que comprende cribar […]
MÉTODO DE DIAGNÓSTICO Y DE SEGUIMIENTO DE UNA VAGINOSIS BACTERIANA MEDIANTE CUANTIFICACIÓN MOLECULAR, del 28 de Diciembre de 2011, de UNIVERSITE DE LA MEDITERRANEE, AIX-MARSEILLE II: Método de diagnóstico y de seguimiento in vitro del estado de la flora bacteriana vaginal frente a la presencia de una vaginosis bacteriana […]
DETECCIÓN DE GÉRMENES ASOCIADOS A PERIODONTITIS, del 9 de Diciembre de 2011, de GREINER BIO-ONE GMBH: Procedimiento para la detección y/o para la determinación de bacterias asociadas a periodontitis de una muestra biológica, que comprende las etapas i) obtención […]
CONFORMACIÓN DE ADN (ESTRUCTURAS DE BUCLE) EN EXPRESIÓN GÉNICA NORMAL Y ANORMAL, del 22 de Noviembre de 2011, de ISIS INNOVATION LIMITED: Procedimiento de diagnóstico de un cancer por detección de expresión genica anormal en un individuo que comprende determinar en una muestra del individuo […]
USO DE POLIMORFISMOS GENÉTICOS QUE SE ASOCIAN CON LA EFICACIA DEL TRATAMIENTO DE UNA ENFERMEDAD INFLAMATORIA, del 4 de Noviembre de 2011, de NOVARTIS AG NOVARTIS PHARMA GMBH: Uso de pimecrolimus en la fabricación de un medicamento para el tratamiento de dermatitis atópica pediátrica en una población seleccionada de pacientes, en donde la población […]
MÉTODOS PARA EVALUAR ESTADOS PATOLÓGICOS USANDO ARN EXTRACELULAR, del 2 de Noviembre de 2011, de ONCOMEDX INC: Método para detectar, diagnosticar o monitorizar una lesión de un órgano en un ser humano, comprendiendo el método la etapa de detectar cualitativa o cuantitativamente […]
MÉTODOS DE TRATAMIENTO DE PSICOSIS Y ESQUIZOFRENIA BASADOS EN POLIMORFISMOS DEL GEN DEL CNTF, del 23 de Agosto de 2011, de NOVARTIS AG: Un método para determinar la capacidad de respuesta de un individuo con un trastorno psicótico al tratamiento con Iloperidona, que comprende; […]