Ácidos ribonucleicos de doble cadena con estructura fisicoquímica robusta y actividad biológica muy específica.
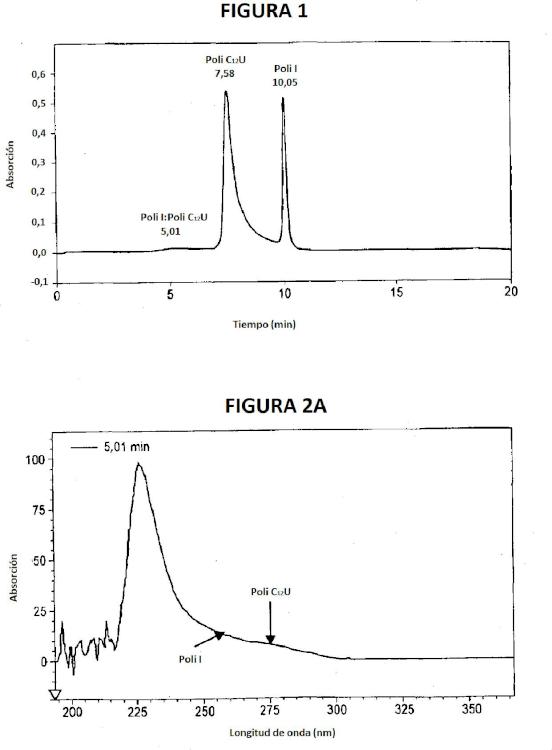
Un ácido ribonucleico de doble cadena (ARNdc) aislado que comprende una cadena sencilla compuesta de poli(ácido ribocitidílico4-29uridílico) y una cadena opuesta compuesta de poli(ácido riboinosínico) de tal manera que las dos cadenas no forman un dúplex en la posición de la base uracilo,
de modo que dichas cadenas están parcialmente hibridadas, caracterizado por que:
el ARNdc tiene un peso molecular de aproximadamente 250 kDa a aproximadamente 320 kDa y es resistente a la desnaturalización en condiciones que son capaces de separar las cadenas de poli(ácido riboinosínico) y poli(ácido ribocitidílico) hibridadas.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2009/005797.
Solicitante: Hemispherx Biopharma, Inc.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: One Penn Center 1617 Jfk Boulevard Philadelphia, PA 19103 ESTADOS UNIDOS DE AMERICA.
Inventor/es: CARTER, WILLIAM, A., STRAYER,DAVID.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- A61K31/713 NECESIDADES CORRIENTES DE LA VIDA. › A61 CIENCIAS MEDICAS O VETERINARIAS; HIGIENE. › A61K PREPARACIONES DE USO MEDICO, DENTAL O PARA EL ASEO (dispositivos o métodos especialmente concebidos para conferir a los productos farmacéuticos una forma física o de administración particular A61J 3/00; aspectos químicos o utilización de substancias químicas para, la desodorización del aire, la desinfección o la esterilización, vendas, apósitos, almohadillas absorbentes o de los artículos para su realización A61L; composiciones a base de jabón C11D). › A61K 31/00 Preparaciones medicinales que contienen ingredientes orgánicos activos. › Acidos nucleicos u oligonucleótidos con estructura en doble hélice.
- C12N15/117 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Acidos nucleicos que tienen propiedades inmunomoduladoras, p.ej. que contienen motivos CpG.
PDF original: ES-2553787_T3.pdf





Patentes similares o relacionadas:
 Compuestos que modulan la actividad de señalización de las interleucinas 17 y 23, del 29 de Julio de 2020, de Sarepta Therapeutics, Inc: Un método ex vivo de modulación de la actividad de señalización de IL-17 en una célula, que comprende poner en contacto la célula con un oligómero antisentido de morfolino […]
Compuestos que modulan la actividad de señalización de las interleucinas 17 y 23, del 29 de Julio de 2020, de Sarepta Therapeutics, Inc: Un método ex vivo de modulación de la actividad de señalización de IL-17 en una célula, que comprende poner en contacto la célula con un oligómero antisentido de morfolino […]
Método para producir inmunoconjugados de anticuerpo-SN-38 con un enlazador CL2A, del 22 de Julio de 2020, de IMMUNOMEDICS, INC.: Un método para producir un compuesto, CL2A-SN-38, que presenta la estructura, **(Ver fórmula)** que comprende realizar un esquema de reacción como el que se muestra: **(Ver […]
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Terapia mediada por ARN interferente para enfermedades neurodegenerativas, del 24 de Junio de 2020, de Università Degli Studi Di Trento: Agente terapéutico para su uso en el tratamiento de enfermedades neurodegenerativas asociadas con anomalías de la proteína tau codificada […]
Compuestos oligonucleotídicos dirigidos a ARNm de huntingtina, del 3 de Junio de 2020, de UNIVERSITY OF MASSACHUSETTS: Una molécula de ARN que tiene una longitud entre 15 y 35 bases, que comprende una región de complementariedad que es completamente complementaria a 5' CAGUAAAGAGAUUAA 3' […]
Agente de direccionamiento para células cancerosas o fibroblastos asociados con cáncer, del 29 de Abril de 2020, de NITTO DENKO CORPORATION: Composición para su uso en el tratamiento de cáncer en el que están implicados fibroblastos asociados con cáncer, caracterizada porque la composición comprende un portador […]
Lípido catiónico, del 29 de Abril de 2020, de TAKEDA PHARMACEUTICAL COMPANY LIMITED: Un compuesto representado por la fórmula: **(Ver fórmula)** donde, W denota la fórmula -NR1R2 o la fórmula -N+R3R4R 5(Z-); R1 y R2 denotan, cada uno de forma independiente, […]
Oligonucleótidos TGF-beta modificados, del 29 de Abril de 2020, de ISARNA Therapeutics GmbH: Oligonucleótido que consiste en CAAAGTATTTGGTCTCC (SEQ ID NO. 46), en el que los nucleótidos en negrita son oligonucleótidos modificados con LNA, complementarios […]