Procedimiento de determinación del genotipo relacionado con la paraqueratosis nasal hereditaria (PQNH) y ácidos nucleicos utilizables en dicho procedimiento.
Un procedimiento in vitro para determinar el genotipo relacionado con la paraqueratosis nasal hereditaria (PQNH) en un perro que comprende la determinación de la presencia o ausencia de una variación genética en la secuencia génica SUV39H2 en una muestra biológica de dicho perro,
en el que la presencia de dicha variación genética indica que dicho perro padece o padecerá dicho trastorno o está en riesgo de trasmitir dicho trastorno a su progenie, en el que dicha variación genética comprende un reemplazo de A por C en la posición 501 en la SEC ID Nº:2 o en una secuencia al menos 80 % idéntica a dicha secuencia o dicha variación genética es una variación de un gen que codifica el polipéptido de SEC ID Nº: 1 que comprende un reemplazo de asparagina por lisina en la posición 324.
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E13167812.
Solicitante: University of Bern.
Inventor/es: LEEB,TOSSO.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2547832_T3.pdf





Fragmento de la descripción:
Procedimiento de determinación del genotipo relacionado con la paraqueratosis nasal hereditaria (PQNH) y ácidos nucleicos utilizables en dicho procedimiento
Campo de la invención
La presente invención se refiere a un procedimiento de diagnóstico in vitro de un genotipo relacionado con la paraqueratosis nasal hereditaria (PQNH) en un perro y a materiales utilizables en dicho procedimiento. Particularmente la presente mención se refiere a la variación genética relacionada con la PQNH en Labradores Retriever.
Antecedentes
La paraqueratosis nasal hereditaria (PQNH) es una genodermatosis de los Labradores Retriever con una herencia monogénica autosómica recesiva (Pagé y col. 2003, Peters et al. 2003). Los perros afectados de PQNH desarrollan, a una edad temprana, costras y fisuras en el plano nasal, aunque son perros sanos. Los cambios histopatológicos consisten en una hiperqueratosis paraqueratósica y en una acumulación de líquido proteico ("lagos de suero") en el interior del estrato córneo y en el estrato espinoso superficial. Actualmente la PQNH no puede curarse, pero los síntomas pueden aliviarse con terapia sintomática.
Debido a la herencia monogénica autosómica recesiva, es muy importante disponer de un procedimiento que determine el genotipo de un perro. Los perros heterocigotos no están afectados y no pueden diferenciarse fenotípicamente de los perros homocigotos libres. Sin embargo, los perros heterocigotos portan el alelo mutante ("portadores") y pueden transmitir esta enfermedad a su descendencia.
Actualmente los criadores pueden identificar a los perros portadores únicamente basándose en la Información del fenotipo obtenida de generaciones anteriores y eliminar a los portadores identificados de los programas de cría, reduciendo de este modo la frecuencia de la enfermedad genética en una raza. Por tanto, hay una gran necesidad en la cría de Labradores Retriever de un ensayo genético que permita la identificación de portadores también sanos de PQNH. Por lo tanto, es muy valioso un procedimiento de ensayo genético que pueda diferenciar los tres genotipos "libre" (=tipo silvestre homocigoto), "portador" (=heterocigoto"), y "afectado" (=mutante homocigoto) para la cría de perros, así como para medicina veterinaria para confirmar el diagnóstico de casos sospechosos.
Sumario de la invención
Un objeto de la presente invención es proporcionar medios para Identificar perros de cría, particularmente Labradores Retriever, que tengan un riego de transmitir a su descendencia el genotipo de PQNH.
Éstos y otros objetos se consiguen a través de la presente Invención como se describe y reivindica más adelante.
El primer aspecto de la invención es un procedimiento in vitro para determinar en un perro el genotipo relacionado con la paraqueratosis nasal hereditaria (PQNH). De acuerdo con la Invención el procedimiento comprende determinar la presencia o ausencia de una variación genética en la secuencia génica SUV39H2 en una muestra biológica de dicho perro, en el que la presencia de dicha variación genética indica que dicho perro padece o padecerá dicho trastorno o que está en riesgo de transmitir dicho trastorno a su progenie, en el que dicha variación genética comprende un reemplazo de A porC en la posición 501 en la SEC ID N°:2 o en una secuencia al menos 80 % idéntica a dicha secuencia o dicha variación genética es una variación de un gen que codifica el polipéptido de SEC ID N°: 1 que comprende un reemplazo de asparaglna por Usina en la posición 324.
El segundo aspecto de la invención es un polinucleótldo. De acuerdo con la invención la secuencia de dicho polinucleótido se selecciona en el grupo que consiste en:
a) una secuencia que tiene la SEC ID N°: 2, en la que dicho polinucleótido comprende el reemplazo del nucleótido A por C en la posición 501 (c.972T>G; CanFam 3 ensamblaje g.CFA2:21,731,842A>C); y
b) una secuencia que es al menos 80% idéntica a la secuencia SEC ID N°: 2, en la que dicho polinucleótido comprende el reemplazo del nucleótido A por C en la posición 501;
c) una secuencia que codifica el polipéptido que tiene la SEC ID N°: 1 que comprende un reemplazo de asparagina por lisina en la posición 324; y
d) una secuencia complementaria a cualquiera de las secuencias de los apartados (a) a (c).
El tercer aspecto de la invención es un uso de la variación genética en el gen SUV39H2 en un perro como un marcador para identificar a un perro que porta la mutación heterocigota u homoclgota relacionada con la paraqueratosis nasal, en el que dicha variación genética comprende un reemplazo de A por C en la posición 501 en la SEC ID N°: 2 con en una secuencia que es al menos 80% idéntica a la secuencia SEC ID N°: 2.
El cuarto aspecto de la invención es un uso de un oligonucleótido que tiene la secuencia SEC ID N°: 2 o una secuencia al menos 80% idéntica a la secuencia SEC ID N° 2 en la que el nucleótido A en la posición 501 del oligonucleótido se ha reemplazado por C o dicho oligonucleótido que codifica el polipéptido de SEC ID N°: 1 comprende un reemplazo de asparagina por lisina en la posición 324 en el diagnóstico del genotipo relacionado con la PQNH.
El quinto aspecto de la invención es un procedimiento in vitro para determinar la paraqueratosis nasal hereditaria (PQNH) en un perro. De acuerdo con la invención dicho procedimiento comprende determinar la presencia o la ausencia de la mutación N324K en un polipéptido que tiene la SEC ID N°: 1 en una muestra biológica de dicho perro, en la el que la presencia de dicha mutación indica que dicho perro padece o padecerá dicho trastorno o que está en riesgo de trasmitir dicho trastorno a su progenie.
El sexto aspecto de la invención es un anticuerpo. De acuerdo con la invención dicho anticuerpo se caracteriza por reconocer específicamente a un epítopo que comprende la mutación N324K de SEC ID N°: 1 de un perro.
Breve descripción de los dibujos
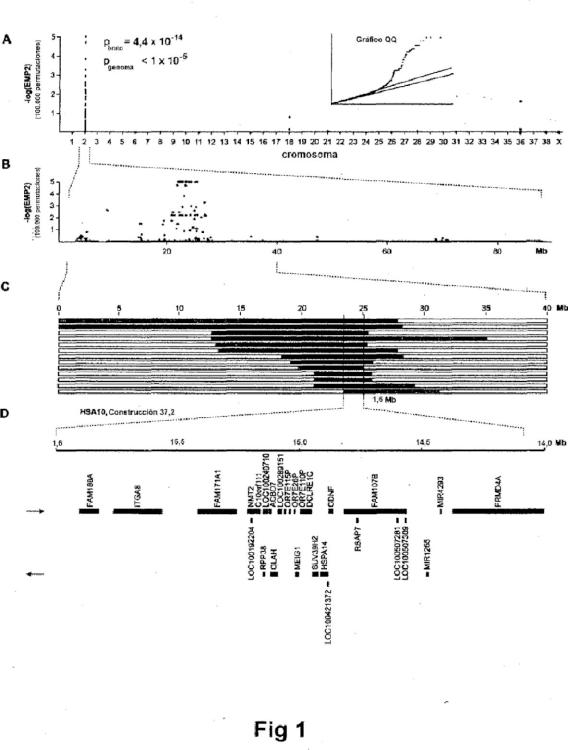
Figura 1. Análisis de asociación de todo el genoma (las coordenadas de la Figura se refieren al conjunto CanFam 2.1 anterior y no son idénticas a las coordenadas de CanFam 3.1 en el texto). (A) Se identificó una sola señal muy significativa en el cromosoma 2. (B) La señal de asociación se localizó en el primer tercio del cromosoma 2. (C) Las barras negras indican segmentos homocigotos en los 14 perros afectados. El mapeo de homocigosidad define un intervalo exacto para la localización de la mutación causante. (D) Contenido génico del intervalo crítico deducido de la anotación de genes humanos correspondiente.
Figura 2. Secuencia contexto de la mutación causante de la PQNH. (A) En la mitad superior, se muestra la secuencia en la misma orientación que la del gen SUV39H2. (B) Este gen está en orientación negativa en el cromosoma 2. Por lo tanto, en la mitad inferior de la figura la secuencia flanqueante también se muestra en la orientación directa, que corresponde a las coordenadas del conjunto del genoma CanFam 3.
Figura 3. Presentación esquemática de la estrategia de resecuenciación de un Labrador Retriever afectado. Se preparó una biblioteca de un fragmento de 200 pb y se recogieron 3 carriles de secuencias de 2x100 pb en un instrumento ilumina HiSec2000.; En total se recopiló 1 billón de lecturas o datos originales de 100 Gb correspondientes a una cobertura de genoma de 38,5. El diagrama de flujo muestra la estrategia de análisis de datos e ilustra cómo se usaron las lecturas de secuencias experimentales para determinar las vahantes entre el genoma de Labrador Retriever afectado y el genoma de referencia del Bóxer.
Figura 4. Gráfico del gen SUV39H2 que muestra la secuenclaclón de "Control" (sano), "portador" y "afectado con PQNH"
Figura 5. Conservación de secuencia de la proteína SUV39H2. La figura muestra un alineamiento de aminoácidos en la región de la variante N324K crítica, que forma parte del dominio SET catalíticamente activo. La secuencia de las proteínas SUV39H2 ortólogas de todos los vertebrados es idéntica en la reglón del resto de N-324 (parte superior del alineamiento). Incluso metlltransferasas H3K9 parálogas distantemente relacionadas comparten un resto de asparagina en esta posición (parte Inferior del alineamiento). La alta conservación de la secuencia confirma la hipótesis de que una mutación de asparagina en la posición 324 afectará a la función de la proteína SUV39H2.
Figura 6. Muestra 27 genes humanos anotados localizados en la región correspondiente a 15 genes anotados en el Labrador Retriever que se analiza en este estudio.
Descripción detallada
La Invención se basa en el sorprendente descubrimiento de que la variación genética... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento in vitro para determinar el genotipo relacionado con la paraqueratosis nasal hereditaria (PQNH) en un perro que comprende la determinación de la presencia o ausencia de una variación genética en la secuencia génica SUV39H2 en una muestra biológica de dicho perro, en el que la presencia de dicha variación genética indica que dicho perro padece o padecerá dicho trastorno o está en riesgo de trasmitir dicho trastorno a su progenie, en el que dicha variación genética comprende un reemplazo de A por C en la posición 501 en la SEC ID N°:2 o en una secuencia al menos 80 % idéntica a dicha secuencia o dicha variación genética es una variación de un gen que codifica el polipéptido de SEC ID N°: 1 que comprende un reemplazo de asparaglna por Usina en la posición 324.
2. El procedimiento de acuerdo con la reivindicación 1, en el que dicho perro es un Labrador Retriever.
3. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 o 2, en el que
- la presencia de dicha variación genética en ambos alelos de dicho gen SUV39H2 indica que dicho perro padece o padecerá PQNH; y
- la presencia de dicha variación genética en uno de dos alelos de dicho gen SUV39H2 indica que dicho perro es un portador sano de PQNH;
- la ausencia de dicha variación genética indica que dicho perro es un no portador sano de PQNH.
4. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 3, en el que la variación se detecta, por ejemplo, por PCR, PCR en tiempo real, análisis del punto de fusión de ADN bicatenario, espectroscopia de masas, secuenciación directa de ADN, polimorfismo de longitud de fragmentos de restricción (RFLP), polimorfismo de conformación de moléculas monocatenarias (SSCP), cromatografía líquida de alto rendimiento (HPLC), extensión de cebador de una sola base, u otros procedimientos relacionados.
5. Un polinucleótido que comprende una secuencia seleccionada del grupo que consiste en:
a) una secuencia que tiene la SEC ID N°: 2, comprendiendo dicho polinucleótido un reemplazo del nucleótido A por C en la posición 501; y
b) una secuencia que es al menos 80% idéntica a la secuencia SEC ID N°: 2, comprendiendo dicho polinucleótido un reemplazo del nucleótido A por C en la posición 501; y
c) una secuencia que codifica el polipéptido que tiene la SEC ID N°: 1, comprendiendo un reemplazo de asparagina por lisina en la posición 324; y
d) una secuencia complementaria a cualquiera de las secuencias de los puntos (a) a (c).
6. El uso de variación genética en el gen SUV39H2 en un perro como un marcador para la identificación de un perro que porta una mutación heterocigota u homocigota relacionada con la paraqueratosis nasal, en el que dicha variación genética comprende un reemplazo de A por C en la posición 501 en la SEC ID N°: 2 o en una secuencia que es al menos 80% idéntica a la secuencia SEC ID N°: 2.
7. El uso de un oligonucleótido que tiene la secuencia SEC ID N°: 2 o una secuencia que es al menos 80% idéntica a dicha secuencia, en el que el nucleótido A en la posición 501 del oligonucleótido ha sido reemplazado por C, o dicho oligonucleótido que codifica el polipéptido de SEC ID N°: 1 comprende un reemplazo de asparagina por lisina en la posición 324 para el diagnóstico del genotipo relacionado con la PQNH.
8. Un procedimiento in vitro de determinación de la paraqueratosis nasal hereditaria (PQNH) en un perro, que comprende la determinación de la presencia o ausencia de una mutación N324K en un polipéptido que tiene la SEC ID N°: 1 en una muestra biológica de dicho perro, en el que la presencia de la mutación indica que dicho perro padece o padecerá dicho trastorno o que está en riesgo de trasmitir dicho trastorno a su progenie.
9. Un procedimiento de acuerdo con la reivindicación 8, en el que la mutación es reconocida por un anticuerpo que se une específicamente a un epítopo portador de la mutación N324K.
10. Un anticuerpo que reconoce específicamente un epítopo que comprende la mutación N324K en la SEC ID N°: 1 de un perro.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]