Suceso de élite A2704-12 y métodos y estuches para identificar a dicho suceso en muestras biológicas.
Un método para identificar al suceso de élite A2704-12 en muestras biológicas,
cuyo método comprende ladetección de una región específica para el A2704-12, que comprende una parte de la región de ADN flanqueadoraen 5' o 3' del suceso de élite A2704-12 y una parte de la región de ADN ajeno contiguo a ella del suceso de éliteA2704-12, con por lo menos dos cebadores específicos o con una sonda específica, en el que dicha región de ADNflanqueadora en 5' comprende la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 1 hasta elnucleótido 209, dicha región de ADN ajeno contiguo a dicha región flanqueadora en 5' comprende la secuencia denucleótidos de SEQ ID NO. 1 desde el nucleótido 210 hasta el nucleótido 720, dicha región de ADN flanqueadora en3' comprende la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 569 hasta el nucleótido 1000, ydicha región de ADN ajeno contiguo a dicha región flanqueadora en 3' comprende la secuencia de nucleótidos deSEQ ID NO. 2 desde el nucleótido 1 hasta el nucleótido 568, en el que dicha sonda específica comprende unasecuencia de entre 50 y 500 pb que es idéntica por lo menos en un 80 % a dicha región específica para el A2704-12que comprende una parte de la región de ADN flanqueadora en 5' o 3' del suceso de élite A2704-12 y una parte dela región de ADN ajeno contiguo a ella del suceso de élite A2704-12; y en el que uno de dichos cebadoresespecíficos es un oligonucleótido de por lo menos 10 a 30 nucleótidos que reconoce a una parte de la región deADN flanqueadora en 5' o 3' del suceso de élite A2704-12, y en el que uno de dichos cebadores específicos es unoligonucleótido de por lo menos 10 a 30 nucleótidos, que reconoce a una parte de la región de ADN ajeno delsuceso de élite A2704-12 contiguo a dicha región flanqueadora en 5' o 3'.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2006/003454.
Solicitante: Bayer CropScience NV .
Nacionalidad solicitante: Bélgica.
Dirección: J.E. Mommaertslaan 14 1831 Diegem BELGICA.
Inventor/es: DE BEUCKELEER, MARC.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/82 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › para células vegetales.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2388548_T3.pdf
Fragmento de la descripción:
Suceso de élite A2704-12 y métodos y estuches para identificar a dicho suceso en muestras biológicas
Campo del invento
Este invento pertenece a métodos y estuches para identificar en muestras biológicas a la presencia de un material vegetal que comprende específicamente el suceso de transformación A2704-12, así como a plantas de soja transgénicas, a un material de plantas y a unas semillas que contienen dicho suceso. Las plantas de soja del invento combinan el fenotipo de tolerancia a herbicidas con un rendimiento agronómico, una estabilidad genética y una adaptabilidad a diferentes trasfondos genéticos que son equivalentes al linaje de planta de soja no transformado en la ausencia de presión de malezas.
Antecedentes del invento
La expresión fenotípica de un transgén en una planta es determinada tanto por la estructura del gen propiamente dicho y por su localización en el genoma de la planta. Al mismo tiempo, la presencia del transgén (en un ADN ajeno) en diferentes localizaciones en el genoma influirá sobre el fenotipo global de la planta de diferentes maneras. La introducción con éxito desde un punto de vista agronómico o industrial de un rasgo comercialmente interesante en una planta por manipulación genética puede ser un proceso largo y tedioso, dependiente de diferentes factores. La transformación y la regeneración reales de plantas transformadas genéticamente son solamente las primeras en una serie de etapas de selección, que incluyen extensos procesos de caracterización genética, crianza y evaluación en pruebas realizadas en el campo, conduciendo finalmente a la selección de un suceso de élite.
La identificación inequívoca de un suceso de élite se está volviendo crecientemente importante a la vista de discusiones acerca de nuevos productos de piensos y alimentos, segregación de productos de GMO (organismos modificados genéticamente y que no son de GMO y la identificación de un material patentado. Idealmente, dicho método de identificación es a la vez rápido y sencillo, sin la necesidad de una extensa organización de laboratorio. Además, el método deberá proporcionar unos resultados que permitan una determinación inequívoca del suceso de élite sin ninguna interpretación por expertos, pero que se mantengan bajo escrutinio de los expertos si fuese necesario.
El A2704-12 se seleccionó como un suceso de élite en el desarrollo de plantas de soja (Glycine max L.) resistentes al herbicida Liberty®, por transformación de una planta de soja con un plásmido que comprende el gen pat sintético que codifica una tolerancia a fosfinotricina, y que se puede vender comercialmente como una planta de soja Liberty Link®. Se describen aquí las herramientas para el uso en métodos simples e inequívocos para la identificación del suceso de élite A2704-12 en muestras biológicas.
Sumario del invento
El presente invento se refiere a unos métodos para la identificación del suceso de élite A2704-12 en muestras biológicas, tal como se definen en las reivindicaciones, cuyos métodos están basados en unos cebadores o unas sondas que reconocen específicamente a las secuencias flanqueadoras en 5’ y/o 3’ del A2704-12 y al ADN ajeno contiguo a ellas.
En el presente invento, se proporciona un método para identificar al suceso de élite A2704-12 en muestras biológicas, cuyo método comprende la detección de una región específica para el A2704-12 que comprende una parte de la región de ADN flanqueadora en 5’ o 3’ del suceso de élite A2704-12 y una parte de la región de ADN ajeno contiguo a ella del suceso de élite A2704-12, con por lo menos dos cebadores específicos o con una sonda específica, en el que dicha región de ADN flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 1 hasta el nucleótido 209, dicha región de ADN ajeno contiguo a dicha región flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 210 hasta el nucleótido 720, dicha región de ADN flanqueadora en 3’ comprende la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 569 hasta el nucleótido 1000, y dicha región de ADN ajeno contiguo a dicha región flanqueadora en 3’ comprende la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 1 hasta el nucleótido 568, en el que dicha sonda específica comprende una secuencia de entre 50 y 500 pb (pares de bases) que es idéntica por lo menos en un 80 % a dicha región específica para el A2704-12 que comprende una parte de la región de ADN flanqueadora en 5’ o 3’ del suceso de élite A2704-12 y una parte de la región de ADN ajeno contiguo a ella del suceso de élite A2704-12; y en el que uno de dichos cebadores específicos es un nucleótido de por lo menos 10 a 30 nucleótidos, que reconoce a una parte de la región de ADN flanqueadora en 5’ o 3’ del suceso de élite A2704-12, y en el que uno de dichos cebadores específicos es un oligonucleótido de por lo menos 10 a 30 nucleótidos, que reconoce a una parte de la región de ADN ajeno del suceso de élite A2704-12, contiguo a dicha región flanqueadora en 5’ o 3’, y formas de realización de dicho método tal como aquí se reivindican.
También se proporciona aquí un estuche para identificar al suceso de élite A2704-12 en muestras biológicas, comprendiendo dicho estuche un cebador que reconoce a la región flanqueadora en 5’ de A2704-12, teniendo dicha región flanqueadora en 5’ la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 1 hasta el nucleótido 209, o un cebador que reconoce a la región flanqueadora en 3’ de A2704-12, teniendo dicha región flanqueadora en 3’ la secuencia de nucleótidos del complemento de SEQ ID NO. 2 desde el nucleótido 569 hasta el nucleótido 1000, y un cebador que reconoce a una secuencia situada dentro del ADN ajeno, teniendo dicho ADN ajeno la secuencia de nucleótidos del complemento de SEQ ID NO. 1 desde el nucleótido 210 hasta el nucleótido 720 o la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 1 hasta el nucleótido 568, en el que dicho cebador es un oligonucleótido de por lo menos 10 a 30 nucleótidos, así como formas de realización relacionadas tal como aquí se reivindican.
Además se proporciona aquí un par de cebadores para usarse en un protocolo de identificación por PCR del A270412, teniendo dicho primer cebador una secuencia que, en condiciones optimizadas de PCR, reconoce a una secuencia situada dentro de la región flanqueadora en 5’ o 3’ de A2704-12, teniendo dicha región flanqueadora en 5’ la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 1 hasta el nucleótido 209 y teniendo dicha región flanqueadora en 3’ la secuencia de nucleótidos del complemento de SEQ ID NO. 2 desde el nucleótido 569 hasta el nucleótido 1000, y teniendo dicho segundo cebador una secuencia que, en condiciones optimizadas de PCR, reconoce específicamente a una secuencia de la región de ADN ajeno del suceso de élite A2704-12 contiguo a dicha región flanqueadora en 5’ o 3’, en el que dicha región de ADN ajeno contiguo a dicha región flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 210 hasta el nucleótido 720, y dicha región de ADN ajeno contiguo a dicha región flanqueadora en 3’ comprende la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 1 hasta el nucleótido 568, y en el que cada uno de dichos cebadores es un oligonucleótido de por lo menos 10 a 30 nucleótidos, así como formas de realización relacionadas como se reivindican aquí.
De acuerdo con este invento se proporciona también un estuche para identificar al suceso de élite A2704-12 en muestras biológicas, comprendiendo dicho estuche una sonda específica, capaz de hibridarse específicamente con una región específica del A2704-12, en el que dicha sonda específica comprende una secuencia de entre 50 y 500 pb que tiene una identidad entre secuencias de por lo menos un 80 % con una secuencia que comprende una parte de la secuencia flanqueadora en 5’ o la secuencia flanqueadora en 3’ de A2704-12 y la secuencia del ADN ajeno contiguo a ella, en el que dicha secuencia flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 1 hasta el nucleótido 209, el ADN ajeno contiguo a ella comprende la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 210 hasta el nucleótido 720, y en el que dicha secuencia flanqueadora... [Seguir leyendo]
Reivindicaciones:
1. Un método para identificar al suceso de élite A2704-12 en muestras biológicas, cuyo método comprende la detección de una región específica para el A2704-12, que comprende una parte de la región de ADN flanqueadora en 5’ o 3’ del suceso de élite A2704-12 y una parte de la región de ADN ajeno contiguo a ella del suceso de élite 5 A2704-12, con por lo menos dos cebadores específicos o con una sonda específica, en el que dicha región de ADN flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 1 hasta el nucleótido 209, dicha región de ADN ajeno contiguo a dicha región flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 210 hasta el nucleótido 720, dicha región de ADN flanqueadora en 3’ comprende la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 569 hasta el nucleótido 1000, y dicha región de ADN ajeno contiguo a dicha región flanqueadora en 3’ comprende la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 1 hasta el nucleótido 568, en el que dicha sonda específica comprende una secuencia de entre 50 y 500 pb que es idéntica por lo menos en un 80 % a dicha región específica para el A2704-12 que comprende una parte de la región de ADN flanqueadora en 5’ o 3’ del suceso de élite A2704-12 y una parte de la región de ADN ajeno contiguo a ella del suceso de élite A2704-12; y en el que uno de dichos cebadores
específicos es un oligonucleótido de por lo menos 10 a 30 nucleótidos que reconoce a una parte de la región de ADN flanqueadora en 5’ o 3’ del suceso de élite A2704-12, y en el que uno de dichos cebadores específicos es un oligonucleótido de por lo menos 10 a 30 nucleótidos, que reconoce a una parte de la región de ADN ajeno del suceso de élite A2704-12 contiguo a dicha región flanqueadora en 5’ o 3’.
2. El método de la reivindicación 1, comprendiendo dicho método amplificar un fragmento de ADN de entre 100 y 500 pb a partir de un ácido nucleico presente en dichas muestras biológicas usando una reacción en cadena de la polimerasa con por lo menos dos cebadores, reconociendo uno de dichos cebadores a la región flanqueadora en 5’ de A2704-12, teniendo dicha región flanqueadora en 5’ la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 1 hasta el nucleótido 209, o a la región flanqueadora en 3’ de A2704-12, teniendo dicha región flanqueadora en 3’ la secuencia de nucleótidos del complemento de SEQ ID NO. 2 desde el nucleótido 569 hasta el
nucleótido 1000, reconociendo el otro cebador de dichos cebadores a una secuencia situada dentro del ADN ajeno que tiene la secuencia de nucleótidos del complemento de SEQ ID NO. 1 desde el nucleótido 210 hasta el nucleótido 720 o la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 1 hasta el nucleótido 568.
3. El método de la reivindicación 2, en el que dicho cebador que reconoce a la región flanqueadora en 5’ consiste en una secuencia de nucleótidos de 17 a 200 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 1 hasta el nucleótido 209, o dicho cebador que reconoce a la región flanqueadora en 3’ de A2704-12 consiste en una secuencia de nucleótidos de 17 a 200 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos del complemento de SEQ ID NO. 2 desde el nucleótido 569 hasta el nucleótido 1000, y dicho cebador que reconoce a una secuencia dentro del ADN ajeno consiste en 17 a 200 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos del complemento de SEQ ID NO. 1 desde
el nucleótido 210 hasta el nucleótido 720 o la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 1 hasta el nucleótido 568.
4. El método de la reivindicación 2, en el que dicho cebador que reconoce a la región flanqueadora en 5’ comprende en su extremo 3’ final una secuencia de nucleótidos de por lo menos 17 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos de SEQ ID NO. 1 desde el nucleótido 1 hasta el nucleótido 209, o dicho cebador que reconoce a la región flanqueadora en 3’ de A2704-12 comprende en su extremo 3’ final una secuencia de nucleótidos de por lo menos 17 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos del complemento de SEQ ID NO. 2 desde el nucleótido 569 hasta el nucleótido 1000, y dicho cebador que reconoce a una secuencia situada dentro del ADN ajeno comprende en su extremo 3’ por lo menos 17 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos del complemento de SEQ ID NO. 1 desde el nucleótido 210 hasta el
45 nucleótido 720 o la secuencia de nucleótidos de SEQ ID NO. 2 desde el nucleótido 1 hasta el nucleótido 568.
5. El método de la reivindicación 4, en el que dichos cebadores comprenden las secuencias de SEQ ID NO. 4 y SEQ ID NO. 8, respectivamente.
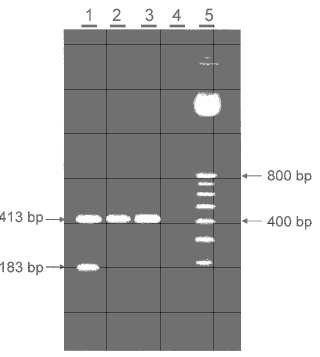
6. El método de la reivindicación 5, cuyo método comprende amplificar un fragmento de aproximadamente 185 pb usando el protocolo de identificación del A2704-12.
7. Un estuche para identificar el suceso de élite en muestras biológicas, comprendiendo dicho estuche un cebador que reconoce a la región flanqueadora en 5’ de A2704-12 teniendo dicha región flanqueadora en 5’ la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 1 hasta el nucleótido 209, o un cebador que reconoce a la región flanqueadora en 3’ de A2704-12, teniendo dicha región flanqueadora en 3’ la secuencia de nucleótidos del complemento de SEQ ID No 2 desde el nucleótido 569 hasta el nucleótido 1000, y un cebador que reconoce a una
55 secuencia situada dentro del ADN ajeno, teniendo dicho ADN ajeno la secuencia de nucleótidos del complemento de SEQ ID No 1 desde el nucleótido 210 hasta el nucleótido 720 o la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 1 hasta el nucleótido 568, en el dicho cebador es un oligonucleótido de por lo menos 10 a 30 nucleótidos.
8. El estuche de la reivindicación 7, en el dicho cebador que reconoce a la región flanqueadora en 5’ consiste en una secuencia de nucleótidos de 17 a 200 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 1 hasta el nucleótido 209, o dicho cebador que reconoce a la región flanqueadora en 3’ de A2704-12 consiste en una secuencia de nucleótidos de 17 a 200 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos del complemento de SEQ ID No 2 desde el nucleótido 569 hasta el nucleótido 1000, y dicho cebador que reconoce a una secuencia dentro del ADN ajeno consiste en 17 a 200 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos del complemento de SEQ ID No 1 desde el nucleótido 210 hasta el nucleótido 720 o la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 1 hasta el nucleótido
568.
9. El estuche de la reivindicación 7, en el dicho cebador que reconoce a la región flanqueadora en 5’ comprende en su extremo 3’ final una secuencia de nucleótidos de por lo menos 17 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 1 hasta el nucleótido 209, o dicho cebador que reconoce a la región flanqueadora en 3’ de A2704-12 comprende en su extremo 3’ final una secuencia de nucleótidos de por lo menos 17 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos del complemento de SEQ ID No 2 desde el nucleótido 569 hasta el nucleótido 1000, y dicho cebador que reconoce a una secuencia dentro del ADN ajeno comprende en su extremo 3’ por lo menos 17 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos del complemento de SEQ ID No 1 desde el nucleótido 210 hasta el nucleótido 720 o la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 1 hasta el nucleótido 568.
10. El estuche de la reivindicación 7, que comprende un cebador que consiste en la secuencia de SEQ ID No 4 y un cebador que consiste en la secuencia de SEQ ID No 8.
11. Un par de cebadores destinado a usarse en un protocolo de identificación por PCR del A2704-12, teniendo dicho primer cebador una secuencia que, en condiciones optimizadas de PCR, reconoce específicamente a una secuencia situada dentro de la región flanqueadora en 5’ o 3’ de A2704-12, teniendo dicha región flanqueadora en 5’ la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 1 hasta el nucleótido 209 y teniendo dicha región flanqueadora en 3’ la secuencia de nucleótidos del complemento de SEQ ID No 2 desde el nucleótido 569 hasta el nucleótido 1000, y teniendo dicho segundo cebador una secuencia que, en condiciones optimizadas de PCR, reconoce específicamente a una secuencia de la región de ADN ajeno del suceso de élite A2704-12, contiguo a dicha región flanqueadora en 5’ o 3’, en el que dicha región de ADN ajeno contiguo a dicha región flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 210 hasta el nucleótido 720, y dicha región de ADN ajeno contiguo a dicha región flanqueadora en 3’ comprende la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 1 hasta el nucleótido 568, y en el que cada uno de dichos cebadores es un oligonucleótido de por lo menos 10 a 30 nucleótidos.
12. El par de cebadores de la reivindicación 11, en el que dicho primer cebador consiste en una secuencia de nucleótidos de 17 a 200 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 1 hasta el nucleótido 209 o una secuencia de nucleótidos de 17 a 200 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 569 hasta el nucleótido 1000.
13. El par de cebadores de la reivindicación 11, en el que dicho primer cebador comprende en su extremo 3’ final una secuencia de nucleótidos de por lo menos 17 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 1 hasta el nucleótido 209 o una secuencia de nucleótidos de por lo menos 17 nucleótidos consecutivos seleccionados entre la secuencia de nucleótidos del complemento de SEQ ID No 2 desde el nucleótido 569 hasta el nucleótido 1000.
14. Un cebador que comprende en su extremo 3’ final la secuencia de SEQ ID No 4.
15. El par de cebadores de la reivindicación 11, que comprende un primer cebador que comprende en su extremo 3’ final la secuencia de SEQ ID No 4 y un segundo cebador que comprende en su extremo 3’ final la secuencia de SEQ ID No 8.
16. El método de la reivindicación 1, en el que dicho método comprende hibridar un ácido nucleico de muestras biológicas con una sonda específica para el A2704-12.
17. El método de la reivindicación 16, en el que la secuencia de dicha sonda específica tiene una identidad entre secuencias de por lo menos un 80 % con la SEQ ID No 1 del nucleótido 160 hasta el 260 o con la SEQ ID No 2 desde el nucleótido 520 hasta el 620, o con el complemento de dichas secuencias.
18. Un estuche para identificar el suceso de élite A2704-12 en muestras biológicas, comprendiendo dicho estuche una sonda específica, capaz de hibridarse específicamente con una región específica del A2704-12, en el que dicha sonda específica comprende una secuencia de entre 50 y 500 pb que tiene una identidad ente secuencias de por lo menos un 80 % con una secuencia que comprende una parte de la secuencia flanqueadora en 5’ o la secuencia flanqueadora en 3’ del A2704-12 y la secuencia del ADN ajeno contiguo a ella, en el que dicha secuencia flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 1 hasta el nucleótido 209, el ADN ajeno contiguo a ella comprende la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 210 hasta el nucleótido 720, y en el que dicha secuencia flanqueadora en 3’ comprende la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 569 hasta el nucleótido 1000, y el ADN ajeno contiguo a ella comprende la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 1 hasta el nucleótido 568.
19. El estuche de la reivindicación 18, en el que la secuencia de dicha sonda específica tiene una identidad entre secuencias de por lo menos un 80 % con la SEQ ID No 1 desde el nucleótido 160 hasta el 260 o con la SEQ ID No 2 desde el nucleótido 520 hasta el 620, o con el complemento de dichas secuencias.
20. Una sonda específica para la identificación del suceso de élite A2704-12 en muestras biológicas, que comprende una secuencia de entre 50 y 500 pb que tiene una identidad entre secuencias de por lo menos un 80 % con una secuencia que comprende una parte de la secuencia flanqueadora 5’ o la secuencia flanqueadora en 3’ del A270412 y la secuencia del ADN ajeno contiguo a ella, o el complemento de la misma, en la que dicha secuencia flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 1 hasta el nucleótido 209, el ADN ajeno contiguo a ella comprende la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 210 hasta el nucleótido 720, y en la que dicha secuencia flanqueadora en 3’ comprende la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 569 hasta el nucleótido 1000, y el ADN ajeno contiguo a ella comprende la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 1 hasta el nucleótido 568.
21. La sonda de la reivindicación 20 que tiene una identidad entre secuencias de por lo menos un 80 % con la SEQ ID No 1 desde el nucleótido 160 hasta el nucleótido 260 o con la SEQ ID No 2 desde el nucleótido 520 hasta el nucleótido a 620, o con el complemento de dichas secuencias.
22. El uso del par de cebadores de una cualquiera de las reivindicaciones 11 hasta 13 o de la reivindicación 15, o de la sonda de la reivindicación 20 ó 21, en un método para confirmar la pureza de las semillas en muestras de semillas.
23. El uso del par de cebadores de una cualquiera de las reivindicaciones 11 hasta 13 o de la reivindicación 15 o de la sonda de la reivindicación 20 o 21, en un método para escrutar semillas en cuanto a la presencia del A2704-12 en muestras de lotes de semillas.
24. Un amplicón discriminante que identifica específicamente al suceso de élite A2704-12 de plantas de soja, cuyo amplicón es obtenible a partir de una muestra de ácido nucleico que comprende dicho suceso de élite por medio de una PCR usando un cebador que reconoce específicamente a una secuencia situada dentro de la región flanqueadora en 5’ o 3’ del suceso de élite y un cebador que reconoce específicamente al ADN ajeno contiguo a ella, en el que dicha región flanqueadora en 5’ comprende la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 1 hasta el nucleótido 209, el ADN ajeno contiguo a ella comprende la secuencia de nucleótidos de SEQ ID No 1 desde el nucleótido 210 hasta el nucleótido 720, y en el que dicha región flanqueadora en 3’ comprende la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 569 hasta el nucleótido 1000, y el ADN ajeno contiguo a ella comprende la secuencia de nucleótidos de SEQ ID No 2 desde el nucleótido 1 hasta el nucleótido 568, y en el que dicho cebador específico es un oligonucleótido de por lo menos 10 a 30 nucleótidos.
Patentes similares o relacionadas:
Composiciones para la supresión de la formación de inhibidores contra el factor VIII en pacientes con hemofilia A., del 22 de Julio de 2020, de THE TRUSTEES OF THE UNIVERSITY OF PENNSYLVANIA: Una composición que comprende material vegetal liofilizado que comprende al menos un fragmento de FVIII conjugado con la subunidad B de la toxina […]
Plantas con rendimiento incrementado y método de obtención de dichas plantas, del 15 de Julio de 2020, de CENTRE NATIONAL DE LA RECHERCHE SCIENTIFIQUE (CNRS): Método para mejorar por lo menos un carácter fenotípico seleccionado de entre el rendimiento en semillas, la velocidad de germinación o el crecimiento […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Moléculas de ácido nucleico de Nucampholin para controlar plagas de insectos coleópteros, del 17 de Junio de 2020, de FRAUNHOFER-GESELLSCHAFT ZUR FORDERUNG DER ANGEWANDTEN FORSCHUNG E.V.: Una molécula de ácido nucleico aislada que comprende un polinucleótido que codifica una molécula de ácido ribonucleico de horquilla (hpARN) con una estructura […]
Polinucleótidos aislados y métodos y plantas que usan los mismos para regular la acidez de las plantas, del 10 de Junio de 2020, de The State of Israel, Ministry of Agriculture and Rural Development, Agricultural Research Organization, (A.R.O.), Volcani Cent: Una célula de planta o una planta que comprende una construcción de ácido nucleico que comprende una secuencia de ácido nucleico que codifica un polipéptido […]
Genes de Chromobacterium subtsugae, del 27 de Mayo de 2020, de Marrone Bio Innovations, Inc: Una célula vegetal que comprende un polipéptido que tiene la secuencia de aminoácidos expuesta en las SEQ ID NO: 1, 2 o 3.
Moléculas polinucleotídicas para la regulación génica en plantas, del 6 de Mayo de 2020, de MONSANTO TECHNOLOGY, LLC: Un procedimiento para regular la expresión de un gen diana endógeno en plantas en crecimiento, que comprende: aplicar por vía tópica sobre […]
Producción de partículas similares al virus de la gripe en plantas, del 6 de Mayo de 2020, de MEDICAGO INC.: Un ácido nucleico que comprende una región reguladora activa en una planta y un potenciador de la expresión activo en una planta, la región […]