PRODUCTO Y METODO.
Un conjunto de menos de 1.000 sondas oligonucleotídicas, en el que dicho conjunto comprende los oligonucleótidos descritos en la Tabla 2b para los que se proporcionan las secuencias que tienen las secuencias como se muestra en la secuencia No.
61, 77, 93, 108, 110, 192, 250, 308, 309, 310, 321, 327, 338, 339, 360, 361, 364, 365, 368, 378, 380, 381, 382, 384, 390, 391, 397, 398, 401, 403, 406, 411, 412, 413, 414, 415, 416, 418, 421, 423, 424, 428, 434, 436, 438, 441, 442, 450, 452, 453, 458, 460, 463, 464, 469, 471, 473, 474, 475, 476, 477, 478, 479, 482, 483, 485, 487, 488, 489, 492, 493, 494, 495, 503, 504, 505, 506, 507, 508, 509, 510, 512, 513, 515, 518, 519, 521, 523, 524, 526, 527, 529, 530, 532, 534, 560, 562, 564, 565, 566, 567, 568, 570, 571, 572, 575, 576, 578, 579, 580, 583, 585, 589, 591, 592, 593, 594, 596, 598, 600, 601, 605, 607, 610, 612, 613, 614, 615, 617, 618, 619, 622, 624, 628, 629, 630, 631, 632, 633, 634, 635, 636, 637, 638, 639, 643, 644, 645, 649, 651, 656, 658, 660, 661, 663, 665, 672, 673, 675, 679, 682, 683, 684, 685, 687, 688, 689, 691, 693, 696, 697, 699, 701, 702, 705, 706, 707, 708, 709, 711, 714, 718, 720, 721, 722, 724, 726, 736, 739, 747, 757, 758, 764, 766, 768, 773, 776, 782, 785, 796, 801, 808, 814, 817, 821, 825, 833, 837, 839, 849, 860, 864, 865, 867, 869, 870, 871, 873, 875, 876, 878, 879, 881, 885, 887, 889, 891, 892, 893, 895, 897, 899, 903, 904, 905, 906, 907, 908, 910, 911, 912, 915, 917, 926, 938, 939, 947, 949, 1.028, 1.056, 1.071, 1.074, 1.081, 1.083, 1.084, 1.099, 1.109, 1.118, 1.125, 1.139, 1.148, 1.160, 1.165, 1.172, 1.178, 1.180, 1.181, 1.182, 1.183, 1.185, 1.186, 1.188, 1.189, 1.190, 1.192, 1.193, 1.195, 1.196, 1.197, 1.198, 1.199, 1.200, 1.201, 1.202, 1.203, 1.204, 1.205, 1.207, 1.208, 1.209, 1.210, 1.211, 1.212, 1.213, 1.214, 1.215, 1.216, 1.217, 1.218, 1.219, 1.220, 1.221, 1.224, 1.226, 1.228, 1.230, 1.231, 1.239, 1.331, 1.332, 1.335, 1.336, 1.337, 1.338, 1.344, 1.348, 1.351, 1.352, 1.353, 1.355, 1.360, 1.361, 1.364, 1.365, 1.366, 1.368, 1.369, 1.370, 1.371, 1.372, 1.374, 1.378, 1.380, 1.382, 1.387, 1.389, 1.390, 1.391, 1.392, 1.394, 1.395, 1.396, 1.397, 1.399, 1.440, 1.448, 1.453, 1.456, 1.460, 1.495 y g6
o un conjunto en el que uno o más de dichos oligonucleótidos está sustituido, en el que cada oligonucleótido que está sustituido está sustituido por una parte de dicho oligonucleótido, parte que tiene una longitud de 15-200 bases, o por un oligonucleótido con una secuencia complementaria a dicho oligonucleótido
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2003/005102.
Solicitante: DIAGENIC AS.
Nacionalidad solicitante: Noruega.
Dirección: GRENSEVEIEN 92,0663 OSLO.
Inventor/es: SHARMA, PRAVEEN, SAHNI,NARINDER,SINGH, LONNEBORG,ANDERS.
Fecha de Publicación: .
Fecha Concesión Europea: 3 de Marzo de 2010.
Clasificación Internacional de Patentes:
- G01N33/68V2
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
Producto y método.
La presente invención se refiere a sondas oligonucleotídicas, para usarse en la evaluación de los niveles de transcritos génicos en una célula, que pueden usarse en técnicas analíticas, particularmente técnicas de diagnóstico. Convenientemente, las sondas se proporcionan en forma de kit. Pueden usarse diferentes conjuntos de sondas en técnicas para preparar patrones de expresión génica e identificar, diagnosticar o monitorizar diferentes estados, tales como enfermedades, condiciones o estadios de éstas. También se proporcionan métodos para identificar sondas adecuadas y su uso en métodos de la invención.
La identificación de métodos rápidos y sencillos para el análisis de muestras, por ejemplo, para aplicaciones diagnósticas, permanece como el objetivo de muchos investigadores. Los usuarios finales solicitan métodos que sean rentables, que produzcan resultados estadísticamente significativos y que puedan implementarse rutinariamente sin la necesidad de individuos con gran experiencia técnica.
El análisis de la expresión génica en las células se ha usado para proporcionar información sobre el estado de esas células y en gran medida sobre el estado del individuo del que se obtienen las células. Se ha identificado que la expresión relativa de varios genes en una célula es el reflejo de un estado particular en un cuerpo. Por ejemplo, se sabe que las células cancerosas presentan una expresión alterada de varias proteínas y que los transcritos o las proteínas expresadas pueden usarse, por lo tanto, como marcadores de ese estado patológico.
Así, puede analizarse el tejido de biopsias para la presencia de estos marcadores y pueden identificarse las células que se originan a partir del sitio de la enfermedad en otros tejidos o fluidos del cuerpo por la presencia de los marcadores.
Además, los productos de la expresión alterada pueden liberarse en la corriente sanguínea y estos productos pueden analizarse. Además, las células que han entrado en contacto con células enfermas pueden resultar afectadas por su contacto directo con esas células lo que resulta en expresión génica alterada y su expresión o productos de la expresión pueden analizarse de manera similar.
Sin embargo, hay algunas limitaciones con estos métodos. Por ejemplo, el uso de marcadores tumorales específicos para identificar cáncer presenta varios defectos, tales como ausencia de especificidad o sensibilidad, asociación del marcador con estados patológicos además del tipo de cáncer específico, y dificultad de detección en individuos asintomáticos.
Además del análisis de uno o dos transcritos o proteínas marcadoras, se han analizado, más recientemente, los patrones de expresión génica. Affymetrix ha descrito un conjunto seleccionado de oligonucleótidos que abarca más de 39.000 transcritos que permitirían dicho análisis (Affymetrix GeneChip Human Genome U133 Assay Set HG-U133A, Base de Datos GEO NCBI, No. de Acceso GPL96). La mayor parte del trabajo que implica análisis de expresión génica a gran escala con implicaciones en el diagnóstico de enfermedades ha implicado muestras clínicas que tienen su origen en tejidos o células enfermas. Por ejemplo, varias publicaciones recientes, que demuestran que los datos de expresión génica pueden usarse para distinguir entre tipos de cáncer similares, han usado muestras clínicas de tejidos o células enfermos (Alon et al. 1999, PNAS, 96, p6745-6750; Golub et al. 1999, Science, 286, p531-537; Alizadeh et al, 2000, Nature, 403, p503-511; Bittner et al., 2000, Nature, 406, p536-540). WO 02/059271 describe genes que se expresan de manera diferente en biopsias tumorales comparado con tejidos normales y su uso en métodos de diagnóstico.
Sin embargo, estos métodos se han basado en el análisis de una muestra que contiene células enfermas o productos de estas células o células que han entrado en contacto con células enfermas. El análisis de dichas muestras se basa en el conocimiento de la presencia de una enfermedad y su localización, que puede ser difícil en pacientes asintomáticos. Además, las muestras no siempre pueden tomarse del sitio de la enfermedad, p. ej., en las enfermedades del cerebro.
En un descubrimiento de gran significancia, los presentes inventores identificaron el potencial previamente sin explotar de todas las células de un cuerpo para proporcionar información respecto al estado del organismo del que se obtuvieron las células. WO98/49342 describe el análisis de la expresión génica de células distantes del sitio de la enfermedad, p. ej. sangre periférica recogida lejos de un sitio de cáncer.
Este descubrimiento se basa en la premisa de que las diferentes partes del cuerpo de un organismo existen en interacción dinámica entre sí. Cuando una enfermedad afecta una parte del cuerpo, también resultan afectadas otras partes del cuerpo. La interacción resulta de una amplio espectro de señales bioquímicas que se liberan desde el área enferma, afectando otras áreas del cuerpo. Aunque la naturaleza de los cambios bioquímicos y fisiológicos inducidos por las señales liberadas puede variar en las diferentes partes del cuerpo, los cambios pueden medirse al nivel de la expresión génica y usarse para propósitos de diagnóstico.
El estado fisiológico de una célula en un organismo se determina por el patrón con el que los genes se expresan en él. El patrón depende de los estímulos biológicos internos y externos a los que está expuesta dicha célula, y cualquier cambio tanto en la magnitud como en la naturaleza de estos estímulos puede dar lugar a un cambio en el patrón con el que se expresan los diferentes genes en la célula. Existe una comprensión creciente de que mediante el análisis de los cambios sistémicos en los patrones de expresión génica en células de muestras biológicas, es posible proporcionar información sobre el tipo y naturaleza de los estímulos biológicos que están actuando sobre ellas. Así, por ejemplo, monitorizando la expresión de una gran número de genes en células de una muestra de ensayo, es posible determinar si sus genes se expresan con un patrón característico para una enfermedad, condición particular o estadio de éstas. La medición de los cambios de las actividades génicas en células, p. ej. de tejidos o fluidos corporales, se está revelando, por lo tanto, como una herramienta potente para el diagnóstico de enfermedades.
Dichos métodos tienen varias ventajas. A menudo, la obtención de muestras clínicas de determinadas áreas del cuerpo que está enfermo puede ser difícil y puede implicar invasiones no deseadas del cuerpo, por ejemplo la biopsia se usa a menudo para obtener muestras de cáncer. En algunos casos, tal como en la enfermedad de Alzheimer, el espécimen de cerebro enfermo sólo puede obtenerse post-mortem. Además, los especímenes de tejido que se obtienen son a menudo heterogéneos y pueden contener una mezcla de células enfermas y no enfermas, lo que hace que el análisis de los datos de expresión génica generados sea complejo y difícil.
Se ha sugerido que un conjunto de tejidos tumorales que parece ser homogéneo patogenéticamente respecto a las apariencias morfológicas del tumor puede ser altamente heterogéneo a nivel molecular (Alizadeh, 2000, supra) y, de hecho, podría contener tumores que representan enfermedades esencialmente diferentes (Alizadeh, 2000, supra; Golub, 1999, supra). Para el propósito de identificar una enfermedad, condición o un estadio de éstas, es altamente deseable cualquier método que no requiera muestras clínicas que se originen directamente de tejidos o células enfermas ya que las muestras clínicas que representan una mezcla homogénea de tipos celulares pueden obtenerse de una región fácilmente accesible del cuerpo.
Whitney et al. (2003, PNAS, Vol. 100(4), p 1896-1901) han analizado la individualidad y variación de los patrones de expresión génica en sangre normal para proporcionar una base de datos con la que pueden compararse los patrones de expresión génica asociados a enfermedades. Los métodos para analizar conjuntos de datos complejos los han revisado Sherlock et al. (2000, Current Opinion in Immunology, Vol. 12, p 201-205).
Ahora hemos identificado un conjunto de sondas con una utilidad sorprendente para identificar una o más enfermedades. Así, describimos ahora sondas y conjuntos de sondas obtenidas de células que no son células enfermas y que no han entrado en contacto con células enfermas, que corresponden a genes que presentan una expresión alterada...
Reivindicaciones:
1. Un conjunto de menos de 1.000 sondas oligonucleotídicas, en el que dicho conjunto comprende los oligonucleótidos descritos en la Tabla 2b para los que se proporcionan las secuencias que tienen las secuencias como se muestra en la secuencia No. 61, 77, 93, 108, 110, 192, 250, 308, 309, 310, 321, 327, 338, 339, 360, 361, 364, 365, 368, 378, 380, 381, 382, 384, 390, 391, 397, 398, 401, 403, 406, 411, 412, 413, 414, 415, 416, 418, 421, 423, 424, 428, 434, 436, 438, 441, 442, 450, 452, 453, 458, 460, 463, 464, 469, 471, 473, 474, 475, 476, 477, 478, 479, 482, 483, 485, 487, 488, 489, 492, 493, 494, 495, 503, 504, 505, 506, 507, 508, 509, 510, 512, 513, 515, 518, 519, 521, 523, 524, 526, 527, 529, 530, 532, 534, 560, 562, 564, 565, 566, 567, 568, 570, 571, 572, 575, 576, 578, 579, 580, 583, 585, 589, 591, 592, 593, 594, 596, 598, 600, 601, 605, 607, 610, 612, 613, 614, 615, 617, 618, 619, 622, 624, 628, 629, 630, 631, 632, 633, 634, 635, 636, 637, 638, 639, 643, 644, 645, 649, 651, 656, 658, 660, 661, 663, 665, 672, 673, 675, 679, 682, 683, 684, 685, 687, 688, 689, 691, 693, 696, 697, 699, 701, 702, 705, 706, 707, 708, 709, 711, 714, 718, 720, 721, 722, 724, 726, 736, 739, 747, 757, 758, 764, 766, 768, 773, 776, 782, 785, 796, 801, 808, 814, 817, 821, 825, 833, 837, 839, 849, 860, 864, 865, 867, 869, 870, 871, 873, 875, 876, 878, 879, 881, 885, 887, 889, 891, 892, 893, 895, 897, 899, 903, 904, 905, 906, 907, 908, 910, 911, 912, 915, 917, 926, 938, 939, 947, 949, 1.028, 1.056, 1.071, 1.074, 1.081, 1.083, 1.084, 1.099, 1.109, 1.118, 1.125, 1.139, 1.148, 1.160, 1.165, 1.172, 1.178, 1.180, 1.181, 1.182, 1.183, 1.185, 1.186, 1.188, 1.189, 1.190, 1.192, 1.193, 1.195, 1.196, 1.197, 1.198, 1.199, 1.200, 1.201, 1.202, 1.203, 1.204, 1.205, 1.207, 1.208, 1.209, 1.210, 1.211, 1.212, 1.213, 1.214, 1.215, 1.216, 1.217, 1.218, 1.219, 1.220, 1.221, 1.224, 1.226, 1.228, 1.230, 1.231, 1.239, 1.331, 1.332, 1.335, 1.336, 1.337, 1.338, 1.344, 1.348, 1.351, 1.352, 1.353, 1.355, 1.360, 1.361, 1.364, 1.365, 1.366, 1.368, 1.369, 1.370, 1.371, 1.372, 1.374, 1.378, 1.380, 1.382, 1.387, 1.389, 1.390, 1.391, 1.392, 1.394, 1.395, 1.396, 1.397, 1.399, 1.440, 1.448, 1.453, 1.456, 1.460, 1.495 y g6
o un conjunto en el que uno o más de dichos oligonucleótidos está sustituido, en el que cada oligonucleótido que está sustituido está sustituido por una parte de dicho oligonucleótido, parte que tiene una longitud de 15-200 bases, o por un oligonucleótido con una secuencia complementaria a dicho oligonucleótido.
2. Un conjunto de menos de 1.000 sondas oligonucleotídicas, en el que dicho conjunto comprende los oligonucleótidos descritos en la Tabla 4b para los que se proporcionan las secuencias que tienen las secuencias como se muestra en la secuencia No. 299, 300, 302, 304, 306, 308, 309, 310, 311, 313, 314, 315, 316, 321, 322, 323, 324, 325, 326, 327, 328, 330, 331, 335, 337, 338, 339, 360, 361, 363, 364, 365, 366, 368, 369, 370, 371, 373, 374, 378, 380, 381, 382, 383, 384, 386, 387, 388, 389, 390, 391, 394, 395, 396, 397, 398, 399, 400, 401, 402, 403, 405, 406, 407, 408, 409, 410, 411, 412, 412, 413, 414, 415, 416, 417, 418, 419, 420, 421, 422, 423, 424, 425, 426, 427, 428, 429, 430, 431, 432, 433, 434, 435, 436, 438, 441, 442, 446, 447, 448, 450, 452, 453, 454, 458, 459, 460, 461, 462, 463, 464, 469, 471, 472, 473, 474, 475, 476, 477, 478, 479, 481, 482, 483, 484, 485, 487, 488, 489, 490, 491, 492, 493, 494, 495, 496, 497, 499, 500, 502, 503, 504, 505, 506, 507, 508, 509, 510, 512, 513, 515, 518, 519, 521, 523, 524, 524, 526, 527, 529, 530, 532, 533, 534, 560, 561, 562, 563, 564, 565, 566, 567, 568, 570, 571, 572, 574, 575, 576, 577, 578, 579, 580, 581, 582, 583, 585, 586, 587, 588, 589, 590, 591, 592, 593, 594, 595, 596, 597, 598, 599, 600, 601, 602, 603, 605, 606, 607, 609, 610, 611, 612, 613, 614, 615, 617, 618, 619, 621, 622, 624, 625, 626, 627, 628, 629, 630, 631, 632, 634, 635, 636, 637, 638, 639, 641, 642, 643, 644, 645, 646, 647, 648, 649, 650, 651, 652, 653, 654, 655, 656, 657, 658, 660, 661, 663, 665, 666, 669, 670, 671, 672, 673, 674, 675, 676, 679, 682, 683, 684, 685, 686, 687, 688, 689, 690, 691, 692, 693, 694, 696, 697, 698, 699, 700, 701, 702, 703, 704, 705, 706, 707, 708, 709, 710, 711, 713, 714, 717, 718, 719, 720, 721, 722, 724, 726, 727, 728, 870, 871, 873, 878, 879, 883, 885, 887, 889, 890, 892, 893, 895, 896, 897, 898, 899, 900, 903, 904, 905, 906, 907, 908, 910, 911, 912, 913, 914, 915, 1.178, 1.180, 1.181, 1.182, 1.183, 1.185, 1.186, 1.188, 1.189, 1.190, 1.191, 1.193, 1.200, 1.332. 1.336, 1.337, 1.348, 1.351, 1.353, 1.355, 1.359, 1.361, 1.364, 1.365, 1.366, 1.367, 1.368, 1.369, 1.370, 1.372, 1.374, 1.382, 1.387, 1.389, 1.390, 1.391, 1.397, 1.399, 1.440, 1.447, 1.448, 1.449, 1.450, 1.453, 1.454, 1.490, 1.491, 1.492, 1.493, 1.494 y 1.495
o un conjunto en el que uno o más de dichos oligonucleótidos está sustituido, en el que cada oligonucleótido que está sustituido está sustituido por una parte de dicho oligonucleótido, parte que tiene una longitud de 15-200 bases, o por un oligonucleótido con una secuencia complementaria a dicho oligonucleótido.
3. Un conjunto de sondas oligonucleotídicas según la reivindicación 1 ó 2, en el que cada una de dichas sondas oligonucleotídicas tiene una longitud de 15 a 200 bases.
4. Un conjunto de sondas oligonucleotídicas según la reivindicación 1 a 3, en el que dichas sondas están inmovilizadas en uno o más soportes sólidos.
5. Un conjunto de sondas oligonucleotídicas según la reivindicación 4, en el que dicho soporte sólido es una lámina, filtro, membrana, placa o biochip.
6. Un kit que comprende un conjunto de sondas oligonucleotídicas según la reivindicación 4 ó 5 inmovilizado en uno o más soportes sólidos.
7. Un kit según la reivindicación 6 en el que dichas sondas están inmovilizadas en un único soporte sólido y cada sonda única se une a una región diferente de dicho soporte sólido.
8. Un kit según la reivindicación 6 ó 7 que comprende además materiales de estandarización.
9. El uso de un conjunto de sondas como se ha descrito en una cualquiera de las reivindicaciones 1 a 5 o un kit según se ha descrito en una cualquiera de las reivindicaciones 6 a 8 para determinar el patrón de expresión génica de una célula en una muestra de sangre reflejando este patrón el nivel de expresión génica de los genes a los que se unen dichas sondas oligonucleotídicas, que comprende al menos las etapas de:
a) aislar el ARNm de dicha célula, que puede opcionalmente transcribirse de forma inversa a ADNc;
b) hibridar el ARNm o ADNc de la etapa (a) con un conjunto de oligonucleótidos o un kit como se ha definido en una cualquiera de las reivindicaciones 1 a 8; y
c) evaluar la cantidad de ARNm o ADNc que hibrida con cada una de dichas sondas para producir dicho patrón.
10. Un método para preparar un patrón de transcrito génico estándar característico de cáncer de mama o enfermedad de Alzheimer o un estadio de ésta en un organismo que comprende al menos las etapas de:
a) aislar el ARNm de las células de una muestra de sangre de uno o más organismos que tienen cáncer de mama o enfermedad de Alzheimer o un estadio de éstas, que puede opcionalmente transcribirse de forma inversa a ADNc;
b) hibridar el ARNm o ADNc de la etapa (a) de un organismo con cáncer de mama o un estadio de éste con un conjunto de oligonucleótidos o un kit como se ha definido en una cualquiera de las reivindicaciones 1 ó 3 a 8 específico para cáncer de mama o un estadio de éste en un organismo y muestra de éste correspondiente al organismo y muestra de éste que se está investigando o hibridar el ARNm o ADNc de la etapa (a) de un organismo con enfermedad de Alzheimer o un estadio de ésta con un conjunto de oligonucleótidos o un kit como se ha definido en una cualquiera de las reivindicaciones 2 a 8 específico para la enfermedad de Alzheimer o un estadio de ésta en un organismo y muestra de éste correspondiente al organismo y muestra de éste que se está investigando; y
c) evaluar la cantidad de ARNm o ADNc que hibrida con cada una de dichas sondas para producir un patrón característico que refleja el nivel de expresión génica de los genes a los que se unen dichos oligonucleótidos, en la muestra con cáncer de mama o enfermedad de Alzheimer o un estadio de éstas.
11. Un método para preparar un patrón de transcritos génicos de ensayo que comprende al menos las etapas de:
a) aislar el ARNm de las células de una muestra de sangre de dicho organismo de ensayo, que puede opcionalmente transcribirse de forma inversa a ADNc;
b) hibridar el ARNm o ADNc de la etapa (a) con un conjunto de oligonucleótidos o un kit como se ha definido en una cualquiera de las reivindicaciones 1 ó 3 a 8 específicos para cáncer de mama o un estadio de éste en un organismo y muestra de éste correspondiente al organismo y muestra de éste que se está investigando, o con un conjunto de oligonucleótidos o un kit como se ha definido en una cualquiera de las reivindicaciones 2 a 8 específicos para enfermedad de Alzheimer o un estadio de ésta en un organismo y muestra de éste correspondiente al organismo y muestra de éste que se está investigando; y
c) evaluar la cantidad de ARNm o ADNc que hibrida con cada una de dichas sondas para producir dicho patrón que refleja el nivel de expresión génica de los genes a los que se unen dichos oligonucleótidos, en dicha muestra de ensayo.
12. Un método para diagnosticar o identificar o monitorizar cáncer de mama o enfermedad de Alzheimer o un estadio de ésta en un organismo, que comprende las etapas de:
a) aislar el ARNm de las células de una muestra de sangre de dicho organismo, que puede opcionalmente transcribirse de forma inversa a ADNc;
b) hibridar el ARNm o ADNc de la etapa (a) con un conjunto de oligonucleótidos o un kit como se ha definido en una cualquiera de las reivindicaciones 1 ó 3 a 8 específicos para cáncer de mama o un estadio de éste en un organismo y muestra de éste correspondiente al organismo y muestra de éste que se está investigando, o con un conjunto de oligonucleótidos o un kit como se ha definido en una cualquiera de las reivindicaciones 2 a 8 específicos para enfermedad de Alzheimer o un estadio de ésta en un organismo y muestra de éste correspondiente al organismo y muestra de éste que se está investigando;
c) evaluar la cantidad de ARNm o ADNc que hibrida con cada una de dichas sondas para producir un patrón característico que refleja el nivel de expresión génica de genes a los que se unen dichos oligonucleótidos en dicha muestra; y
d) comparar dicho patrón con un patrón estándar de diagnóstico preparado como se ha descrito en la reivindicación 10 usando una muestra de un organismo correspondiente al organismo y muestra que se está investigando para determinar el grado de correlación indicativo de la presencia de cáncer de mama o enfermedad de Alzheimer o un estadio de ésta en el organismo que se está investigando.
13. Un método según una cualquiera de las reivindicaciones 10 a 12 en el que dicho ARNm o ADNc se amplifica antes de la etapa b).
14. Un método según una cualquiera de las reivindicaciones 10 a 13 en el que los oligonucleótidos y/o el ARNm o ADNc están marcados.
15. Un método según una cualquiera de las reivindicaciones 10 a 14 en el que dicho patrón se expresa como una matriz de números que se refieren al nivel de expresión asociado con cada sonda.
16. Un método según una cualquiera de las reivindicaciones 10 a 15 en el que dicho organismo es un organismo eucariota, preferiblemente un mamífero.
17. Un método según la reivindicación 16 en el que dicho organismo es un ser humano.
18. Un método según una cualquiera de las reivindicaciones 10 a 15 en el que los datos que constituyen dicho patrón se proyectan matemáticamente en un modelo de clasificación.
19. Un método según una cualquiera de las reivindicaciones 10 a 18 en el que las células de la muestra no son células enfermas, no han estado en contacto con dichas células y no se originan del sitio de la enfermedad o condición.
20. Un método para identificar sondas útiles para diagnosticar o identificar o monitorizar cáncer de mama o enfermedad de Alzheimer o un estadio de ésta en un organismo, que comprende las etapas de:
a) inmovilizar un conjunto de sondas oligonucleotídicas, como se ha descrito en la reivindicación 1 para cáncer de mama o reivindicación 2 para la enfermedad de Alzheimer en un soporte sólido;
b) aislar el ARNm de una muestra de sangre de un organismo normal (muestra normal), que opcionalmente puede transcribirse de manera inversa a ADNc;
c) aislar el ARNm de una muestra de un organismo, correspondiente a la muestra y organismo de la etapa (b), que se sabe que tiene cáncer de mama o enfermedad de Alzheimer o un estadio de ésta (muestra enferma), que opcionalmente puede transcribirse de manera inversa a ADNc;
d) hibridar el ARNm o ADNc de las etapas (b) y (c) de dicho organismo con cáncer de mama o enfermedad de Alzheimer con dicho conjunto de sondas oligonucleotídicas inmovilizado de la etapa (a) para cáncer de mama o enfermedad de Alzheimer, respectivamente; y
e) evaluar la cantidad de ARNm o ADNc que hibrida con cada una de dichas sondas oligonucleotídicas para determinar el nivel de expresión génica de los genes a los que se unen dichas sondas oligonucleotídicas en dichas muestras normal y enferma para generar un conjunto de datos de expresión génica para cada muestra;
f) normalizar y estandarizar dicho conjunto de datos de la etapa (e);
g) construir un modelo de calibración para la clasificación, preferiblemente usando las técnicas estadísticas Análisis Discriminante de Mínimos Cuadrados Parciales (PLS-DA) y Análisis Discriminante Lineal (LDA);
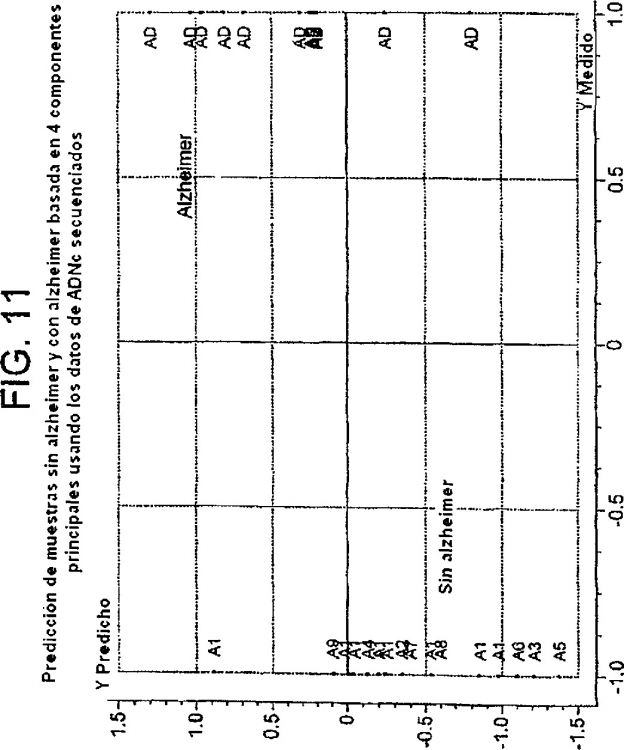
h) realizar un análisis JackKnife e identificar aquellas sondas oligonucleotídicas que se requieren para la clasificación de dichas muestras enfermas y normales en sus grupos respectivos.
Patentes similares o relacionadas:
ENSAYO DE PRIONES, del 8 de Febrero de 2012, de NOVARTIS AG: Un procedimiento de ensayo, que comprende las etapas de: (a) obtener una muestra que se sospecha que contiene una forma patógena de proteína priónica (PrP Sc ) que […]
COMPOSICIONES Y USOS PARA ESTABILIZAR LA TRANSTIRETINA E INHIBIR EL PLEGAMIENTO ANÓMALO DE LA TRANSTIRETINA, del 7 de Febrero de 2012, de THE SCRIPPS RESEARCH INSTITUTE: Diflunisal para uso en el tratamiento de una enfermedad amiloide por transtiretina, en donde el diflunisal se prepara para ser administrado en una cantidad de 250 mg […]
EMPLEO DE INHIBIDORES DE ADAM-17 EN REGENERACIÓN NEURONAL, del 5 de Diciembre de 2011, de UNIVERSIDAD DE CADIZ: La presente invención se refiere al uso de un agente inhibidor de la actividad y/o expresión de ADAM-17 en la preparación de un medicamento, o composición farmacéutica, para […]
PROCEDIMIENTO IN VITRO PARA LA DIAGNOSIS DE ENFERMEDADES NEURODEGENERATIVAS, del 1 de Diciembre de 2011, de B.R.A.H.M.S GMBH: Procedimiento in vitro para la detección, para determinación del grado de severidad y para la valoración evolutiva y la prognosis de enfermedades neurodegenerativas, caracterizado […]
MÉTODO DE DIAGNÓSTICO IN VITRO DE LA ENFERMEDAD DE ALZHEIMER MEDIANTE UN ANTICUERPO MONOCLONAL, del 8 de Noviembre de 2011, de CONSEJO SUPERIOR DE INVESTIGACIONES CIENTIFICAS: Método de diagnóstico in vitro de la enfermedad de Alzheimer medante un anticuerpo monoclonal. Dicho anticuerpo es capaz de unire al menos a los aminoácidos 12-16 del […]
PRODUCTOS DE DEGRADACIÓN PROTEOLÍTICA DE MAP-2 COMO BIOMARCADORES DE DIAGNÓSTICO PARA LAS LESIONES NEURALES, del 2 de Noviembre de 2011, de UNIVERSITY OF FLORIDA RESEARCH FOUNDATION, INC. BANYAN BIOMARKERS: Método in vitro para determinar la presencia de una lesión neural en un sujeto, que comprende detectar por lo menos uno o más productos de degradación proteolítica de una […]
PROCEDIMIENTO DE COMPROBACIÓN Y CONTROL DEL PROCESO DE FERMENTACIÓN DEL ÁCIDO LÁCTICO EN MAMÍFEROS/RUTA METABÓLICA DE FERMENTACIÓN AEROBIA DE LA GLUCOSA EN EL ORGANISMO DE MAMÍFEROS, del 5 de Septiembre de 2011, de COY, JOHANNES, DR.: El uso de la enzima TKTL1 como molécula indicadora y diana para la detección cualitativa y cuantitativa del grado de uso y del flujo correcto del proceso de la ruta […]
MÉTODOS DE TRATAMIENTO DE PSICOSIS Y ESQUIZOFRENIA BASADOS EN POLIMORFISMOS DEL GEN DEL CNTF, del 23 de Agosto de 2011, de NOVARTIS AG: Un método para determinar la capacidad de respuesta de un individuo con un trastorno psicótico al tratamiento con Iloperidona, que comprende; […]