Método para una detección y cuantificación altamente sensible de biomoléculas usando espectrometría de masas de iones secundarios (SIMS).
Un método para detectar y cuantificar en al menos una muestra la presencia o ausencia de al menos una biomolécula,
que comprende:
(a) poner en contacto dicha al menos una muestra con una matriz,
(b) lavar y secar la matriz,
(c) detectar y contar mediante SIMS los iones secundarios comunes junto con los iones secundarios raros correspondientes,
donde dicha una micromatriz comprende un sustrato plano que tiene una superficie conductora y una serie de áreas discretas que contienen sondas que están marcadas con al menos un isótopo raro, estable o inestable o isótopo exógeno, donde dichas sondas se unen a biomoléculas diana presentes en una muestra, y están hechas de cualquier molécula biológica o no biológica.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/IB2009/052735.
Solicitante: BIOSIMS TECHNOLOGIES.
Nacionalidad solicitante: Francia.
Dirección: 75, route de Lyons-la-forêt, Seine Biopolis 2 76000 Rouen FRANCIA.
Inventor/es: RIPOLL,CAMILLE, NORRIS,VICTOR, MISEVIC,GRADIMIR, LEGENT,GUILLAUME, DELAUNE,ANTHONY.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- G01N33/68 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › en los que intervienen proteínas, péptidos o aminoácidos.
PDF original: ES-2505390_T3.pdf



Fragmento de la descripción:
Método para una detección y cuantificación altamente sensible de biomoléculas usando espectrometría de masas de iones secundarios (SIMS) 5
Campo de la invención La invención se refiere a un método para la detección y cuantificación de biomoléculas, tales como ADN, ARN o proteínas, usando marcaje isotópico y espectrometría de masas de iones secundarios, y matrices diseñadas para llevar a cabo dicho método.
Antecedentes de la invención A principios de los sesenta, Castaing y Slodzian desarrollaron la microscopía de emisión de iones de masa filtrada usando iones secundarios, que es parte de una técnica que más tarde se denominó espectrometría de masas de iones secundarios (SIMS) . Con esta técnica, se usa un haz de iones (el haz de iones primarios) como sonda para la pulverización catódica de las capas atómicas superficiales de una muestra en átomos o grupos atómicos, una pequeña fracción de los cuales está ionizada. En un instrumento SIMS, estos iones secundarios se separan de acuerdo con la masa y después se usan para medir una corriente de iones secundarios para crear, por ejemplo, una imagen de la masa atómica cuantitativa de la superficie analizada.
La SIMS se ha convertido en una herramienta principal en estudios de ciencia de semiconductores y superficies, geoquímica, la caracterización de material orgánico, y cosmoquímica. Sin embargo, la microscopía de iones se ha considerado durante mucho tiempo solamente un método marginal para resolver problemas en ciencias de la vida,
debido principalmente a la poca resolución lateral (1-0, 5 μm) y capacidad de separación de masas insuficiente.
Las mejoras tecnológicas y conceptuales condujeron a un progreso significativo tanto en la capacidad de resolución lateral como en la resolución de masas, particularmente debido al uso de un haz de iones primarios finamente ajustado. La microscopía SIMS por lo tanto se ha convertido en una herramienta de captura de imágenes muy potente. Por ejemplo, Lechen et al. fueron capaces de usar la técnica SIMS para tomar imágenes de estereocilios individuales, de los orgánulos mecanosensoriales de las células internas de la cóclea (Lechene et al. Journal of Biology, 2006, 5:20) . En otro experimento, fueron capaces de estudiar la fijación del nitrógeno en bacterias cultivadas en una atmósfera de 15N. El uso de la técnica SIMS también les permitió a Lechene et al. localizar, cuantificar y comparar la fijación de nitrógeno en células únicas y estructuras subcelulares (Lechene et al. Science 2007, 317 (1563) ) . Por lo tanto, la tecnología SIMS se usa ampliamente para tomar imágenes de células o tejidos, y es una herramienta diagnóstica potente.
La técnica SIMS también se usó para detectar la hibridación de ADN no marcado con micromatrices de ácidos nucleicos peptídicos (ANP) (Brandt et al, 2003, Nucleic Acids Research, 31: 19; Brand et al., 2006, Applied Surface Science, 252; 6935) . En estos experimentos, las dobles cadenas ANP/ADN y ANP/ARN se visualizaron mediante SIMS detectando los fosfatos que son una parte integral de los ácidos nucleicos pero que no aparecen en absoluto en los ANP.
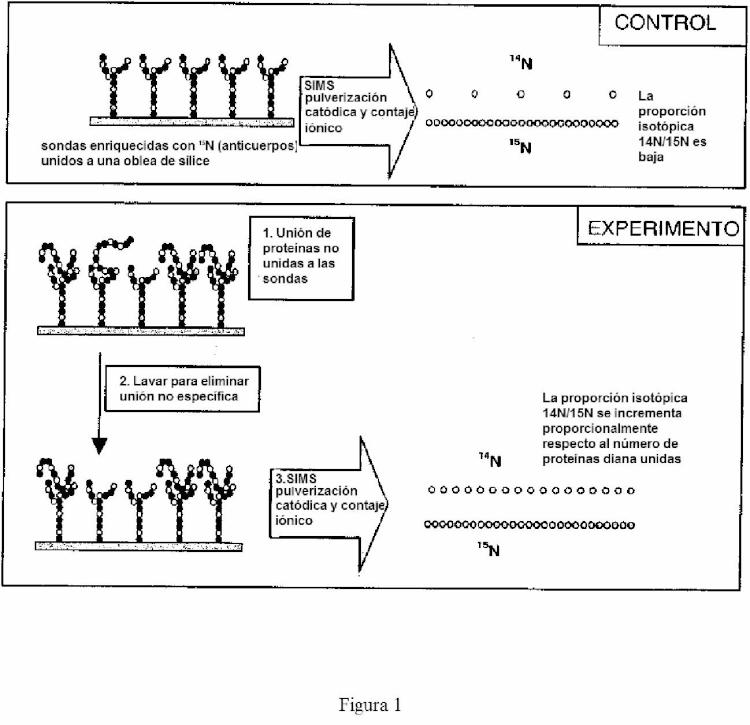
La invención tiene como objetivo proporcionar un método para detectar y cuantificar la presencia o ausencia de una 45 serie de biomoléculas en una muestra usando la técnica SIMS. El método descrito en Brandt et al. presenta los siguientes inconvenientes (i) solo se puede aplicar con sondas ANP o sondas que no contengan fosfatos y (ii) no permite la cuantificación de la interacción sonda/diana. Por lo tanto, los solicitantes tienen como objetivo proporcionar un método universal que se pueda aplicar a un gran número de muestras para la detección y cuantificación de un gran número de interacciones sonda/diana en cada muestra usando la técnica SIMS, determinándose la detección y la cuantificación de dichas interacciones mediante el cálculo de la proporción isotópica sonda/diana (véase Figura 1) :
Sumario de la invención 55 La presente invención usa una matriz que comprende un sustrato plano que tiene una superficie conductora y una serie de áreas discretas que contienen sondas que están marcadas con al menos un isótopo raro, estable o inestable o isótopo exógeno.
De acuerdo con una realización, dicho sustrato plano que tiene una superficie conductora es una oblea de silicio.
De acuerdo con una realización, dichas sondas están marcadas con al menos un isótopo pesado estable seleccionado del grupo 2H, 13C, 15N, 17O, 18O, 33S, 14S y 36S, o con al menos un isótopo inestable seleccionado del grupo 3H y 14C o con al menos un isótopo exógeno seleccionado del grupo de 79Br y 81Br.
De acuerdo con una realización, dicha matriz es una micromatriz donde cada área discreta tiene una de las dimensiones de longitud, anchura o diámetro que es de 1 μm a 1000 μm.
De acuerdo con una realización, cada área discreta es un micropocillo que comprende sondas que están marcadas con al menos un isótopo raro, estable o inestable o un isótopo exógeno.
De acuerdo con una realización, dicha matriz es una nanomatriz donde cada área discreta tiene una de las 5 dimensiones de longitud, anchura o diámetro que es de 1 nm a 1000 nm.
De acuerdo con una realización, cada área discreta es un nanopocillo que comprende sondas que están marcadas con al menos un isótopo raro, estable o inestable o isótopo exógeno.
De acuerdo con una realización, cada micropocillo comprende una serie de nanopocillos, y dichos nanopocillos comprenden las sondas que están marcadas con al menos un isótopo raro, estable o inestable o isótopo exógeno.
La presente invención es un método para detectar y cuantificar en al menos una muestra la presencia o ausencia de al menos una biomolécula, que comprende:
(a) poner en contacto dicha al menos una muestra con una matriz como se ha descrito anteriormente en el presente documento,
(b) lavar y secar la matriz,
(c) detectar y contar mediante SIMS los iones secundarios comunes junto con los iones secundarios raros correspondientes.
De acuerdo con una realización, cada muestra a ensayar se pone en contacto con una o más áreas discretas de la matriz.
De acuerdo con una realización, dicha muestra a ensayar es una única célula.
De acuerdo con una realización, dicho método es para determinar un atlas molecular de la muestra ensayada, donde dicho atlas molecular es la determinación del transcriptoma, proteoma, lipidoma, metaboloma, glicoma y/o interactoma de dicha muestra.
De acuerdo con una realización, dicho método es para predecir una predisposición a una enfermedad, o para diagnosticar una enfermedad en un sujeto que lo necesite.
De acuerdo con una realización, dicho método es para controlar la eficacia de un agente terapéutico administrado a 35 un sujeto para tratar una enfermedad.
De acuerdo con una realización, dicho método es para el cribado de agentes terapéuticos.
Breve descripción de las figuras Figura 1: Ilustración esquemática del principio de la detección SIMS de la unión de las sondas a sus proteínas diana. Las biomoléculas (sondas y dianas) se fragmentan hasta el nivel atómico (condiciones de SIMS dinámica) . Por lo tanto, cada par individual de sonda/diana da cientos de átomos de 15N y 14N (en la forma de iones moleculares secundarios CN-en un experimento real) que se cuentan fácilmente en el analizador SIMS
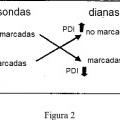
(amplificación) . La medida del incremento de la proporción isotópica 14N/15N permite la cuantificación de aquellas proteínas diana unidas a anticuerpos. Figura 2: incremento o disminución de la PDI dependiendo de si la sonda o la diana están marcadas. Figura 3: fracción isotópica de 15N. Figura 4: fracción isotópica de 13C.
Descripción detallada de la invención
Definiciones 55 • La expresión "ácido nucleico" como se usa en el presente documento significa un polímero compuesto de nucleótidos, por ejemplo, desoxirribonucleótidos o ribonucleótidos, o compuestos producidos sintéticamente tales como los ANP que pueden hibridar con ácidos nucleicos de origen natural de un modo específico de secuencia análogo al de los dos ácidos nucleicos de origen natural, por ejemplo, pueden participar en interacciones de emparejamiento de bases de Watson y Crick.
• La expresión "ácido ribonucleico" y el término "ARN" como se usan en el presente documento significan un polímero compuesto de ribonucleótidos,
• La expresión "ácido desoxirribonucleico" y el término "ADN" como se usan en el presente documento significan un polímero compuesto de desoxirribonucleótidos,
• El término "oligonucleótido"... [Seguir leyendo]
Reivindicaciones:
1. Un método para detectar y cuantificar en al menos una muestra la presencia o ausencia de al menos una biomolécula, que comprende: 5
(a) poner en contacto dicha al menos una muestra con una matriz,
(b) lavar y secar la matriz,
(c) detectar y contar mediante SIMS los iones secundarios comunes junto con los iones secundarios raros correspondientes,
donde dicha una micromatriz comprende un sustrato plano que tiene una superficie conductora y una serie de áreas discretas que contienen sondas que están marcadas con al menos un isótopo raro, estable o inestable o isótopo exógeno, donde dichas sondas se unen a biomoléculas diana presentes en una muestra, y están hechas de cualquier molécula biológica o no biológica.
2. Un método de acuerdo con la reivindicación 1, donde dicho sustrato plano que tiene una superficie conductora es una oblea de silicio.
3. Un método de acuerdo con las reivindicaciones 1 o 2, donde dichas sondas están marcadas con al menos un
isótopo pesado estable seleccionado del grupo 2H, 13C, 15N, 17O, 18O, 33S, 34S y 36S, o con al menos un isótopo inestable seleccionado del grupo 3H y 14C o con al menos un isótopo exógeno seleccionado del grupo de 79Br y 81Br.
4. Un método de acuerdo con una cualquiera de las reivindicaciones de 1 a 3, donde dicha matriz es una micromatriz donde cada área discreta tiene una de las dimensiones de longitud, anchura o diámetro que son de 1 μma
5. Un método de acuerdo con la reivindicación 4, donde cada área discreta es un micropocillo que comprende sondas que están marcadas con al menos un isótopo raro, estable o inestable o un isótopo exógeno.
7. Un método de acuerdo con la reivindicación 6, donde cada área discreta es un nanopocillo que comprende sondas que están marcadas con al menos un isótopo raro, estable o inestable o un isótopo exógeno. 35
8. Un método de acuerdo con la reivindicación 5, donde cada micropocillo comprende una serie de nanopocillos, y dichos nanopocillos comprenden las sondas que están marcadas con al menos un isótopo raro, estable o inestable o un isótopo exógeno.
9. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 8, donde cada muestra a ensayar se pone en contacto con una o más áreas discretas de la matriz.
10. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 9, donde dicha muestra a ensayar es una única célula. 45
11. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 10, donde dichas biomoléculas diana están marcadas con al menos un isótopo raro, estable o inestable o un isótopo exógeno.
12. El método de acuerdo con una cualquiera de las reivindicaciones 9 a 11 para determinar un atlas molecular de la
muestra ensayada, donde dicho atlas molecular es la determinación del transcriptoma, proteoma, lipidoma, metaboloma, glicoma y/o interactoma de dicha muestra.
13. El método de acuerdo con una cualquiera de las reivindicaciones 9 a 11 para predecir una predisposición a una enfermedad, o para diagnosticar una enfermedad en un sujeto que lo necesite. 55
14. El método de acuerdo con una cualquiera de las reivindicaciones 9 a 11 para controlar la eficacia de un agente terapéutico administrado a un sujeto para tratar una enfermedad.
15. El método de acuerdo con una cualquiera de las reivindicaciones 9 a 11 para el cribado de agentes terapéuticos. 60
Patentes similares o relacionadas:
Inmunomoduladores, del 29 de Julio de 2020, de BRISTOL-MYERS SQUIBB COMPANY: Un compuesto de la fórmula (I) **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo, en donde: A se selecciona de **(Ver fórmula)** en donde: […]
Métodos y composiciones para el diagnóstico y pronóstico de lesión renal e insuficiencia renal, del 29 de Julio de 2020, de Astute Medical, Inc: Un método para evaluar el estado renal en un sujeto, que comprende: realizar una pluralidad de ensayos configurados para detectar una […]
Neuregulina para tratar la insuficiencia cardíaca, del 29 de Julio de 2020, de Zensun (Shanghai) Science & Technology, Co., Ltd: Neuregulina para usar en un método para tratar la insuficiencia cardíaca crónica en un paciente, donde el paciente tiene un nivel plasmático de NT-proBNP […]
Procedimiento para evaluación de la función hepática y el flujo sanguíneo portal, del 15 de Julio de 2020, de The Regents of the University of Colorado, a body corporate: Procedimiento in vitro para la estimación del flujo sanguíneo portal en un individuo a partir de una única muestra de sangre o suero, comprendiendo el procedimiento: […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Método para llevar a cabo el seguimiento de la enfermedad de Gaucher, del 15 de Julio de 2020, de Centogene GmbH: Un método para determinar la evolución de la enfermedad de Gaucher en un sujeto, que comprende la etapa de determinar en varios puntos en el […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos de monitorización terapéutica de profármacos de ácido fenilacético, del 24 de Junio de 2020, de Immedica Pharma AB: Glicerilo tri-[4-fenilbutirato] (HPN-100) para su uso en un método para tratar un trastorno del ciclo de la urea en un sujeto que tiene discapacidad […]