Factor de predicción de supervivencia para linfoma difuso de células B grandes.
Método de predicción del resultado de supervivencia basándose en una o más muestras de linfoma difuso de células B grandes (DLBCL),
método que comprende:
a) aislar productos de expresión génica de una o más muestras de biopsia de DLBCL de un sujeto tratado con un régimen terapéutico que incluye quimioterapia y rituximab (R-CHOP);
b) obtener un perfil de expresión génica a partir de los productos de expresión génica, en el que el perfil de expresión comprende un nivel de expresión para cada uno de al menos 31 de los genes enumerados en la tabla 1 para la firma de expresión génica de células B de centro germinal (GCB) y al menos 226 de los genes enumerados en la tabla 1 para la firma de expresión génica estromal-1;
c) determinar un valor de firma de GCB y un valor de firma estromal-1 a partir del perfil de expresión génica, en el que el valor de firma de GCB es un promedio de los niveles de expresión de los al menos 31 genes enumerados en la tabla 1 para la firma de expresión génica de GCB y el valor de firma de estromal-1 es un promedio de los niveles de expresión de los al menos 226 genes enumerados en la tabla 1 para la firma de expresión génica estromal-1;
d) calcular una puntuación de factor de predicción de supervivencia ≥ A - [(x)*(el valor de firma de GCB)] - [(y)*(el valor de firma de estromal-1)], en la que A es un término de desviación, (x) es un factor de escala de entre 0,200 y 0,625, e (y) es un factor de escala de entre 0,800 y 1,250, y
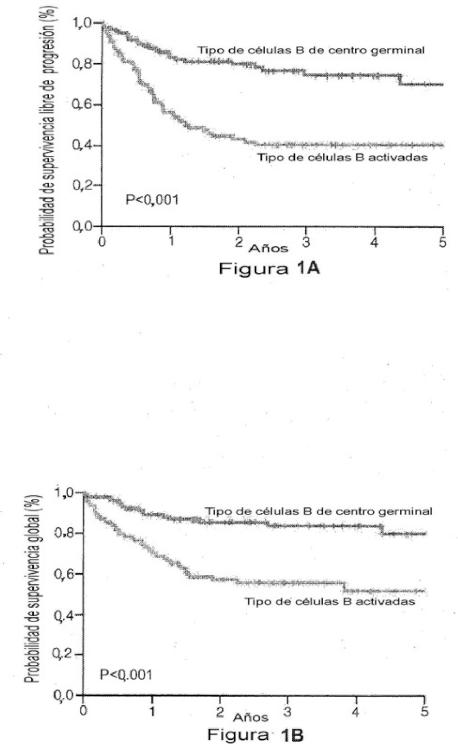
e) determinar el resultado de supervivencia para el sujeto basándose en la puntuación de factor de predicción de supervivencia calculada en la etapa d) para cualquier valor igual fijo de A y para cualquier valor igual fijo de (x) e (y), en el que una puntuación inferior de factor de predicción de supervivencia indica un resultado de supervivencia más favorable y una puntuación superior de factor de predicción de supervivencia indica un resultado de supervivencia menos favorable para el sujeto.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2009/046421.
Solicitante: The United States Of America, As Represented By The Secretary, Dept. Of Health And Human Services.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: Office Of Technology Transfer National Insitutes of Health 6011 Executive Boulevard Suite 325, MSC 7660 Bethesda, MD 20892-7660 ESTADOS UNIDOS DE AMERICA.
Inventor/es: MULLER-HERMELINK, HANS KONRAD, WRIGHT, GEORGE, OTT,GERMAN, ROSENWALD,ANDREAS, LOPEZ GUILLERMO,ARMANDO, STAUDT,LOUIS M, RIMSZA,LISA, LISTER,ANDREW T, WEISENBURGER,DENNIS, DELABIE,JAN, SMELAND,ERLEND B, HOLTE,HARALD, KVALOY,STEIN, BRAZIEL,RITA M, FISHER,RICHARD I, JARES,PEDRO, GUERRI,ELÍAS CAMPO, JAFFE,ELAINE S, LENZ,GEORG, WYNDHAM,H.WILSON, DAVE,SANDEEP S, GASCOYNE,RANDY D, CONNORS,JOSEPH M, WING,C CHAN.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- G01N33/574 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › para el cáncer.
PDF original: ES-2552937_T3.pdf



Patentes similares o relacionadas:
Composiciones para la supresión del cáncer por inhibición de TMCC3, del 29 de Julio de 2020, de Development Center For Biotechnology: Un anticuerpo contra la proteína con dominios de superhélice transmembrana 3 (TMCC3), o un fragmento de unión de la misma, en donde el anticuerpo […]
Métodos para detectar distintivos de enfermedades o afecciones en fluidos corporales, del 15 de Julio de 2020, de PRESIDENT AND FELLOWS OF HARVARD COLLEGE: Un método in vitro para diagnosticar la presencia de un agente infeccioso en un individuo que comprende los pasos de: obtener un primer perfil de expresión […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos para clasificar pacientes con un cáncer sólido, del 24 de Junio de 2020, de INSERM (INSTITUT NATIONAL DE LA SANTE ET DE LA RECHERCHE MEDICALE): Un método in vitro para el pronóstico del tiempo de supervivencia de un paciente que padece un cáncer sólido, cuyo método comprende los siguientes […]
Procedimiento de detección del cáncer, del 24 de Junio de 2020, de Sienna Cancer Diagnostics Ltd: La presente invención proporciona un procedimiento para resolver una evaluación citológica no concluyente de células epiteliales de vejiga en […]
Método para el diagnóstico/pronóstico de cáncer colorrectal, del 17 de Junio de 2020, de CONSEJO SUPERIOR DE INVESTIGACIONES CIENTIFICAS (CSIC): La presente invención se refiere a un método de obtención de datos útiles para el diagnóstico, el pronóstico o la monitorización de la evolución de cáncer colorrectal (CCR), […]
Criterio de valoración terapéutico equivalente para inmunoterapia de enfermedades basada en antiCTLA-4, del 10 de Junio de 2020, de E. R. Squibb & Sons, L.L.C: Un anticuerpo antiCTLA-4 para su uso en el tratamiento de cáncer en un sujeto, tratamiento que comprende inducir un acontecimiento liminar […]
Método de deducción de un valor de positividad de biomarcador en porcentaje para células seleccionadas presentes en un campo de visión, del 10 de Junio de 2020, de NOVARTIS AG: Método de deducción de un valor para el % de positividad de biomarcador (PBP) para todas las células u, opcionalmente, uno o más subconjuntos de las […]