VARIANTES DE LA MEGANUCLEASA I-CREI CON ESPECIFICIDAD MODIFICA, METODO DE PREPARACION Y USOS DE LAS MISMAS.
Método de preparación de una variante de meganucleasa I-CreI que tiene una especificidad de escisión modificada,
comprendiendo dicho método: en la que: ES 2 347 684 T3 (a) reemplazar los aminoácidos Q44, R68 y/o R70, en referencia al código de acceso pdb de I-CreI lg9y, con un aminoácido seleccionado del grupo que consiste en A, D, E, G, H, K, N, P, Q, R, S, T e Y; (b) seleccionar las variantes de meganucleasa I-CreI obtenidas en el paso (a) que tienen al menos uno de los siguientes perfiles de escisión de tripletes R3 en referencia a las posiciones -5 a -3 en una diana de ADN bicatenario, correspondiendo dichas posiciones -5 a -3 a R3 de la siguiente fórmula I: 5'- R1CAAAR2R3R4R'4R'3R'2TTTGR'1, -3', R1 está ausente o presente; y cuando está presente representa un fragmento de ácido nucleico que comprende de 1 a 9 nucleótidos que corresponden a una secuencia de ácido nucleico aleatoria o a un fragmento del sitio de movimiento dirigido de meganucleasas I-CreI situado desde la posición -20 a -12 (de 5' a 3'), correspondiendo R1 al menos a la posición -12 de dicho sitio de movimiento dirigido, R2 representa el doblete de ácido nucleico ac o ct y corresponde a las posiciones -7 a -6 de dicho sitio de movimiento dirigido, R3 representa un triplete de ácido nucleico que corresponde a dichas posiciones -5 a -3, seleccionadas de entre g, t, c y a, excepto los siguientes tripletes: gtc, gcc, gtg, gtt y gct, R4 representa el doblete de ácido nucleico gt o tc y corresponde a las posiciones -2 a -1 de dicho sitio de movimiento dirigido, R'1 está ausente o presente; y cuando está presente representa un fragmento de ácido nucleico que comprende de 1 a 9 nucleótidos que corresponden a una secuencia de ácido nucleico aleatoria o a un fragmento de un sitio de movimiento dirigido de meganucleasa I-CreI situado desde la posición +12 a +20 (de 5' a 3'), correspondiendo R'1 al menos a la posición +12 de dicho sitio de movimiento dirigido, R'2 representa el doblete de ácido nucleico ag o gt y corresponde a las posiciones +6 a +7 de dicho sitio de movimiento dirigido, R'3 representa un triplete de ácido nucleico que corresponde a dichas posiciones +3 a +5, seleccionadas de entre g, t, c y a; siendo R'3 diferente de gac, ggc, cac, aac y agc, cuando R3 y R'3 no son palindrómicos, R'4 representa el doblete de ácido nucleico ga o ac y corresponde a las posiciones +1 a +2 de dicho sitio de movimiento dirigido
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/IB2006/001203.
Solicitante: CELLECTIS.
Nacionalidad solicitante: Francia.
Dirección: 102, ROUTE DE NOISY, 93235 ROMAINVILLE CEDEX.
Inventor/es: PAQUES,FREDERIC, DUCHATEAU,PHILIPPE.
Fecha de Publicación: .
Fecha Concesión Europea: 5 de Mayo de 2010.
Clasificación Internacional de Patentes:
- C12N9/22 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 9/00 Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas. › Ribonucleasas.
Clasificación PCT:
- C12N15/10 C12N […] › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/55 C12N 15/00 […] › Hidrolasas (3).
- C12N15/64 C12N 15/00 […] › Métodos generales para la preparación del vector, para su introducción en la célula o para la selección del huésped que contiene el vector.
- C12N15/79 C12N 15/00 […] › Vectores o sistemas de expresión especialmente adaptados a huéspedes eucariotas.
- C12N15/90 C12N 15/00 […] › Introducción estable de ADN extraño en el cromosoma.
- C12N9/22 C12N 9/00 […] › Ribonucleasas.
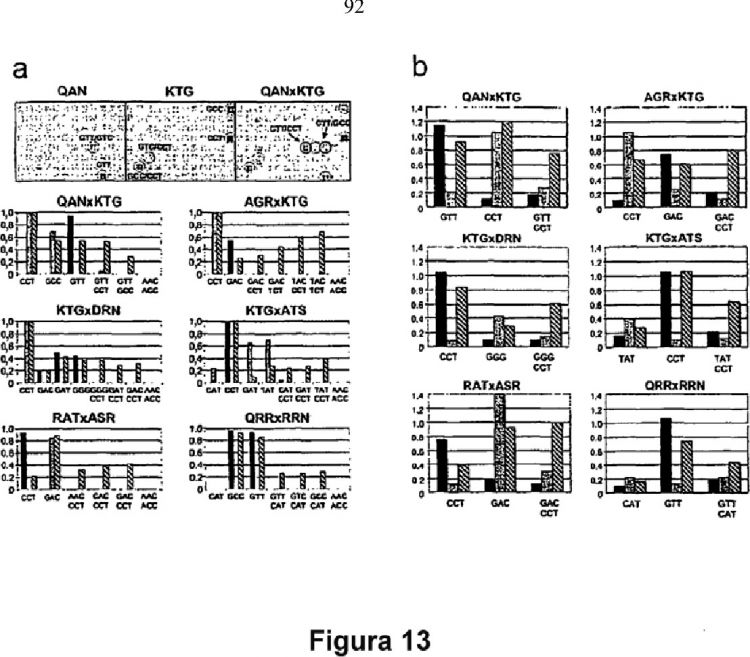
Fragmento de la descripción:
La presente invención se refiere a un método para preparar variantes de meganucleasa I-CreI que tienen una especificidad de escisión modificada. La invención se refiere también a las variantes de meganucleasa I-CreI que pueden obtenerse mediante dicho método y a sus aplicaciones tanto para la escisión de una nueva diana de ADN como para ingeniería genética e ingeniería genómica para propósitos no terapéuticos.
La invención también se refiere a ácidos nucleicos que codifican dichas variantes, a los casetes de expresión que comprenden dichos ácidos nucleicos, a los vectores que comprenden dichos casetes de expresión, a células u organismos, plantas o animales excepto seres humanos, transformados mediante dichos vectores.
Las meganucleasas son endonucleasas específicas de secuencia que reconocen sitios de escisión de ADN grandes (>12pb; habitualmente 14-40 pb) (Thierry y Dujon, 1992). En la naturaleza, las meganucleasas están representadas esencialmente por endonucleasas de movimiento dirigido, generalmente codificadas por elementos genéticos móviles tales como inteínas e intrones de clase I (Belfort y Roberts, 1997; Chevalier y Stoddard, 2001). El movimiento dirigido se refiere a la movilización de estos elementos, que se basa en la reparación de una ruptura de doble cadena de ADN (DSB), iniciada por la actividad endonucleasa de la meganucleasa. Estudios tempranos de las proteínas HO (Haber, 1998; Klar et al., 1984; Kostriken et al., 1983), I-SceI (Colleaux et al., 1988; Jacquier y Dujon, 1985; Perrin et al., 1993; Plessis et al., 1992) e I-TevI (Bell-Pedersen et al., 1990; Bell-Pedersen et al., 1989; Bell-Pedersen et al., 1991; Mueller et al., 1996) han ilustrado la biología del proceso de movimiento dirigido. Por otro lado, estos estudios también han proporcionado un paradigma para el estudio de la reparación de DSB en células vivas.
La asimetría general de las secuencias diana de endonucleasa de movimiento dirigido contrasta con la simetría de díada característica de la mayoría de los sitios de reconocimiento de enzimas de restricción. Se ha demostrado que varias endonucleasas de movimiento dirigido codificadas por ORF de intrones o inteínas promueven el movimiento dirigido de sus elementos genéticos respectivos a sitios alélicos sin intrones o sin inteínas. Al realizar una rotura de doble cadena específica de sitio en los alelos sin intrón y sin inteína, estas nucleasas crean extremos recombinantes, que entablan un proceso de conversión génica que duplica la secuencia codificante y conduce a la inserción de un intrón o una secuencia intercalada a nivel de ADN.
Las endonucleasas de movimiento dirigido se incluyen en 4 familias separadas en base a motivos aminoacídicos muy bien conservados [para revisión, véase Chevalier y Stoddard (Nucleic Acids Research, 2001, 29, 3757-3774)]. Una de ellas es la familia de los dodecapéptidos (dodecámero, DOD, D1D2, LAGLIDADG, P1-P2). Esta es la familia más grande de proteínas agrupadas por su motivo de secuencia conservada más general: una o dos copias (amplia mayoría) de una secuencia de doce restos: el dodecapéptido. Las endonucleasas de movimiento dirigido con un dodecapéptido (D) tienen alrededor de 20 kDa de masa molecular y funcionan como homodímeros. Las que tienen dos copias (DD) varían desde 25 kDa (230 aminoácidos) a 50 kDa (HO, 545 aminoácidos) con de 70 a 150 restos entre cada motivo y actúan como un monómero. La escisión se da dentro del sitio de reconocimiento, dejando un corte escalonado de 4 nucleótidos con salientes 3'OH. Las enzimas que contienen una copia única del motivo LAGLIDADG, tales como I-CeuI e I-CreI actúan como homodímeros y reconocen un sitio de movimiento dirigido casi palindrómico.
La secuencia y la estructura de la endonucleasa de movimiento dirigido I-CreI (código de acceso pdb 1g9y) se ha determinado (Rochaix JD et al., NAR, 1985, 13, 975-984; Heath PJ et al., Nat. Struct. Biol., 1997, 4, 468-476; Wang et al., NAR, 1997, 25, 3767-3776; Jurica et al. Mol. Cell, 1998, 2, 469-476) y se han generado modelos estructurales usando cristalografía de rayos X (Heath et al., 1997).
I-CreI comprende 163 aminoácidos (código de acceso pdb 1g9y); dicha endonucleasa se corta como un dímero. El motivo LAGLIDADG corresponde a los restos 13 a 21; a cada lado de las αhélices LAGLIDADG, lámina β cuádruple (posiciones 21-29; 37-48; 66-70 y 73-78) proporciona una interfaz de unión a ADN que dirige la interacción de la proteína con el semisitio de la secuencia de ADN diana. La interfaz de dimerización implica a las dos hélices LAGLIDADG así como otros restos.
El sitio de movimiento dirigido reconocido y escindido por I-CreI es de 22-24 pb de longitud y es un palíndromo degradado (véase figura 2 de Jurica MS et al, 1998 y SEC ID Nº:65). De manera más precisa, el sitio de movimiento dirigido de dicha I-CreI es una secuencia semi-palindrómica de 22 pb, con 7 de 11 pb idénticos en cada semisitio (Seligman LM et al., NAR, 2002, 30,3870-3879).
La interfaz de endonucleasa-ADN también se ha descrito (véase figura 4 de Jurica MS et al, 1998) y ha conducido a varias predicciones acerca de contactos específicos proteína-ADN (Seligman LM et al., Genetics, 1997, 147, 1653-1664; Jurica MS et al., 1998; Chevalier B. et al., Biochemistry, 2004, 43, 14015-14026).
Surge de dichos documentos que: - los restos G19, D20, Q47, R51, K98 y D137 son parte del sitio endonucleolítico de I-CreI; - las secuencias de sitio de movimiento dirigido deben tener al menos 20 pb para conseguir una afinidad de enlace máxima de 0,2 nM; - los contactos específicos de secuencia se distribuyen a lo largo de la longitud completa del sitio de movimiento dirigido; - las sustituciones de pares de bases pueden tolerarse en muchas posiciones diferentes del sitio de movimiento dirigido, sin perturbar seriamente la unión o escisión del sitio de movimiento dirigido; - R51 y K98 se localizan en el sitio de activo de la enzima y son candidatos para actuar como ácido de Lewis o para activar un donante de protones en la región de escisión; las mutaciones en cada uno de estos restos se ha observado que reducen en gran medida la actividad endonucleolítica de I-CreI (R51G, K98Q);- cinco restos adicionales, que cuando mutan suprimen la actividad endonucleasa de I-CreI se
localizan en o cerca del sitio activo de enzima (R70A, L39R, L91R, D75G, Q47H).
Estos estudios han preparado el terreno para un uso general de meganucleasa para ingeniería genómica. La dirección génica homologa es la forma más precisa para modificar de forma estable un locus cromosómico en células vivas, pero su baja eficacia sigue siendo un inconveniente principal. Puesto que la DSB inducida por meganucleasa estimula la recombinación homóloga hasta
10.000 veces, las meganucleasas son actualmente la mejor manera para mejorar la eficacia de la inserción génica dirigida en células mamíferas (Choulika et al., 1995; Cohen-Tannoudji et al., 1998; Donoho et al., 1998; Elliott et al., 1998; Rouet et al., 1994), y para llevarlas a eficacias viables en organismos tales como plantas (Puchta et al., 1993; Puchta et al., 1996) e insectos (Rong y Golic, 2000; Rong y Golic, 2001; Rong et al., 2002).
Las meganucleasas se han usado para inducir diversos tipos de sucesos de recombinación homóloga, tales como recombinación de repetición directa en células de mamífero (Liang et al., 1998), plantas (Siebert y Puchta, 2002), insectos (Rong et al., 2002), y bacterias (Posfai et al., 1999), o recombinación intercromosómica (Moynahan y Jasin, 1997; Puchta, 1999; Richardson et al., 1998).
Sin embargo, esta tecnología aun está limitada por el bajo número de sitios diana naturales potenciales para meganucleasas: aunque se han identificado varios cientos de endonucleasas de movimiento dirigido naturales (Belfort y Roberts, 1997; Chevalier y Stoddard, 2001), la probabilidad de tener una meganucleasa natural que escinda un gen de interés es extremadamente baja. La realización de meganucleasas artificiales con especificidades dedicadas evitaría esta limitación.
Se han hecho endonucleasas artificiales con nuevas especificidades, basándose en la fusión de dominios de endonucleasas a dominios de unión a ADN de dedo de cinc (Bibikova et al., 2003; Bibikova et al., 2001; Bibikova et al., 2002; Porteus y Baltimore, 2003).
Las endonucleasas de movimiento dirigido también se han usado como armazones para realizar endonucleasas nuevas, bien mediante fusión de diferentes dominios de proteínas (Chevalier et al., 2002; Epinat et al., 2003)...
Reivindicaciones:
1. Método de preparación de una variante de meganucleasa I-CreI que tiene una especificidad de escisión modificada, comprendiendo dicho método:
(a) reemplazar los aminoácidos Q44, R68 y/o R70, en referencia al código de acceso pdb de I-CreI lg9y, con un aminoácido seleccionado del grupo que consiste en A, D, E, G, H, K, N, P, Q, R, S, T e Y;
(b) seleccionar las variantes de meganucleasa I-CreI obtenidas en el paso (a) que tienen al menos uno de los siguientes perfiles de escisión de tripletes R3 en referencia a las posiciones -5 a -3 en una diana de ADN bicatenario, correspondiendo dichas posiciones -5 a -3 a R3 de la siguiente fórmula I:
5'- R1CAAAR2R3R4R'4R'3R'2TTTGR'1, -3', en la que: R1 está ausente o presente; y cuando está presente representa un fragmento de ácido nucleico que comprende de 1 a 9 nucleótidos que corresponden a una secuencia de ácido nucleico aleatoria o a un fragmento del sitio de movimiento dirigido de meganucleasas I-CreI situado desde la posición -20 a -12 (de 5' a 3'), correspondiendo R1 al menos a la posición -12 de dicho sitio de movimiento dirigido, R2 representa el doblete de ácido nucleico ac o ct y corresponde a las posiciones -7 a -6 de dicho sitio de movimiento dirigido, R3 representa un triplete de ácido nucleico que corresponde a dichas posiciones -5 a -3, seleccionadas de entre g, t, c y a, excepto los siguientes tripletes: gtc, gcc, gtg, gtt y gct, R4 representa el doblete de ácido nucleico gt o tc y corresponde a las posiciones -2 a -1 de dicho sitio de movimiento dirigido, R'1 está ausente o presente; y cuando está presente representa un fragmento de ácido nucleico que comprende de 1 a 9 nucleótidos que corresponden a una secuencia de ácido nucleico aleatoria o a un fragmento de un sitio de movimiento dirigido de meganucleasa I-CreI situado desde la posición +12 a +20 (de 5' a 3'), correspondiendo R'1 al menos a la posición +12 de dicho sitio de movimiento dirigido, R'2 representa el doblete de ácido nucleico ag o gt y corresponde a las posiciones +6 a +7 de dicho sitio de movimiento dirigido, R'3 representa un triplete de ácido nucleico que corresponde a dichas posiciones +3 a +5, seleccionadas de entre g, t, c y a; siendo R'3 diferente de gac, ggc, cac, aac y agc, cuando R3 y R'3 no son palindrómicos, R'4 representa el doblete de ácido nucleico ga o ac y corresponde a las posiciones +1 a +2 de dicho sitio de movimiento dirigido.
2. Método de acuerdo con la reivindicación 1, caracterizado por que dicho triplete R3 de ácido nucleico se selecciona preferiblemente de entre los siguientes tripletes: ggg., gga, ggt, ggc, gag, gaa, gat, gac, gta, gcg, gca, tgg, tga, tgt, tgc, tag, taa, tat, tac, ttg, tta, ttt, ttc, tcg, tca, tct, tcc, agg, aga, agt, agc,
aag, aaa, aat, aac, atg, ata, att, atc, acg, aca, act, acc, cgg, cga, cgt, cgc, cag, caa, cat, cac, ctg, cta, ctt, ctc, ccg, cca, cct y ccc y más preferiblemente de entre los siguientes tripletes: ggg., ggt, ggc, gag, gat, gac, gta, gcg, gca, tag, taa, tat, tac, ttg, ttt, ttc, tcg, tct, tcc, agg, aag, aat, aac, att, atc, act, acc, cag, cat, cac, ctt, ctc, ccg, cct y ccc.
3. Método de acuerdo con la reivindicación 1 o reivindicación 2, caracterizado por que la variante de meganucleasa I-CreI obtenida en el paso (b) se selecciona del grupo que consiste en:: A44/A68/A70, A44/A68/G70,A44/A68/H70,A44/A68/K70,A44/A68/N70,A44/A68/Q70,A44/G68/H70,A44/H68/H70,A4 4/K68/A70,A44/K68/570,A44/N68/N70,A44/Q68/G70,A44/R68/E70,A44/R68/S70,A44/S68/R70,A44/T6 8/N70,D44/R68/A70,E44/H68/H70,G44/H68/K70,H44/A68/S70,H44/R68/R70,H44/T68/T70,K44/A68/Q 70,K44/E68/S70,K44/H68/E70,K44/K68/H70,K44/N68/N70,K44/Q68/E70,K44/R68/H70,K44/S68/H70, K44/T68/G70,N44/A68/R70,N44/N68/R70,N44/R68/G70,N44/S68/G70,N44/T68/R70,Q44/G68/K70,A44 /A68/R70,A44/G68/K70,A44/H68/K70,A44/K68/G70,A44/K68/T70,A44/N68/Q70,A44/Q68/H70,A44/R 68/G70,A44/R68/T70,A44/S68/S70,A44/T68/Q70,D44/R68/K70,E44/R68/A70,G44/Q68/H70,H44/A68/T 70H44/R68/S70,K44/A68/A70,K44/A68/S70,K44/G68/A70,K44/H68/G70,K44/K68/T70,K44/N68/Q70; K44/Q68/S70,K44/R68/N70,K44/S68/N70,K44/T68/H70,N44/H68/N70,N44/P68/D70,N44/R68/H70,N44 /S68/H70,N44/T68/S70,Q44/G68/R70,A44/A68/S70, A44/G68/N70, A44/H68/N70, A44/K68/H70, A44/N68/A70, A44/N68/R70, A44/Q68/N70, A44/R68/H70, A44/S68/A70, A44/S68/T70, A44/T68/R70, D44/R68/N70, E44/R68/H70, G44/R68/Q70, H44/R68/A70, H44/R68/T70, K44/A68/D70, K44/A68/T70, K44/G68/G70, K44/H68/N70, K44/N68/A70, K44/N68/S70, K44/Q68/T70, K44/R68/Q70, K44/S68/S70, K44/T68/N70, N44/H68/R70, N44/Q68/H70, N44/R68/K70, N44/S68/K70, P44/N68/D70, Q44/K68/G70,A44/A68/T70, A44/G68/P70, A44/H68/Q70, A44/K68/K70, A44/N68/E70, A44/N68/S70, A44/Q68/R70, A44/R68/K70, A44/S68/G70, A44/T68/A70, A44/T68/S70, D44/R68/Q70, E44/R68/N70, G44/R68/R70, H44/R68/D70, H44/S68/G70, K44/A68/E70, K44/D68/A70, K44/G68/N70, K44/H68/S70, K44/N68/D70, K44/N68/T70, K44/R68/A70, K44/R68/S70, K44/S68/T70, K44/T68/Q70, N44/K68/G70, N44/Q68/R70, N44/R68/N70, N44/S68/R70, P44/T68/T70,A44/D68/H70, A44/G68/R70, A44/H68/R70, A44/K68/N70, A44/N68/G70, A44/N68/T70, A44/Q68/S70, A44/R68/L70, A44/S68/K70, A44/T68/G70, A44/T68/T70, D44/R68/R70, E44/R68/S70, G44/T68/D70, H44/R68/E70, H44/S68/S70, K44/A68/G70, K44/D68/T70, K44/G68/S70, K44/H68/T70, K44/N68/E70, K44/P68/H70, K44/R68/D70, K44/R68/T70, K44/T68/A70, K44/T68/S70, N44/K68/H70, N44/R68/A70, N44/R68/R70, N44/T68/H70, Q44/A68/A70,Q44/N68/A70, Q44/N68/H70,A44/D68/K70, A44/H68/A70, A44/H68/S70, A44/K68/Q70, A44/N68/H70, A44/Q68/A70, A44/R68/A70, A44/R68/N70, A44/S68/N70, A44/T68/H70, D44/D68/H70, D44/R68/S70, E44/R68/T70, G44/T68/P70, H44/R68/G70, H44/S68/T70, K44/A68/H70, K44/E68/G70, K44/G68/T70, K44/K68/A70, K44/N68/G70, K44/Q68/A70, K44/R68/E70, K44/S68/A70, K44/T68/D70, K44/T68/T70, N44/K68/R70, N44/R68/D70, N44/R68/S70, N44/T68/K70, Q44/A68/H70, Q44/N68/S70,A44/D68/R70, A44/H68/G70, A44/H68/T70, A44/K68/R70, A44/N68/K70, A44/Q68/D70, A44/R68/D70, A44/R68/R70, A44/S68/Q70, A44/T68/K70, D44/N68/S70, D44/R68/T70, E44/S68/T70,
G44/T68/R70, H44/R68/N70, H44/T68/S70, K44/A68/N70, K44/E68/N70, K44/H68/D70, K44/K68/D70, K44/N68/H70, K44/Q68/D70, K44/R68/G70, K44/S68/D70, K44/T68/E70, N44/A68/H70, N44/K68/S70, N44/R68/E70, N44/R68/T70, N44/T68/Q70, Q44/A68/R70, Q44/P68/P70,Q44/Q68/G70, Q44/R68/Q70, Q44/T68/H70, R44/N68/T70, R44/R68/S70, S44/H68/R70, T44/H68/R70, T44/R68/D70,Q44/R68/A70, Q44/R68/S70, Q44/T68/R70, R44/R68/A70, R44/R68/T70, S44/R68/G70, T44/K68/R70, T44/R68/E70,Q44/R68/D70, Q44/S68/H70, R44/A68/G70, R44/R68/D70, R44/S68/G70, S44/R68/N70, T44/N68/P70, T44/R68/G70,Q44/R68/E70, Q44/S68/R70, R44/A68/T70, R44/R68/E70, R44/S68/N70, S44/R68/R70, T44/N68/R70, T44/R68/H70,Q44/R68/G70, Q44/S68/S70, R44/G68/T70, R44/R68/G70, R44/S68/S70, S44/R68/S70, T44/Q68/K70, T44/R68/K70,Q44/R68/H70, Q44/T68/A70, R44/H68/D70, R44/R68/N70, R44/S68/T70, T44/A68/K70, T44/Q68/R70, T44/R68/N70,Q44/R68/N70, Q44/T68/G70, R44/H68/T70, R44/R68/Q70, S44/D68/K70, T44/A68/R70, T44/R68/A70, T44/R68/Q70,T44/R68/R70, T44/R68/S70, T44/R68/T70, T44/S68/K70, T44/S68/R70, T44/T68/K70, y T44/T68/R70.
4. Método de acuerdo con una cualquiera de las reivindicaciones 1 a 3, caracterizado por que la etapa (b) de selección de dicha variante de meganucleasa I-CreI se realiza in vivo en células de levadura.
5. Uso de una variante de meganucleasa I-CreI, obtenible por el método de acuerdo con una cualquiera de las reivindicaciones 1 a 4, in vitro o in vivo para propósitos no terapéuticos, para la escisión de una diana de ácido nucleico bicatenaria que comprende al menos una secuencia parcialmente palindrómica de 20-24 pb, en la que al menos la secuencia en las posiciones +/- 8 a 11 es palindrómica y el triplete de nucleótidos en las posiciones -5 a -3 y/o el triplete de nucleótidos de las posiciones +3 a +5 difiere de gtc, gcc, gtg, gtt y gct y de gac, ggc, cac, aac y agc, respectivamente.
6. El uso de acuerdo con la reivindicación 5, caracterizado por que dicha variante de meganucleasa I-CreI se selecciona del grupo que consiste en A44/A68/A70, A44/A68/G70, A44/A68/H70, A44/A68/K70, A44/A68/N70, A44/A68/Q70, A44/A68/R70, A44/A68/S70, A44/A68/T70, A44/D68/H70, A44/D68/K70, A44/D68/R70, A44/G68/H70, A44/G68/K70, A44/G68/N70, A44/G68/P70, A44/G68/R70, A44/H68/A70, A44/H68/G70, A44/H68/H70, A44/H68/K70, A44/H68/N70, A44/H68/Q70, A44/H68/R70, A44/H68/S70, A44/H6870, A44/K68/A70, A44/K68/G70, A44/K68/H70, A44/K68/K70, A44/K68/N70, A44/K68/Q70, A44/K68/R70, A44/K68/S70,A44/K68/T70,A44/N68/A70,A44/N68/K70,A44/Q68/D70,A44/R68/D70,A44/R68/R70,A4 4/S68/Q70,A44/T68/K70,D44/N68/S70,D44/R68/T70,A44/N68/E70,A44/N68/S70,A44/Q68/R70,A44/R6 8/K70,A44/S68/G70,A44/T68/A70,A44/T68/S70,D44/R68/Q70,E44/R68/N70,G44/R68/R70,H44/R8/D70 ,H44/S68/G70,K44/A68/E70,K44/D68/A70,K44/G68/N70,K44/H68/S70,K44/N68/D70,K44/N68/T70,K4 4/R68/A70,K44/R68/S70,K44/S68/T70,K44/T68/Q70,N44/K68/G70,N44/Q68/R70,N44/R68/N70,N44/S6 8/R70,P44/T68/T70,Q44/N68/A70,Q44/R68/E70,Q44/S68/R70,R44/A68/T70,R44/R68/E70,R44/S68/N70 ,A44/N68/G70,A44/N68/T70,A44/Q68/S70,A44/R68/L70,A44/S68/K70,A44/T68/G70,A44/T68/T70,D44
/R68/R70,E44/R68/S70,G44/T68/D70,H44/R68/E70,H44/S68/S70,K44/A68/G70,K44/D68/T70,K44/G68/ S70,K44/H68/T70,K44/N68/E70,K44/P68/H70,K44/R68/D70,K44/R68/T70,K44/T68/A70,K44/T68/S70, N44/K68/H70,N44/R68/A70,N44/R68/R70,N44/T68/H70,A44/N68/H70, A44/Q68/A70, A44/R68/A70, A44/R68/N70, A44/S68/N70, A44/T68/H70, D44/D68/H70, D44/R68/S70, E44/R68/T70, G44/T68/P70, H44/R68/G70, H44/S68/T70, K44/A68/H70, K44/E68/G70, K44/G68/T70, K44/K68/A70, K44/N68/G70, K44/Q68/A70, K44/R68/E70, K44/S68/A70, K44/T68/D70, K44/T68/T70, N44/K68/R70, N44/R68/D70, N44/R68/S70, N44/T68/K70,A44/N68/Q70, A44/Q68/H70, A44/R68/G70, A44/R68/T70, A44/S68/S70, A44/T68/Q70, D44/R68/K70, E44/R68/A70,Q44/A68/A70, Q44/A68/H70,Q44/N68/H70, Q44/N68/S70,Q44/R68/G70,Q44/S68/S70,R44/G68/T70,R44/R68/G70,R44/S68/S70,A44/N68/N70,A44/ Q68/G70,A44/R68/E70,A44/R68/S70,A44/S68/R70,A44/T68/N70,D44/R68/A70,E44/H68/H70,E44/S68/ T70, G44/H68/K70, G44/Q68/H70,G44/T68/R70, H44/A68/S70, H44/A68/T70,H44/R68/N70, H44/R68/R70, H44/R68/S70,H44/T68/S70, H44/T68/T70, K44/A68/A70,K44/A68/N70, K44/A68/Q70, K44/A68/S70,K44/E68/N70, K44/E68/S70, K44/G68/A70,K44/H68/D70, K44/H68/E70, K44/H68/G70,K44/K68/D70, K44/K68/H70, K44/K68/T70,K44/N68/H70, K44/N68/N70, K44/N68/Q70,K44/Q68/D70, K44/Q68/E70, K44/Q68/S70,K44/R68/G70, K44/R68/H70, K44/R68/N70,K44/S68/D70, K44/S68/H70, K44/S68/N70,K44/T68/E70, K44/T68/G70, K44/T68/H70,N44/A68/H70, N44/A68/R70, N44/H68/N70,N44/K68/S70, N44/N68/R70, N44/P68/D70,N44/R68/E70, N44/R68/G70, N44/R68/H70,N44/R68/T70, N44/S68/G70, N44/S68/H70,N44/T68/Q70, N44/T68/R70, N44/T68/S70,Q44/A68/R70, Q44/G68/K70, Q44/G68/R70,Q44/P68/P70, Q44/Q68/G70, Q44/R68/A70,Q44/R68/H70, Q44/R68/N70, Q44/R68/Q70, Q44/R68/S70,Q44/T68/A70, Q44/T68/G70, Q44/T68/H70, Q44/T68/R70,R44/H68/D70, R44/H68/T70, R44/N68/T70, R44/R68/A70,R44/R68/N70, R44/R68/Q70, R44/R68/S70, R44/R68/T70,R44/S68/T70, S44/D68/K70, S44/H68/R70, S44/R68/G70,A44/N68/R70, A44/Q68/N70, A44/R68/H70, A44/S68/A70, A44/S68/T70, A44/T68/R70, D44/R68/N70, E44/R68/H70, G44/R68/Q70, H44/R68/A70, H44/R68/T70, K44/A68/D70, K44/A68/T70, K44/G68/G70, K44/H68/N70, K44/N68/A70, K44/N68/S70, K44/Q68/T70, K44/R68/Q70, K44/S68/S70, K44/T68/N70, N44/H68/R70, N44/Q68/H70, N44/R68/K70, N44/S68/K70, P44/N68/D70, Q44/K68/G70, Q44/R68/D70, Q44/S68/H70, R44/A68/G70, R44/R68/D70, R44/S68/G70, S44/R68/N70,S44/R68/R70, S44/R68/S70, T44/A68/K70, T44/A68/R70, T44/H68/R70, T44/K68/R70, T44/N68/P70,T44/N68/R70, T44/Q68/K70, T44/Q68/R70, T44/R68/A70, T44/R68/D70, T44/R68/E70, T44/R68/G70,T44/R68/H70, T44/R68/K70, T44/R68/N70, T44/R68/Q70, T44/R68/R70, T44/R68/S70, T44/R68/T70, T44/S68/K70, T44/S68/R70, T44/T68/K70.
7. El uso de acuerdo con la reivindicación 5 o con la reivindicación 6 caracterizado por que dicha variante de meganucleasa I-CreI es un homodímero.
8. El uso de acuerdo con una cualquiera de las reivindicaciones 5 y 6, caracterizado por que dicha variante de meganucleasa I-CreI es un heterodímero.
9. El uso de acuerdo con una cualquiera de las reivindicaciones 5 a 8, caracterizado por que la secuencia de posiciones +/- 3 a 5 de dicha diana de ADN es palindrómica.
10. El uso, de acuerdo con una cualquiera de las reivindicaciones 5 a 6, caracterizada por que la secuencia en las posiciones +/- 3 a 5 de dicha diana de ADN es no palindrómica.
11. El uso de acuerdo con una cualquiera de las reivindicaciones 5 a 10, caracterizado por que dichas secuencias palindrómicas en las posiciones -11 a -8 y +8 a +11 son caaa y tttg, respectivamente.
12. El uso de acuerdo con una cualquiera de las reivindicaciones 5 a 11, caracterizado por que dicha variante de meganucleasa I-CreI comprende adicionalmente una mutación en la posición 75.
13. El uso de acuerdo con la reivindicación 12, caracterizada por que dicha mutación es D75N o D75V.
14. El uso de acuerdo con una cualquiera de las reivindicaciones 5 a 13, caracterizado por que dicha variante de meganucleasa I-CreI tiene una alanina (A) o una asparagina (N) en la posición 44 para escindir una diana de ADN que comprende un nucleótido a en una posición -4 y/o t en posición +4.
15. El uso de acuerdo con una cualquiera de las reivindicaciones 5 a 13, en el que dicha variante de meganucleasa tiene una lisina (K) en la posición 44 para escindir una diana que comprende un nucleótido c en la posición -4 y/o g en la posición +4.
16. El uso de acuerdo con una cualquiera de las reivindicaciones 6 a 13, en el que dicha variante de meganucleasa tiene una glutamina (Q) en la posición 44 para escindir una diana que comprende un nucleótido t en la posición -4 y/o a en la posición +4.
17. Una variante de meganucleasa I-CreI, obtenible por el método de preparación de acuerdo con las reivindicaciones 1 a 4; que se selecciona del grupo que consiste en: A44/A68/A70, A44/A68/G70, A44/A68/H70, A44/A68/K70, A44/A68/N70, A44/A68/Q70, A44/A68/S70, A44/A68/T70, A44/G68/N70, A44/G68/P70, A44/H68/Q70, A44/H68/S70, A44/K68/Q70, A44/K68/R70, A44/N68/H70, A44/N68/K70, A44/Q68/A70, A44/Q68/D70, A44/R68/K70, A44/R68/L70, A44/S68/S70, A44/S68/T70, A44/T68/S70, A44/T68/T70, D44/R68/R70, D44/R68/S70, E44/R68/570, E44/R68/T70, G44/T68/P70, G44/T68/R70, H44/R68/N70, H44/R68/R70, H44/T68/T70, K44/A68/A70, K44/A68/Q70, K44/D68/A70, K44/G68/N70, K44/G68/S70, K44/H68/S70, K44/H68/T70, K44/N68/D70, K44/N68/E70, K44/N68/T70, K44/P68/H70, K44/R68/A70, K44/R68/D70, K44/S68/A70, K44/S68/D70, K44/T68/D70, K44/T68/E70,A44/D68/H70, A44/H68/A70, A44/H68/T70, A44/K68/S70, A44/N68/N70, A44/Q68/G70, A44/S68/A70, A44/T68/A70, D44/D68/H70, D44/R68/T70, E44/S68/T70, H44/A68/S70, H44/R68/S70, K44/A68/D70, K44/D68/T70,
K44/G68/T70, K44/K68/A70, K44/N68/G70, K44/Q68/A70, K44/R68/E70, K44/S68/H70, K44/T68/G70,A44/D68/K70, A44/H68/G70, A44/K68/A70, A44/K68/T70, A44/N68/Q70, A44/Q68/H70, A44/S68/G70, A44/T68/G70,A44/N68/S70, E44/H68/H70,G44/H68/K70, H44/A68/T70, H44/S68/G70, K44/A68/E70, K44/E68/G70, K44/H68/D70, K44/K68/D70, K44/N68/H70, K44/Q68/D70, K44/R68/G70, K44/S68/N70, K44/T68/H70,A44/D68/R70, A44/H68/H70, A44/K68/G70, A44/N68/A70, A44/N68/R70, A44/Q68/N70, A44/S68/N70, A44/T68/H70, D44/R68/A70, E44/R68/A70, G44/Q68/H70, H44/R68/D70, H44/S68/S70, K44/A68/G70, K44/E68/S70, K44/H68/E70, K44/K68/H70, K44/N68/N70, K44/Q68/E70, K44/R68/H70, K44/S68/S70, K44/T68/N70,A44/G68/H70, A44/H68/K70, A44/K68/H70, A44/N68/E70, A44/N68/S70, A44/Q68/S70, A44/S68/Q70, A44/T68/N70, D44/R68/N70, E44/R68/H70, G44/R68/Q70, H44/R68/E70, H44/S68/T70, K44/A68/H70, K44/G68/A70, K44/H68/G70, K44/K68/T70, K44/N68/Q70, K44/Q68/S70, K44/R68/N70, K44/S68/T70, K44/T68/Q70,A44/G68/K70, A44/H68/N70, A44/K68/N70, A44/N68/G70, A44/N68/T70, A44/R68/E70, A44/S68/R70, A44/T68/Q70, D44/R68/Q70, E44/R68/N70, G44/T68/D70, H44/R68/G70, H44/T68/S70, K44/A68/N70, K44/G68/G70, K44/H68/N70, K44/N68/A70, K44/N68/S70, K44/Q68/T70, K44/R68/S70, K44/T68/A70, K44/T68/S70,K44/T68/T70, N44/A68/H70, N44/K68/S70, N44/P68/D70, N44/S68/G70, N44/S68/H70, N44/T68/S70, P44/N68/D70, Q44/N68/H70, Q44/N68/S70, Q44/R68/Q70, Q44/S68/S70, R44/G68/T70, R44/H68/D70, R44/R68/G70, R44/R68/Q70, R44/S68/T70, S44/D68/K70, T44/R68/E70, T44/R68/Q70, y T44/S68/K70.
18. La variante de megaucleasa I-CreI de acuerdo con la reivindicación 17, que tiene una alanina (A) o una asparagina (N) en la posición 44 y escinde una diana que comprende un nucleótido a en la posición -4 y/o t en la posición +4.
19. La variante de megaucleasa I-CreI de acuerdo con la reivindicación 17, que tiene una lisina (K) en la posición 44 y escinde una diana que comprende un nucleótido c en la posición -4 y /o g en la posición +4.
20. La variante de megaucleasa I-CreI de acuerdo con la reivindicación 17, que tiene una glutamina (Q) en la posición 44 y escinde una diana que comprende un nucleótido t en la posición -4 y/o a en la posición +4.
21. La variante de meganucleasa I-CreI de acuerdo con una cualquiera de las reivindicaciones 17 a 20, que es un homodímero.
22. La variante de meganucleasa I-CreI de acuerdo con una cualquiera de las reivindicaciones 17 a 20, que es un heterodímero que consiste en dos monómeros, cada uno de una variante diferente como se define en una cualquiera de las reivindicaciones 17 a 20.
23. Un polinucleótido, caracterizado por que codifica una variante de meganucleasa I-CreI de acuerdo con una cualquiera de las reivindicaciones 17 a 22.
24. Un casete de expresión, que comprende un polinucleótido de acuerdo con la reivindicación 23 y las secuencias de regulación.
25. Un vector de expresión, caracterizado por que comprende un casete de expresión de acuerdo con la reivindicación 24.
26. Un vector de expresión, de acuerdo con la reivindicación 25, caracterizado por que comprende adicionalmente una construcción de ADN diana.
27. Un vector de expresión, de acuerdo con la reivindicación 26, caracterizado por que dicha construcción de ADN diana comprende una secuencia que comparte homologías con la región que rodea el sitio de escisión de la variante de meganucleasa I-CreI de acuerdo con una cualquiera de las reivindicaciones 17 a 22.
28. El vector de expresión, de acuerdo con la reivindicación 27 caracterizado por que dicha
construcción de ADN diana comprende: a) secuencias que comparten homologías con la región que rodea el sitio de escisión de la variante de acuerdo con las reivindicaciones 17 a 22, y b) secuencias a introducir flanqueadas por una secuencia como en a).
29. Una célula, caracterizada por que está modificada por un polinucleótido de acuerdo con la reivindicación 23 o por un vector de acuerdo con una cualquiera de las reivindicaciones 25 a 28.
30. Una planta transgénica, caracterizada por que comprende un polinucleótido de acuerdo con la reivindicación 23 o un vector de acuerdo con una cualquiera de las reivindicaciones 25 a 28.
31. Un mamífero transgénico no humano, caracterizado por que comprende un polinucleótido de acuerdo con la reivindicación 23 o un vector de acuerdo con una cualquiera de las reivindicaciones 25 a 28.
32. Uso de una variante de meganucleasa I-CreI de acuerdo con una cualquiera de las reivindicaciones 17 a 22, un polinucleótido de acuerdo con la reivindicación 23, un vector de acuerdo con una cualquiera de las reivindicaciones 25 a 28, una célula de acuerdo con la reivindicación 29, una planta transgénica de acuerdo con la reivindicación 30, un mamífero transgénico no humano de acuerdo con la
reivindicación 31, para biología molecular, para ingeniería genética in vivo o in vitro y para ingeniería genómica in vivo o in vitro.
33. El uso de acuerdo con la reivindicación 32, para inducir una ruptura de ácido nucleico de doble cadena en un sitio de interés que comprende una secuencia diana de ADN, induciendo de este modo un suceso de recombinación de ADN, una pérdida de ADN o muerte celular.
34. El uso, de acuerdo con la reivindicación 32 o la reivindicación 33, caracterizado por que dicha rotura de ácido nucleico de doble cadena es para: reparar una secuencia específica, modificar una secuencia específica, restaurar un gen funcional en lugar de un gen mutado, atenuar o activar un gen endógeno de interés, introducir una mutación en un sitio de interés, introducir un gen exógeno o una parte del mismo, inactivar o suprimir un gen endógeno o una parte del mismo, traslocar un brazo cromosómico o dejar el ADN sin reparación y degradado.
35. El uso de acuerdo con una cualquiera de las reivindicaciones 32 a 34, caracterizado por que dicha variante de meganucleasa I-CreI, polinucleótido, vector, célula, planta transgénica o mamífero transgénico no humano se asocia con una construcción de ADN diana como se ha definido en la reivindicación 26.
36. El método de ingeniería genética, caracterizado por que comprende un paso de rotura de ácido nucleico de doble cadena en un sitio de interés localizado en un vector, que comprende una diana de ADN de una variante de meganucleasa de I-CreI de acuerdo con las reivindicaciones 17 a 22 mediante el contacto de dicho vector con una variante de meganucleasa I-CreI como se ha definido anteriormente, induciendo de este modo una recombinación homóloga con otro vector que presenta homología con la secuencia que rodea el sitio de escisión de dicha variante de meganucleasa I-CreI.
37. Un método de ingeniería genómica, caracterizado por que comprende los siguientes pasos: 1) rotura de doble cadena de un locus genómico que comprende al menos un sitio de reconocimiento y escisión de una variante de meganucleasa I-CreI de acuerdo con las reivindicaciones 17 a 22, mediante el contacto de dicho sitio de escisión con dicha variante de meganucleasa I-CreI; 2) mantener dicho locus genómico roto en condiciones apropiadas para recombinación homóloga con una construcción de ADN diana que comprende la secuencia a introducir en dicho locus, flanqueada por secuencias que comparten homologías con el locus diana.
38. Un método de ingeniería genómica, caracterizado por que comprende los siguientes pasos: 1) rotura de doble cadena de un locus genómico que comprende al menos un sitio de reconocimiento y escisión de una variante de meganucleasa I-CreI de acuerdo con las reivindicaciones 17 a 22, mediante el contacto de dicho sitio de escisión con dicha variante de meganucleasa I-CreI; 2) mantener dicho locus
genómico roto en condiciones apropiadas para recombinación homóloga con ADN cromosómico que comparte homologías con regiones que rodean el sitio de escisión.
39. Una composición caracterizada por que comprende al menos una variante de meganucleasa I-CreI de acuerdo con las reivindicaciones 17 a 22, un polinucleótido de acuerdo con la reivindicación 23 o un vector de acuerdo con una cualquiera de las reivindicaciones 25 a 28.
40. La composición de acuerdo con la reivindicación 39, caracterizada por que dicha composición comprende adicionalmente una construcción de ADN diana que comprende la secuencia que repara el sitio de interés flanqueado por secuencias que comparten homologías con el locus diana.
41. Uso de al menos una variante de meganucleasa I-CreI de acuerdo con las reivindicaciones 17 a 22, un polinucleótido de acuerdo con la reivindicación 23 o un vector de acuerdo con una cualquiera de las reivindicaciones 25 a 28 para la preparación de un medicamento para la prevención, mejora o cura de una enfermedad genética en un individuo que lo necesite, administrándose dicho medicamento por cualquier medio a dicho individuo.
42. Uso de al menos una variante de meganucleasa I-CreI de acuerdo con las reivindicaciones 17 a 22, un polinucleótido de acuerdo con la reivindicación 23 o un vector de acuerdo con una cualquiera de las reivindicaciones 25 a 28, para la preparación de un medicamento para prevenir, mejorar o curar una enfermedad causada por un agente infeccioso que presenta un intermedio de ADN en un individuo que lo necesite, administrándose dicho medicamento mediante cualquier medio a dicho individuo.
43. Uso de al menos una variante de meganucleasa I-CreI de acuerdo con las reivindicaciones 17 a 22, un polinucleótido de acuerdo con la reivindicación 23 o un vector de acuerdo con una cualquiera de las reivindicaciones 25 a 28, in vitro, para inhibir la propagación, inactivar o suprimir un agente infeccioso que presenta un intermedio de ADN, en productos derivados biológicos o productos dirigidos a usos biológicos o para desinfectar un objeto.
44. El uso de acuerdo con una cualquiera de las reivindicaciones 41 a 43, caracterizado por que dicho agente infeccioso es un virus.
Patentes similares o relacionadas:
Procedimientos y composiciones para el tratamiento de una afección genética, del 24 de Junio de 2020, de Sangamo Therapeutics, Inc: Una célula precursora de glóbulos rojos genomanipulada caracterizada por una modificación genómica dentro del exón 2 o el exón 4 de BCL11A o dentro de BCL11A-XL […]
Métodos y composiciones para ingeniería genómica, del 3 de Junio de 2020, de Sangamo Therapeutics, Inc: Una pareja de nucleasas de dedo de zinc (ZFN) que comprende una ZFN izquierda y una ZFN derecha, comprendiendo cada ZFN un dominio de escisión […]
Métodos y composiciones para el tratamiento de enfermedades por almacenamiento lisosomal, del 27 de Mayo de 2020, de Sangamo Therapeutics, Inc: Uno o más transgenes y una o más nucleasas con dedos de zinc (ZFN) para su uso en un método de tratamiento de una enfermedad por almacenamiento lisosomal, el método […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Métodos y composiciones para modular PD1, del 13 de Mayo de 2020, de Sangamo Therapeutics, Inc: Célula aislada que comprende una inserción o una deleción en un gen de PD1 endógeno dentro de, o entre, las secuencias mostradas en SEQ ID NO: 56 y SEQ ID NO: 60 del gen de PD1 […]
Cepas bacterianas que expresan genes de metilasa y sus usos, del 22 de Abril de 2020, de LOMA LINDA UNIVERSITY: Una bacteria aislada para usar en la producción de ADN plasmídico metilado, en donde la bacteria comprende un polinucleótido exógeno que codifica una CpG metilasa […]
Método y composiciones para reducir productos de amplificación no específicos, del 25 de Marzo de 2020, de Paragon Genomics, Inc: Un método para reducir productos de amplificación no específicos de una reacción de extensión de cebadores dependiente de plantilla, comprendiendo el método: […]
Proteínas que tienen actividad nucleasa, proteínas de fusión y usos de estas, del 18 de Marzo de 2020, de HELMHOLTZ ZENTRUM MUNCHEN DEUTSCHES FORSCHUNGSZENTRUM FUR GESUNDHEIT UND UMWELT (GMBH): Una molécula de ácido nucleico que codifica (I) un polipéptido que tiene la actividad de una endonucleasa, que es (a) una molécula de ácido nucleico que […]