Sonda de ácido nucleico lineal de doble hebra y usos de la misma.
Un método para cuantificar ARN en una muestra de ensayo, comprendiendo el método
(A) mezclar la muestra de ensayo con los reactivos de amplificación,
los reactivos de transcripción inversa y unasonda de ácido nucleico que comprende un primer ácido oligonucleico y el segundo ácido oligonucleico, donde(I)
a) el primer ácido oligonucleico tiene 30 - 34 bases (m) de longitud y tiene una homología de 80% o máscon un ácido nucleico de interés, y comprende adicionalmente un fluoróforo;
b) el segundo ácido oligonucleico tiene n bases de longitud donde n está relacionado con m de manera quecuando m es de 30 a 34, n es de 8 a 15; y comprende adicionalmente un extintor;
c) el primer ácido oligonucleico y, el segundo ácido oligonucleico se caracterizan porque:
i) el segundo ácido oligonucleico tiene una homología de 80% o más con el primer ácido oligonucleico, y
ii) el primer ácido oligonucleico y el segundo ácido oligonucleico se pueden unir entre sí en ausencia delácido nucleico de interés en condiciones apropiadas para formar un ácido nucleico de doble hebra demanera que la emisión de fluorescencia del fluoróforo anclado al primer ácido oligonucleico cuando elprimer ácido oligonucleico está unido al segundo ácido oligonucleico es detectable menos que la emisióndel mismo fluoróforo cuando el primer ácido oligonucleico y el segundo ácido oligonucleico no estánunidos entre sí, o
(II)a) el primer ácido oligonucleico tiene 30 - 34 bases (m) de longitud y tiene una homología de 80% o máscon un ácido nucleico de interés, y comprende un extintor,
b) el segundo ácido oligonucleico tiene n bases de longitud donde n está relacionado con m de manera quecuando m es de 30 a 34, n es de 8 a 15; y comprende un fluoróforo;
c) el primer ácido oligonucleico y el segundo ácido oligonucleico se caracterizan porque:
i) el segundo ácido oligonucleico tiene una homología de 80% o más con primer ácido oligonucleico, yii) el primer ácido oligonucleico y el segundo ácido oligonucleico se pueden unir entre sí en ausencia delácido nucleico de interés en condiciones apropiadas para formar un ácido nucleico de doble hebra demanera que la emisión fluorescente del fluoróforo anclado al segundo ácido oligonucleico cuando elprimer ácido oligonucleico está unido con respecto al segundo ácido oligonucleico es detectable menosque la emisión del mismo fluoróforo cuando el primer ácido oligonucleico y el segundo ácido oligonucleicono están unidos entre sí, para crear una mezcla en un recipiente de reacción,
(B) colocar la muestra de ensayo en condiciones permisivas de transcripción inversa, que opcionalmentetambién pueden ser permisivas de amplificación, de manera que se produce un ADNc,
(C) someter a ciclo térmico la mezcla de ensayo de manera que se amplifique el ADNc, y
(D) medir la fluorescencia de la mezcla de ensayo durante la reacción de amplificación como una indicación dela cantidad de ARN de la muestra de ensayo.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2004/038129.
Solicitante: ABBOTT LABORATORIES.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: DEPT. 377, BLDG AP6A-1 100 ABBOTT PARK ROAD ABBOTT PARK, IL 60064-6008 ESTADOS UNIDOS DE AMERICA.
Inventor/es: HACKETT, JOHN, R., JR., SALITURO, JOHN, A., ABRAVAYA,KLARA, MORRISON,Larry,E, HUANG,Shihai, LUK,Ka-Cheung.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07H21/04 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07H AZUCARES; SUS DERIVADOS; NUCLEOSIDOS; NUCLEOTIDOS; ACIDOS NUCLEICOS (derivados de ácidos aldónicos o sacáricos C07C, C07D; ácidos aldónicos, ácidos sacáricos C07C 59/105, C07C 59/285; cianohidrinas C07C 255/16; glicales C07D; compuestos de constitución indeterminada C07G; polisacáridos, sus derivados C08B; ADN o ARN concerniente a la ingeniería genética, vectores, p. ej. plásmidos o su aislamiento, preparación o purificación C12N 15/00; industria del azúcar C13). › C07H 21/00 Compuestos que contienen al menos dos unidades mononucleótido que tienen cada una grupos fosfato o polifosfato distintos unidos a los radicales sacárido de los grupos nucleósido, p. ej. ácidos nucleicos. › con desoxirribosilo como radical sacárido.
- C12Q1/68 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2385485_T3.pdf
Fragmento de la descripción:
Sonda de Ácido Nucleico Lineal de Doble Hebra y Usos de la Misma.
Campo de la Invención
La invención se refiere generalmente al campo de la amplificación y detección de ácidos nucleicos. Adicionalmente, la invención se refiere a composiciones y métodos para realizar la PCR y la hibridación con sondas utilizando una única mezcla de reactivos.
Antecedentes
Los análisis basados en ADN se utilizan rutinariamente en una amplia gama de escenarios, incluyendo la hematología clínica, la genética molecular, la microbiología y la inmunología. Muchas técnicas actuales cuentan con la amplificación mediante PCR de un polinucleótido de interés (en adelante "molécula diana") junto con varios tipos de técnicas de detección post-amplificación. Otros mecanismos de amplificación no basados en PCR son bien conocidos en la técnica incluyendo, pero no limitados a, análisis de ligación de oligonucleótidos (OLA) , reacción en cadena de la ligasa (LCR) , amplificación mediada por transcripción (TMA) , y amplificación con desplazamiento de la hebra (SDA) . Adicionalmente, estas técnicas son susceptibles de mezclado. Esto es, el producto de una reacción de amplificación se puede utilizar como la diana de otra reacción de amplificación, lo que permite una gran sensibilidad con una etapa adicional que tiende a aumentar la sensibilidad.
Un formato de amplificación preferido es conocido como análisis homogéneo en tiempo real. Un análisis en tiempo real es aquel que produce datos indicativos de la presencia o cantidad de una molécula diana durante el proceso de amplificación, en oposición al final del proceso de amplificación. Un análisis homogéneo es aquel en el que los reactivos de amplificación y detección se mezclan entre sí y se ponen en contacto simultáneamente con una muestra, que puede contener una molécula de ácido nucleico diana. De este modo, la capacidad para detectar y cuantificar dianas de ADN en sistemas homogéneos en tiempo real a medida de que la amplificación avanza se centra en análisis de un solo tubo en los cuales los procesos requeridos para la amplificación y la detección de la molécula diana tienen lugar en un formato de reacción en un único "tubo cerrado". Por ejemplo, las técnicas actuales que utilizan la amplificación mediante PCR y que tienen estas características son conocidas generalmente como técnicas de PCR en Tiempo Real. De un modo similar, las tecnologías no basadas en la PCR también están dentro del conocimiento práctico del experto normal en la técnica y son aptas para métodos de detección homógeneos.
En la mayor parte de las técnicas de amplificación y detección se utiliza una sonda para detectar un producto de amplificación. Varios sistemas de sondas conocidos en la técnica utilizan un fluoróforo y un extintor. Por ejemplo, las sondas baliza moleculares son sondas de ácidos oligonucleicos de hebra sencilla que pueden formar una estructura en horquilla en la cual un fluoróforo y un extintor están colocados normalmente en los extremos opuestos del oligonucleótido. En cualquier extremo de la sonda secuencias complementarias cortas permiten la formación de un tallo intramolecular, que permite que el fluoróforo y el extintor se acerquen suficientemente. La porción de bucle de la baliza molecular es complementaria a un ácido nucleico diana de interés. La unión de esta sonda a su ácido nucleico diana de interés forma un híbrido que mantiene el tallo alejado. Esto ocasiona un cambio de conformación que separa el fluoróforo y el extintor entre sí y conduce a una señal fluorescente más intensa. Las sondas baliza moleculares son, sin embargo, muy sensibles a una pequeña variación en la secuencia en la sonda diana (Tyagi S. y Kramer F.R., Nature Biotechnology, Vol. 14, páginas 303-308 (1996) ; Tyagi et al., Nature Biotechnology, Vol. 16, páginas 49-53 (1998) ; Piatek et al., Nature Biotechnology, Vol.16, páginas 359-363 (1998) ; Marras S. et al., Genetic Analysis: Biomolecular Engineering. Vol. 14, páginas 151-156 (1999) ; Täpp I. et al, BioTechniques, Vol 28, páginas 732-738 (2000) ) .
A diferencia de las sondas baliza moleculares, algunas sondas lineales de hebra sencilla que poseen también un extintor y un fluoróforo unidos a extremos opuestos de un oligonucleótido no forman una estructura en horquilla. En lugar de eso, esta clase de sondas de oligonucleótidos lineales en solución se comportan como una espiral al azar, sus dos extremos se aproximan entre sí ocasionalmente, dando como resultado un cambio medible en la transferencia de energía. Sin embargo, cuando la sonda se une a su diana, el híbrido sonda-diana mantiene alejados los dos extremos de la sonda, interrumpiendo la interacción entre los dos radicales terminales, y restaurando de este modo la señal fluorescente del fluoróforo. Además, las sondas lineales de hebra sencilla se pueden diseñar como "sondas TaqMan", que se unen a hebras de la diana durante la PCR y de este modo pueden ser escindidas enzimáticamente por la actividad exonucleasa 5'º 3' de la ADN polimerasa Taq durante la fase de extensión del cebador del ciclo de la PCR dando como resultado un incremento en la fluorescencia en cada ciclo proporcional a la cantidad de producto específico generado. Se ha informado de que las sondas lineales de hebra sencilla largas adolecen de señales de "fondo" elevadas, mientras las más cortas son sensibles a emparejamientos erróneos de una sola base (Lee L.G. et al., Nucleic Acids Research, Vol. 21, páginas 3761-3766 (1993) ; Täpp I. et al. (más arriba) ; Patente de los Estados Unidos Núm. 6.258.569; Patente de los Estados Unidos Núm. 6.030.787) .
Las sondas lineales de doble hebra también son conocidas en la técnica. Las sondas lineales de doble hebra tienen dos oligonucleótidos complementarios. Las sondas descritas en la técnica anterior tenían la misma longitud, donde al menos uno de los oligonucleótidos actúa como sonda para una secuencia diana en una conformación de hebra sencilla. El extremo 5' de uno de los oligonucleótidos está marcado con un fluoróforo y el extremo 3' del otro oligonucleótido está marcado con un extintor, p. ej., un fluoróforo aceptor, o vice versa. Cuando estos dos oligonucleótidos se recuecen entre sí, las dos marcas están próximas entre sí, extinguiendo de ese modo la fluorescencia. Los ácidos nucleicos diana, sin embargo, compiten por la unión a la diana, dando como resultado un incremento de la fluorescencia de la sonda inferior al que proporcionalmente se esperaría con la concentración creciente de ácido nucleico diana (Morrison L. et al., Anal. Biochem., Vol. 183, páginas 231-244 (1989) ; Patente de los Estados Unidos Núm. 5.928.862) .
También se conocen en la técnica sondas lineales de doble hebra modificadas por acortamiento de uno de los dos oligonucleótidos complementarios en unas pocas bases para elaborar una sonda lineal de doble hebra parcial. En dichas sondas lineales de doble hebra de la técnica anterior, el oligonucleótido más largo ha sido marcado en el extremo con un fluoróforo y el oligonucleótido ligeramente más corto ha sido marcado en el extremo con un extintor. En la forma de doble hebra, la sonda es menos fluorescente debido a la íntima proximidad del fluoróforo y el extintor. En presencia de una diana, sin embargo, el oligonucleótido extintor más corto es desplazado por la diana. Como resultado, el oligonucleótido más largo (en forma de un híbrido sonda-diana) se vuelve sustancialmente más fluorescente.
Las sondas de doble hebra conocidas en la técnica anterior que tienen oligonucleótidos de longitudes desiguales presentan una discriminación completa entre una diana perfectamente emparejada y dianas con emparejamientos erróneos de un solo nucleótido. Asimismo, estas sondas no tienen cinéticas de reacción óptimas especialmente cuando se encuentran presentes bajas cantidades de ácido nucleico diana. (Li et al., Nucleic Acids Research, Vol. 30, No. 2, e5 (2002) ) .
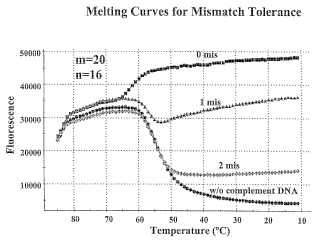
La detección de ARN virales presenta ciertos desafíos, que no son planteados para detectar los ADN de interés. Las sondas de la técnica anterior son adecuadas para la detección de ARN virales, pero podrían ser mejoradas. En primer lugar, algunas dianas de ARN viral son propensas a rápidas mutaciones en los organismos de sus anfitriones. Para asegurarse de que las secuencias de ARN viral mutado son detectadas junto con las denominadas secuencias "de tipo salvaje", las sondas de ácido nucleico utilizadas para detectar los ARN virales deben ser tolerantes a los emparejamientos erróneos, pero a pesar de ello suficientemente específicas para evitar la interacción con ácidos... [Seguir leyendo]
Reivindicaciones:
1. Un método para cuantificar ARN en una muestra de ensayo, comprendiendo el método (A) mezclar la muestra de ensayo con los reactivos de amplificación, los reactivos de transcripción inversa y una sonda de ácido nucleico que comprende un primer ácido oligonucleico y el segundo ácido oligonucleico, donde (I)
a) el primer ácido oligonucleico tien.
30. 34 bases (m) de longitud y tiene una homología de 80% o más con un ácido nucleico de interés, y comprende adicionalmente un fluoróforo; b) el segundo ácido oligonucleico tiene n bases de longitud donde n está relacionado con m de manera que cuando m es de 30 a 34, n es de 8 a 15; y comprende adicionalmente un extintor; c) el primer ácido oligonucleico y, el segundo ácido oligonucleico se caracterizan porque:
i) el segundo ácido oligonucleico tiene una homología de 80% o más con el primer ácido oligonucleico, y ii) el primer ácido oligonucleico y el segundo ácido oligonucleico se pueden unir entre sí en ausencia del ácido nucleico de interés en condiciones apropiadas para formar un ácido nucleico de doble hebra de manera que la emisión de fluorescencia del fluoróforo anclado al primer ácido oligonucleico cuando el primer ácido oligonucleico está unido al segundo ácido oligonucleico es detectable menos que la emisión del mismo fluoróforo cuando el primer ácido oligonucleico y el segundo ácido oligonucleico no están unidos entre sí, o (II)
a) el primer ácido oligonucleico tien.
30. 34 bases (m) de longitud y tiene una homología de 80% o más con un ácido nucleico de interés, y comprende un extintor, b) el segundo ácido oligonucleico tiene n bases de longitud donde n está relacionado con m de manera que cuando m es de 30 a 34, n es de 8 a 15; y comprende un fluoróforo; c) el primer ácido oligonucleico y el segundo ácido oligonucleico se caracterizan porque:
i) el segundo ácido oligonucleico tiene una homología de 80% o más con primer ácido oligonucleico, y ii) el primer ácido oligonucleico y el segundo ácido oligonucleico se pueden unir entre sí en ausencia del ácido nucleico de interés en condiciones apropiadas para formar un ácido nucleico de doble hebra de manera que la emisión fluorescente del fluoróforo anclado al segundo ácido oligonucleico cuando el primer ácido oligonucleico está unido con respecto al segundo ácido oligonucleico es detectable menos que la emisión del mismo fluoróforo cuando el primer ácido oligonucleico y el segundo ácido oligonucleico no están unidos entre sí, para crear una mezcla en un recipiente de reacción,
(B) colocar la muestra de ensayo en condiciones permisivas de transcripción inversa, que opcionalmente también pueden ser permisivas de amplificación, de manera que se produce un ADNc,
(C) someter a ciclo térmico la mezcla de ensayo de manera que se amplifique el ADNc, y
(D) medir la fluorescencia de la mezcla de ensayo durante la reacción de amplificación como una indicación de la cantidad de ARN de la muestra de ensayo.
2. El método de la reivindicación 1, donde en dicha sonda de ácido nucleico, el primer y el segundo ácidos oligonucleicos comprenden nucleobases adicionales que no son complementarias al ácido nucleico de interés y el primer oligonucleótido, respectivamente, en los extremos 5' y 3'.
3. El método de la reivindicación 1, opción (I) , donde en dicha sonda de ácido nucleico la razón del segundo ácido oligonucleico con respecto al primer ácido oligonucleico es mayor de 1, 1 o donde en dicha sonda de ácido nucleico de la opción (II) , la razón del primer ácido oligonucleico con respecto al segundo ácido oligonucleico es mayor de 1, 1.
4. El método de la reivindicación 1, opción (I) , donde en dicha sonda de ácido nucleico la razón del segundo ácido oligonucleico con respecto al primer ácido oligonucleico es mayor de 0, 1 y menor de 0, 9 o donde en dicha sonda de ácido nucleico de la opción (II) , la razón del primer ácido oligonucleico con respecto al segundo ácido oligonucleico es mayor de 0, 1 y menor de 0, 9.
5. El método de la reivindicación 1, opción (I) , donde en dicha sonda de ácido nucleico el primer ácido oligonucleico comprende adicionalmente un extintor o donde en dicha sonda de ácido nucleico de la opción (II) , el segundo ácido oligonucleico comprende adicionalmente un extintor.
6. El método de la reivindicación 5, donde, en la opción (I) , en dicha sonda de ácido nucleico la razón del segundo ácido oligonucleico con respecto al primer ácido oligonucleico es mayor de 0, 1 y menor de 0, 9 o donde, en la opción
(II) , en dicha sonda de ácido nucleico la razón del primer ácido oligonucleico con respecto al segundo ácido oligonucleico es mayor de 0, 1 y menor de 0, 9.
7. El método de la reivindicación 5, donde, en la opción (I) , en dicha sonda de ácido nucleico la razón del segundo ácido oligonucleico con respecto al primer ácido oligonucleico es mayor de 1, 1 o donde, en la opción (II) , en dicha sonda de ácido nucleico la razón del primer ácido oligonucleico con respecto al segundo ácido oligonucleico es mayor de 1, 1.
8. El método de la reivindicación 1, opción I, donde en dicha sonda de ácido nucleico el primer ácido oligonucleico comprende al menos dos fluoróforos para permitir que la fluorescencia generada por el primer ácido oligonucleico que comprende dos fluoróforos cuando está unido a un ácido nucleico de interés sea sustancialmente mayor que la fluorescencia generada cuando solamente está presente un fluoróforo.
9. El método de cualquiera de las reivindicaciones 1-8, donde dicho primer ácido oligonucleico se selecciona del grupo que consiste en el SEQ ID NO: 5.
10. El método de las reivindicaciones 1 – 8, donde dicho segundo ácido oligonucleico se selecciona del grupo que consiste en el SEQ ID NO: 11.
11. El uso de una sonda de ácido nucleico que comprende un primer ácido oligonucleico y un segundo ácido oligonucleico, donde
(I)
a) el primer ácido oligonucleico tien.
30. 34 bases (m) de longitud y tiene una homología de 80% o más con un ácido nucleico de interés, y comprende adicionalmente un fluoróforo; b) el segundo ácido oligonucleico tiene n bases de longitud donde n está relacionado con m de manera que cuando m es de 30 a 34, n es de 8 a 15; y comprende adicionalmente un extintor; c) el primer ácido oligonucleico y, el segundo ácido oligonucleico se caracterizan porque:
i) el segundo ácido oligonucleico tiene una homología de 80% o más con el primer ácido oligonucleico, y ii) el primer ácido oligonucleico y el segundo ácido oligonucleico se pueden unir entre sí en ausencia del ácido nucleico de interés en condiciones apropiadas para formar un ácido nucleico de doble hebra de manera que la emisión fluorescente del fluoróforo anclado al primer ácido oligonucleico cuando el primer ácido oligonucleico está unido con respecto al segundo ácido oligonucleico es detectable menos que la emisión del mismo fluoróforo cuando el primer ácido oligonucleico y el segundo ácido oligonucleico no están unidos entre sí, o (II)
a) el primer ácido oligonucleico tien.
30. 34 bases (m) de longitud y tiene una homología de 80% o más con un ácido nucleico de interés, y comprende un extintor, b) el segundo ácido oligonucleico tiene n bases de longitud donde n está relacionado con m de manera que cuando m es de 30 a 34, n es de 8 a 15; y comprende un fluoróforo; c) el primer ácido oligonucleico y el segundo ácido oligonucleico se caracterizan porque:
i) el segundo ácido oligonucleico tiene una homología de 80% o más con el primer ácido oligonucleico, y ii) el primer ácido oligonucleico y el segundo ácido oligonucleico se pueden unir entre sí en ausencia del ácido nucleico de interés en condiciones apropiadas para formar un ácido nucleico de doble hebra de manera que la emisión de fluorescencia del fluoróforo anclado al segundo ácido oligonucleico cuando el primer ácido oligonucleico está unido con respecto al segundo ácido oligonucleico es detectable menos que la emisión del mismo fluoróforo cuando el primer ácido oligonucleico y el segundo ácido oligonucleico no están unidos entre sí, en un método para la reivindicación 1.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]