Sistema de expresión para proteínas.
Una célula huésped capaz de expresar ARN polimerasa de T7 tras inducción,
comprendiendo la célula huésped:
un primer ácido nucleico que tiene un gen de la lisozima de T7 o un gen variante de la lisozima de T7 que codifica una variante de la lisozima de T7 que tiene la capacidad de inhibir la actividad de la ARN polimerasa de T7, bajo el control de un promotor, y un segundo ácido nucleico que tiene un promotor de T7 unido operablemente a una secuencia de ácido nucleico que codifica un polipéptido diana, que se caracteriza porque el gen de la lisozima de T7 o el gen variante de la lisozima de T7 está bajo el control de un promotor ajustable;
el promotor ajustable tiene un efecto sobre el índice de transcripción del gen de la lisozima de T7 que depende de la concentración o intensidad de un inductor del promotor, de forma que el índice de transcripción del gen de la lisozima de T7 aumenta con la concentración o intensidad creciente del inductor y que la expresión de la lisozima de T7 se puede ajustar de forma continua mediante dicho promotor ajustable;
y porque el promotor ajustable se selecciona de un modo tal que la expresión del polipéptido diana se puede ajustar mediante el control de la expresión del gen de la lisozima de T7 o el gen variante de la lisozima de T7 cuando se induce la expresión de la ARN polimerasa de T7.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2009/052414.
Solicitante: Xbrane Bioscience Aktiebolag.
Nacionalidad solicitante: Suecia.
Dirección: Stureplan 15 111 45 Stockholm SUECIA.
Inventor/es: WILLEM DE GIER,Jan, WAGNER,Samuel.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/63 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Introducción de material genético extraño utilizando vectores; Vectores; Utilización de huéspedes para ello; Regulación de la expresión.
- C12N15/67 C12N 15/00 […] › Métodos generales para favorecer la expresión.
- C12N9/36 C12N […] › C12N 9/00 Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas. › actúan sobre los enlaces beta-1,4 del ácido N-acetilmurámico con aceitilamino-2 deoxi-2-D-glucosa, p. ej. lisozima.
PDF original: ES-2381433_T3.pdf
Fragmento de la descripción:
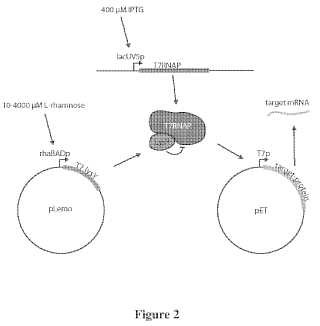
Sistema de expresión para proteínas Campo de la invención La invención se refiere a un sistema de expresión para polipéptidos. En concreto, la invención se refiere a plásmidos y células huésped para expresión de polipéptidos, que pueden ajustar la intensidad de la expresión mejorando de este modo los rendimientos de la expresión. Más específicamente, la presente invención se refiere a vectores nuevos que se expresan en un huésped, que comprende un promotor ajustable unido operablemente a una secuencia de ácido nucleico que codifica la lisozima T7. Además, la invención se refiere al uso de estos vectores para la expresión de ácidos nucleicos que codifican la lisozima de T7. La invención también se refiere al uso de la lisozima de Y7 para ajustar la actividad de la ARN polimerasa de T7 de un modo para dar una expresión óptima de ácidos nucleicos que codifican, por ejemplo, polipéptidos, en concreto proteínas con una compleja biogénesis postraduccional, por ejemplo proteínas de membrana, proteínas secretoras, proteínas modificadas postraduccionalmente (p. ej., glicosiladas) y proteínas que requieren la ayuda de chaperonas para su plegamiento.
Antecedentes de la invención Se han descrito muchos sistemas para la expresión de ácidos nucleicos que codifican, por ejemplo, polipéptidos tales como proteínas recombinantes en sistemas procariotas. Los sistemas de expresión basados en la ARN polimerasa de T7 (T7RNAP, SEC ID Nº 17) son los sistemas más usados para producir proteínas recombinantes en Escherichia coli (Studier, y col. (1990) Methods Enzymol 185:60-89) . Cabe destacar que además de en E. coli, la expresión basada en T7RNAP se ha usado en un huésped de diferentes organismos procariotas y eucariotas (p. ej., Kang y col. (2007) Protein Expr Purif. 55 (2) :325-33; Dower y Robasch (2002) RNA 8:686-697; Nguyen y col. (2004) Plant Biotechnol. J. 2: 301-310; Polkinghorne y Roy (1995) Nucleic Acids Res. 23 (1) : 188-191; Wang et al. (2007) Analytical Biochemistr y , en prensa (doi:10.1016/j.ab.2007.11.037) ) . La T7RNAP reconoce un promotor muy específico, es decir un promotor de T7 (Chamberlin y col., (1970) Nature 228, 227-231; Oakley y Coleman (1977) PNAS 74, 4266-4270; Rosa (1979) Cell 16, 815-825 ;Panayotatos y Wells (1979) Nucl. Acids Res. 13, 2227-2240; Dunn y Studier (1983) J. Mol. Biol. 166, 477-535/175, 111-112) , y transcribe ADN con una tasa de transcripción ocho veces mayor que la ARN polimerasa de E. coli (lost, y col. (1992) J Bacteriol 174:619-22) . Esto es una gran ventaja para la producción de alto rendimiento de la proteína recombinante en E. coli. El inconveniente de la buena eficiencia es, por ejemplo, la gran carga metabólica y la tensión de plegamiento impuesta sobre la célula huésped. Aunque la producción de proteínas en el citoplasma de E. coli es relativamente sencilla, la sobreexpresión de proteínas de membrana en E. coli sigue suponiendo un reto (Wagner, y col. (2006) Trends Biotechnol 24:364-71) . Aunque las proteínas de membrana a menudo pueden expresarse fácilmente en los cuerpos de inclusión, su replegamiento en proteínas funcionales no suele tener éxito. La sobreexpresión de las proteínas de membrana por la acumulación en el sistema de la membrana citoplásmica evita este problema de replegamiento, pero suele ser tóxico, de modo que se reducen mucho los rendimientos. Esto se debe principalmente a los complejos requisitos de la biogénesis de las proteínas de membrana, en las que, tras el inicio de la traducción en el ribosoma, los complejos de las cadenas nacientes de proteína de membrana y ribosoma están dirigidos a la membrana plasmática (Luirink, y col. (2005) Annu Rev Microbiol 59:329-55) . Los dominios transmembranales (DTM) de las proteínas de membrana quedan atrapados en el translocón Sec y, después, se dividen en la bicapa lipídica. La sobreexpresión de la proteína de membrana satura fácilmente una o varias etapas de la vía de la biogénesis que conduce a la agregación indeseada y la degradación de proteínas recombinantes en el citoplasma. Además, el bloqueo de la vía secretora tiene como resultado una toxicidad intensa para la célula huésped (Wagner, y col. (2007) Mol Cell Proteomics 6:1527-50) . Una menor viabilidad de la célula huésped y el plegamiento incorrecto de la proteína recombinante sobreexpresada tiene como resultado bajos rendimientos del producto deseado. En este caso, el principal problema es que la expresión de los polipéptidos por la T7RNAP es demasiado fuerte. La mayoría de los sistemas de expresión basados en T7RNAP han dependido exclusivamente de una actividad fija de la T7RNAP que tiene como resultado una intensidad fija de expresión de proteínas recombinantes. Las cepas más usadas para este fin son BL21 (DE3) y sus derivados BL21 (DE3) pLysS, BL21 (DE3) pLysE, C41 (DE3) y C43 (DE3) (Studier (1991) J Mol Biol 219:37-44) (Miroux (1996) J Mol Biol 260: 289-98) . Los huéspedes portadores de los plásmidos pLysS y pLysE expresan la lisozima de T7, , pLysS a un nivel bajo establecido y , pLysE a un nivel alto establecido. Todos estos sistemas fijos no dejan oportunidad a ajustar la intensidad de la expresión a los requisitos individuales de diferentes proteínas diana. Dado que es impredecible qué intensidad de expresión es óptima para obtener los mejores rendimientos de sobreexpresión de una proteína concreta, la expresión se tiene que cribar en diferentes cepas con intensidades de expresión fijas basadas en T7RNAP. Sería ideal que se pudieran someter a detección selectiva diferentes intensidades de expresión en solo un sistema de expresión basado en T7RNAP. Este sistema de expresión "todo en uno" facilitaría enormemente la detección selectiva de la sobreexpresión. Por tanto, existe la necesidad de proporcionar sistemas de expresión procariotas y eucariotas alternativos con la capacidad para ajustar de forma continua la intensidad de la expresión de secuencias de ácido nucleico, en particular de las que codifican proteínas de membrana y otros polipéptidos con complejos requisitos biogenéticas postraduccionales; por ejemplo proteínas secretoras, proteínas que se modifican postraduccionalmente (p. ej., glicosiladas (Wacker y col., (2002) Science 298. páginas 1790 - 1793) , y proteínas que requieren la ayuda de chaperonas para su plegamiento. Encontrar la cepa óptima basada en T7RNAP para la sobreexpresión de una proteína es cuestión de "ensayo y error" que requiere tiempo, de modo que se usan, por ejemplo, BL21 (DE3) , BL21 (DE3) pLysS, BL21 (DE3) pLysS
(Studier (1991) J Mol Biol 219: 37 44) , C41 (DE3) y C43 (DE3) (Miroux (1996) J Mol Biol 260, 289-98) . En estas cepas, la actividad de T7RNAP está fijada y se debe realizar detección selectiva de la cepa más adecuada según la sobreexpresión de la proteína deseada. La cepa que será la mejor es algo que no se puede predecir.
Técnica anterior Studier ( (1991) Journal of Molecular Biology 219:37-44) divulga varios sistemas basados en la ARN polimerasa de T7 para la expresión de proteínas. En los sistemas de expresión divulgados se usan niveles en equilibrio de la lisozima T7 con el fin de inhibir la actividad de fondo de la ARN polimerasa de T7 y, por tanto, prevenir la expresión con fugas y/o reducir los niveles de expresión de las proteínas diana tóxicas. Reduciendo la expresión con fugas de la proteína diana antes de la inducción de la expresión y/o reduciendo los niveles de la expresión inducida de las proteínas diana se mejora la viabilidad de la célula huésped y, por tanto, se incrementan los rendimientos proteicos.
Se han desarrollado varios sistemas de expresión en los que la ARN polimerasa de T7 está bajo el control directo de un promotor inducible:
Hartnett y col. ( (2006) Promega notes 94:27-30) y el documento EP 1 847 611 A2 divulgan sistemas para expresión de proteínas, en los que la ARN polimerasa de T7 está bajo el control directo de un promotor rhaBAD inducible por ramnosa.
McKinney y col. ( (2002) Journal of Bacteriology 184: 6056-6059) divulgan una cepa de Salmonella para expresión génica regulada, en la que la ARN polimerasa de T7 está bajo el control de un elemento de control araC-PBAD que es inducible por arabinosa.
Sistemas similares se divulgan en el documento WO 2008/017073 A, Wycuff y col. ( (2000) Analytical Biochemistr y 277:67-73 y Chao y col. ( (2002) Biotechnology progress 18:8756-7938.
Otro sistema basado en la ARN polimerasa de T7 inducible o ajustable, en el que la ARN polimerasa de T7 está directamente regulada por, por ejemplo, calor o luz, incluye sistemas divulgados en Chao y col. (2002) Applied Microbiology and Biotechnology 58:446-453) , Wang y col. (2004) Biotechnology... [Seguir leyendo]
Reivindicaciones:
1. Una célula huésped capaz de expresar ARN polimerasa de T7 tras inducción, comprendiendo la célula huésped:
un primer ácido nucleico que tiene un gen de la lisozima de T7 o un gen variante de la lisozima de T7 que codifica una variante de la lisozima de T7 que tiene la capacidad de inhibir la actividad de la ARN polimerasa de T7, bajo el control de un promotor, y un segundo ácido nucleico que tiene un promotor de T7 unido operablemente a una secuencia de ácido nucleico que codifica un polipéptido diana, que se caracteriza porque el gen de la lisozima de T7 o el gen variante de la lisozima de T7 está bajo el control de un promotor ajustable;
el promotor ajustable tiene un efecto sobre el índice de transcripción del gen de la lisozima de T7 que depende de la concentración o intensidad de un inductor del promotor, de forma que el índice de transcripción del gen de la lisozima de T7 aumenta con la concentración o intensidad creciente del inductor y que la expresión de la lisozima de T7 se puede ajustar de forma continua mediante dicho promotor ajustable;
y porque el promotor ajustable se selecciona de un modo tal que la expresión del polipéptido diana se puede ajustar mediante el control de la expresión del gen de la lisozima de T7 o el gen variante de la lisozima de T7 cuando se induce la expresión de la ARN polimerasa de T7.
2. La célula huésped de la reivindicación 1, en la que dicho ácido nucleico comprende el promotor ajustable es un primer vector y dicho segundo ácido nucleico que comprende el promotor de T7 es un segundo vector y en el que el primero y el segundo vector son compatibles.
3. La célula huésped de cualquiera de las reivindicaciones 1 o 2, en la que dicho promotor ajustable es ajustable por ramnosa o arabinosa.
4. La célula huésped de cualquiera de las reivindicaciones 1 o 2, en la que dicho promotor ajustable es ajustable por luz.
5. La célula huésped de cualquiera de las reivindicaciones 1 o 2, en la que dicho promotor ajustable es ajustable por temperatura.
6. La célula huésped de cualquiera de las reivindicaciones 1 a 5, en la que dicho primer ácido nucleico comprende un marcador de selección.
7. La célula huésped de la reivindicación 6, en la que el marcador de selección es un marcador de selección antibiótico.
8. La célula huésped de cualquiera de las reivindicaciones 1 a 7, en la que dicha variante de la lisozima de T7 es LysY.
9. La célula huésped de cualquiera de las reivindicaciones 1 a 8, en la que dicho primer ácido nucleico es al menos un 80 % idéntico a un ácido nucleico escogido del grupo que comprende la SEC ID Nº 1-5.
10. La célula huésped de la reivindicación 9, en la que dicho primer ácido nucleico es idéntico a un ácido nucleico escogido del grupo que comprende la SEC ID Nº 1-5.
11. Una célula huésped de las reivindicaciones 1 a 10, seleccionada del grupo de E. coli, Pseudomonas aeruginosa, Erwinia carotovora, Salmonella choleraesuis, Agrobacterium tumefaciens, Chromobacterium violaceum, Lactococcus lactis, Bacillus subtilis, Salmonella, Saccharomyces cerevisiae, Pichia pastoris, Kluyveromyces lactis, CHO, NS0, HEK293, HeLa, Sf9, tabaco, arroz y Leishmania tarentolae.
12. Un procedimiento para producir un polipéptido diana, que comprende las etapas de: a) proporcionar una célula huésped de acuerdo con una cualquiera de las reivindicaciones 1 a 11; b) inducir la expresión del polipéptido diana c) controlar la expresión del polipéptido diana ajustando dicho promotor ajustable y, por tanto, la expresión de dicha
lisozima de T7; y opcionalmente d) aislar el polipéptido.
13. El procedimiento de la reivindicación 12, en el que la inducción de la expresión del polipéptido diana se realiza induciendo la expresión de la ARN polimerasa de T7 o una variante de la misma.
14. El procedimiento de cualquiera de las reivindicaciones 12 a 13, en el que el ajuste del promotor ajustable, o una
variante del mismo, se consigue mediante cualquiera de: inducción ligera, ajuste de la temperatura, adición de un 5 inductor químico, en concreto ramnosa o arabinosa.
15. Un ácido nucleico que comprende un gen de la lisozima de T7 o un gen variante de la lisozima de T7 que codifica una variante de la lisozima de T7 que tiene la capacidad de inhibir la actividad de la ARN polimerasa de T7, bajo el control de un promotor, que se caracteriza porque el gen de la lisozima de T7 o el gen variante de la lisozima de T7 está bajo el control de un promotor rhaBAD ajustable y que el promotor ajustable tiene un efecto sobre el índice de transcripción del gen de la lisozima de T7 que depende de la concentración de un inductor del promotor rhaBAD, de forma que el índice de transcripción del gen de la lisozima de T7 aumenta con la concentración creciente del inductor y que la expresión de la lisozima de T7 se puede ajustar de forma continua mediante dicho promotor rhaBAD ajustable.
16. Un ácido nucleico de acuerdo con la reivindicación 15, que es al menos un 80 % idéntico con un ácido nucleico escogido del grupo de las SEC ID Nº 1-5.
17. Un ácido nucleico de acuerdo con la reivindicación 16 escogido del grupo de las SEC ID Nº 1-5.
18. Uso del ácido nucleico de cualquiera de las reivindicaciones 15 a 17 para controlar la expresión de un polipéptido diana mediante la lisozima de T7.
Patentes similares o relacionadas:
 Métodos para producir proteínas bicatenarias en bacterias, del 29 de Julio de 2020, de GENENTECH, INC.: Un método para producir un receptor de linfocitos T monoclonal de movilización inmunitaria contra el cáncer (ImmTAC) que comprende una cadena alfa del receptor de linfocitos […]
Métodos para producir proteínas bicatenarias en bacterias, del 29 de Julio de 2020, de GENENTECH, INC.: Un método para producir un receptor de linfocitos T monoclonal de movilización inmunitaria contra el cáncer (ImmTAC) que comprende una cadena alfa del receptor de linfocitos […]
Anticuerpo biespecífico o mezcla de anticuerpos con cadenas ligeras comunes, del 15 de Julio de 2020, de Jiangsu Alphamab Biopharmaceuticals Co., Ltd: Anticuerpo biespecífico o parte de unión a antígeno del mismo, en el que el anticuerpo biespecífico o la parte de unión a antígeno del mismo tiene una cadena […]
Vacunas y diagnóstico de torque teno virus porcino, del 18 de Junio de 2020, de VIRGINIA TECH INTELLECTUAL PROPERTIES, INC.: Composición inmunogénica que comprende una proteína según SEQ ID NO. 16.
Producción de FDCA catalizada por deshidrogenasa, del 17 de Junio de 2020, de PURAC BIOCHEM B.V.: Proceso para oxidar ácido 5-hidroximetil-2-furancarboxílico (HMFCA) a ácido 5-formil-2-furoico (FFA), donde el proceso comprende la etapa de incubar una […]
Vectores de AAV dirigidos a oligodendrocitos, del 10 de Junio de 2020, de THE UNIVERSITY OF NORTH CAROLINA AT CHAPEL HILL: Un ácido nucleico que codifica una cápside de AAV, comprendiendo el ácido nucleico una secuencia codificante de la cápside de AAV que es al menos el 96 % idéntica […]
Ácido nucleico que comprende o codifica para un tallo-bucle de histona y una secuencia poli(A) o una señal de poliadenilación para aumentar la expresión de un antígeno patogénico codificado, del 3 de Junio de 2020, de CureVac AG: Secuencia de ácido nucleico que comprende o codifica en la dirección 5' → 3' para: i) • una región codificante, que codifica para al menos […]
Microorganismo con productividad de l-lisina aumentada y procedimiento para producir l-lisina utilizando el mismo, del 27 de Mayo de 2020, de CJ CHEILJEDANG CORPORATION: Una subunidad beta prima (subunidad-β') mutante de la ARN polimerasa, en la que la subunidad beta prima (subunidad-β') mutante de la ARN polimerasa tiene […]
Agrupación de genes de biosíntesis de carrimicina, del 27 de Mayo de 2020, de Shenyang Fuyang Pharmaceutical Technology Co., Ltd: Agrupación de genes de biosíntesis de carrimicina, que consiste en 44 genes que comprende: 1) cinco genes de policétido sintasa, incluyendo los residuos […]