Proteínas de unión aprisionadas como biosensores.
Un biosensor de la glucosa que comprende al menos una proteína de unión a glucosa/galactosa mutada (GGBP) y al menos un grupo indicador luminiscente (marcador luminiscente) unido mediante enlace covalente a la mismo,
en el que dicho al menos un grupo indicador luminiscente es capaz de proporcionar una señal detectable y reversible como respuesta a la unión con dicha GGBP mutante, y en el que dicha GGBP mutada tiene al menos una sustitución de aminoácido de un aminoácido no reactivo con un aminoácido reactivo que se puede modificar con un agente de marcaje análogo al etiquetado de cisteína con un colorante reactivo al tiol, y dicha GGBP mutada se une a la glucosa y se encapsula en una matriz permeable al analito que comprende sol-geles aumentados con silicatos modificados orgánicamente o con hidrogeles reticulados covalentemente, o con combinaciones de los mismos.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2003/000201.
Solicitante: BECTON, DICKINSON AND COMPANY.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: INTELLECTUAL PROPERTY DEPARTMENT, 1 BECTON DRIVE, MAIL CODE 089 FRANKLIN LAKES, NJ 07417-1880 ESTADOS UNIDOS DE AMERICA.
Inventor/es: PITNER, J. BRUCE, KNORS, CHRISTOPHER, J., ALARCON,Javier.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07K14/195 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 14/00 Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados. › de origen bacteriano.
- C07K17/00 C07K […] › Péptidos fijados sobre un soporte o inmovilizados; Su preparación.
- C07K17/04 C07K […] › C07K 17/00 Péptidos fijados sobre un soporte o inmovilizados; Su preparación. › encerrados en el interior del soporte, p. ej. en un gel, en una fibra hueca.
- C12P39/00 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › Procesos que hacen intervenir simultáneamente microorganismos de diferentes clases en el mismo proceso.
- C12Q1/00 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones.
- C12Q1/54 C12Q […] › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que interviene la glucosa o la galactosa.
- G01N21/78 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 21/00 Investigación o análisis de los materiales por la utilización de medios ópticos, es decir, utilizando rayos infrarrojos, visibles o ultravioletas (G01N 3/00 - G01N 19/00 tienen prioridad). › produciendo un cambio de color.
- G01N27/00 G01N […] › Investigación o análisis de materiales mediante el empleo de medios eléctricos, electroquímicos o magnéticos (G01N 3/00 - G01N 25/00 tienen prioridad; medida o ensayo de variables eléctricas o magnéticas o de las propiedades eléctricas o magnéticas de los materiales G01R).
- G01N31/00 G01N […] › Investigación o análisis de materiales no biológicos mediante el empleo de los métodos químicos especificados en los subgrupos; Aparatos especialmente adaptados a tales métodos.
- G01N31/22 G01N […] › G01N 31/00 Investigación o análisis de materiales no biológicos mediante el empleo de los métodos químicos especificados en los subgrupos; Aparatos especialmente adaptados a tales métodos. › Utilización de reactivos químicos (G01N 31/02 tiene prioridad).
- G01N33/52 G01N […] › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › Utilización de compuestos o de composiciones para investigaciones colorimétricas, espectrofotométricas o fluorométricas, p. ej. utilización de cintas de papel indicador.
- G01N33/66 G01N 33/00 […] › en los que intervienen azúcares de la sangre, p. ej. la galactosa.
PDF original: ES-2380922_T3.pdf
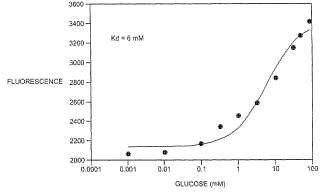
Fragmento de la descripción:
Proteínas de unión aprisionadas como biosensores Antecedentes de la invención
1. Campo de la invención
La invención pertenece al campo de la Biotecnología. Específicamente, la invención se dirige a proteínas de unión mutadas aprisionadas, proteínas de unión mutadas que contienen grupos indicadores, composiciones de proteínas de unión mutadas que contienen grupos indicadores en matrices permeables a analitos y a su uso como biosensores de analitos tanto in vitro como in vivo.
2. Descripción de la técnica relevante
La monitorización de las concentraciones de glucosa para facilitar el control metabólico adecuado en diabéticos es un objetivo deseable y mejoraría la vida de muchos individuos. Actualmente, la mayoría de los diabéticos usan el procedimiento del "pinchazo en la yema del dedo" para controlar sus niveles de glucosa en sangre, siendo su aceptación problemática por parte del paciente debido al dolor provocado por los frecuentes pinchazos (varias veces al día) . Por consiguiente, se han hecho esfuerzos por desarrollar procedimientos no invasivos o mínimamente invasivos in vivo e in vitro más eficientes para el control frecuente y/o continuo de la glucosa en sangre o de otros fluidos biológicos que contienen glucosa. Algunos de estos procedimientos más prometedores implican el uso de un biosensor. Los biosensores son dispositivos capaces de proporcionar información analítica cuantitativa o semi-cuantitativa específica usando un elemento de reconocimiento biológico combinado con un elemento de transducción (detección) .
El elemento de reconocimiento biológico de un biosensor determina la selectividad, de manera que sólo el compuesto que se desee medir produzca una señal. La selección se puede basar en el reconocimiento bioquímico del ligando, en el que la estructura química del ligando (p. ej., glucosa) permanece invariable, o en la biocatálisis en la que el elemento de reconocimiento biológico cataliza una reacción bioquímica del analito.
El transductor traduce el reconocimiento del elemento de reconocimiento biológico en una señal semicuantitativa o cuantitativa. Las posibles tecnologías de transductores son óptica, electroquímica, acústica/mecánica o colorimétrica. Las propiedades ópticas que se han aprovechado incluyen la absorbancia, la fluorescencia/fosforescencia, la bio/quimioluminiscencia, la reflectancia, la difusión de la luz y el índice de refracción. Se pueden usar grupos indicadores convencionales, tales como compuestos fluorescentes o, alternativamente, existe la oportunidad de realizar la detección óptica directa sin la necesidad de un marcador.
Los biosensores específicamente diseñados para la detección de la glucosa que usan elementos biológicos para la transducción de señales usan comúnmente la detección electroquímica o colorimétrica de la actividad de la glucosa oxidasa. El uso de este procedimiento está asociado con dificultades que resultan de la influencia de los niveles de oxígeno, la presencia de inhibidores en la sangre y problemas con electrodos, entre otros. Además, la detección produce el consumo del analito que puede provocar dificultades al medir bajas concentraciones de glucosa.
Una parte del desarrollo de los biosensores que avanza rápidamente es el uso de proteínas de unión periplásmicas marcadas fluorescentemente. Según lo publicado por Cass (Anal Chem. 1994, 66 3840-3847) , se demostró la utilidad de una proteína de unión a la maltosa marcada (MBP) como sensor de la maltosa. En este trabajo, se mutó la MBP, que no tiene residuos de cisteína nativos, para proporcionar una proteína con un solo residuo de cisteína en la posición 337 (S337C) . La posición de esta mutación se encontraba en la hendidura de la unión a maltosa y experimentó un gran cambio ambiental tras la unión a maltosa. Se estudiaron numerosos fluoróforos, algunos bien bloqueaban la unión a ligandos o interferían en el cambio de configuración de la proteína. Entre los estudiados, el N- ( (2-yodoacetoxi) etil) -N-metil) amino-7-nitrobenzoxadiazol (IANBD) produjo un aumento sustancial de la intensidad de la fluorescencia (160%) tras la unión a maltosa. Este resultado coincide con el cambio de ubicación del fluoróforo de un medio hidrófilo o expuesto a un disolvente a un medio más hidrófobo, tal y como se habría predicho teóricamente mediante el cierre de la bisagra tras la unión a maltosa. En cualquiera caso, esta proteína mutante y el grupo indicador asociado no se unen diagnósticamente con azúcares importantes en fluidos corporales de mamíferos. Cass también reveló la asociación de esta proteína con superficies de TiO2. Sin embargo, la proteína unida a la superficie sufrió la reducción de la actividad con el tiempo y requirió una hidratación constante (Analytical Chemistr y 1998, 70 (23) , 5111-5113) .
Hellinga, et al., (US 6.277.627) publicaron la ingeniería de un biosensor de glucosa mediante la introducción de un transductor fluorescente en una proteína de unión a galactosa/glucosa (GGBP) mutada para contener un residuo de cisteína, aprovechando los grandes cambios de configuración que tienen lugar tras la unión con glucosa. Hellinga et al (US 6.277.627) revelan que se puede aprovechar la transmisión de los cambios de configuración en GGBP mutadas para crear funciones de transducción de señales integradas que conviertan una unión a la glucosa en un cambio en la fluorescencia mediante un mecanismo de acoplamiento alostérico. Se ha publicado que las funciones de transducción fluorescente interfieren mínimamente en las propiedades de unión intrínsecas del bolsillo de unión a azúcar de la GGBP.
Para determinar con exactitud la concentración de glucosa de soluciones biológicas, tales como sangre, fluidos intersticiales, soluciones oculares, transpiraciones, etc., puede ser deseable ajustar la constante de unión de la molécula de detección de un biosensor para que coincida con el intervalo operativo fisiológico y/o patológico de la solución biológica de interés. Sin la constante de unión apropiada, la señal puede quedar fuera del intervalo para una determinada concentración fisiológica y/o patológica. Además, se pueden configurar biosensores usando más de una proteína, cada una con una constante de unión diferente para proporcionar mediciones exactas en un intervalo amplio de concentraciones de glucosa según lo revelado por Lakowicz (US 6.197.534) .
A pesar de la utilidad de las GGBP mutadas, han sido pocas de estas proteínas las que se han diseñado y examinado, bien con o sin grupos indicadores. Las mutaciones específicas de los sitios y/o la unión de ciertos grupos indicadores pueden actuar para modificar una constante de unión de un modo impredecible. Además, un biosensor que contenga grupos indicadores puede tener una constante de unión deseable, pero no producir una señal fácilmente detectable tras la unión al analito. Uno de los factores primordiales que determina la sensibilidad de una determinada sonda indicadora unida con una determinada proteína para la detección de un analito específico es la naturaleza de las interacciones específicas entre la sonda seleccionada y los residuos de aminoácido de la proteína. Actualmente, no es posible predecir estas interacciones con proteínas usando procedimientos de cálculo, ni es posible emplear una metodología de un diseño racional para optimizar la elección de las sondas indicadoras. Además, no es posible predecir el efecto ni en la constante de unión ni en la selectividad basadas en la posición de cualquier grupo indicador o sustitución de aminoácido en la proteína (o viceversa) .
Para desarrollar biosensores sin reactivos, auto-contenidos y/o implantables y/o reutilizables con el uso de proteínas, el elemento de transducción debe estar en comunicación con un dispositivo de detección para interrogar a la señal que va hacia y procede del elemento de transducción. Los procedimientos más comunes incluyen la colocación de proteínas en o sobre la superficie de fibras ópticas o guías de onda planificadoras empleando estrategias de inmovilización. Tales estrategias de inmovilización incluyen, sin limitaciones, el aprisionamiento de la proteína en membranas semipermeables, matrices de polímeros orgánicos o matrices de polímeros inorgánicos. La estrategia de inmovilización usada puede determinar en última instancia el rendimiento de un biosensor en funcionamiento. La técnica anterior detalla numerosos problemas asociados con la inmovilización de moléculas biológicas. Por ejemplo, muchas proteínas sufren cambios irreversibles de configuración, desnaturalización... [Seguir leyendo]
Reivindicaciones:
1. Un biosensor de la glucosa que comprende al menos una proteína de unión a glucosa/galactosa mutada (GGBP) y al menos un grupo indicador luminiscente (marcador luminiscente) unido mediante enlace covalente a la mismo, en el que dicho al menos un grupo indicador luminiscente es capaz de proporcionar una señal detectable y reversible como respuesta a la unión con dicha GGBP mutante, y en el que dicha GGBP mutada tiene al menos una sustitución de aminoácido de un aminoácido no reactivo con un aminoácido reactivo que se puede modificar con un agente de marcaje análogo al etiquetado de cisteína con un colorante reactivo al tiol, y dicha GGBP mutada se une a la glucosa y se encapsula en una matriz permeable al analito que comprende sol-geles aumentados con silicatos modificados orgánicamente o con hidrogeles reticulados covalentemente, o con combinaciones de los mismos.
2. El biosensor de la reivindicación 1, en el que el grupo indicador luminiscente es un marcador fluorescente o un marcador fosforescente.
3. El biosensor de la reivindicación 1 ó 2, en el que dicha sustitución de aminoácido se selecciona entre una cisteína en la posición 1, una serina en la posición 1, una cisteína en la posición 11, una cisteína en la posición 14, una cisteína en la posición 19, una cisteína en la posición 43, una cisteína en la posición 74, una cisteína en la posición 107, una cisteína en la posición 110, una cisteína en la posición 112, una cisteína en la posición 113, una cisteína en la posición 137, una cisteína en la posición 149, una cisteína en la posición 213, una cisteína en la posición 216, una cisteína en la posición 238, una cisteína en la posición 287, una cisteína en la posición 292, una cisteína en la posición 152, una cisteína en la posición 182, una cisteína en la posición 236 y una cisteína en la posición 296 correspondiente a las posiciones de aminoácidos de la GGBP de E. coli.
4. El biosensor de la reivindicación 3, en el que dicha sustitución de aminoácido se selecciona entre una cisteína en la posición 1, una serina en la posición 1, una cisteína en la posición 11, una cisteína en la posición 14, una cisteína en la posición 19, una cisteína en la posición 43, una cisteína en la posición 74, una cisteína en la posición 107, una cisteína en la posición 110, una cisteína en la posición 112, una cisteína en la posición 113, una cisteína en la posición 137, una cisteína en la posición 149, una cisteína en la posición 213, una cisteína en la posición 216, una cisteína en la posición 238, una cisteína en la posición 287, una cisteína en la posición 292, una cisteína en la posición 152, una cisteína en la posición 182, una cisteína en la posición 236 y una cisteína en la posición 296 de la GGBP de E. coli.
5. El biosensor de la reivindicación 3 que comprende al menos dos sustituciones de aminoácidos seleccionadas entre una cisteína en la posición 112 and una serina en la posición 238, una cisteína en la posición 149 y una serina en la posición 238, una cisteína en la posición 152 y una cisteína en la posición 182, una cisteína en la posición 152 y una serina en la posición 213, una cisteína en la posición 213 y una cisteína en la posición 238, una cisteína en la posición 149 y una arginina en la posición 213, una cisteína en la posición 149 y una cisteína en la posición 213, una cisteína en la posición 149 y una treonina en la posición 213, una cisteína en la posición 149 y una leucina en la posición 213, una cisteína en la posición 149 y una tirosina en la posición 213, una cisteína en la posición 149 y una asparagina en la posición 223, una cisteína en la posición 149 y una cisteína en la posición 238, una cisteína en la posición 149 y una serina en la posición 256, una cisteína en la posición 149 y una arginina en la posición 256, una cisteína en la posición 152 y una arginina en la posición 213, una cisteína en la posición 152 y una asparagina en la posición 223, una cisteína en la posición 213 y una cisteína en la posición 255 de la GGBP de E. coli.
6. El biosensor de la reivindicación 3 que comprende al menos tres sustituciones de aminoácidos seleccionadas entre una cisteína en la posición 149, una serina en la posición 213 y una serina en la posición 238; una cisteína en la posición 149, una arginina en la posición 213 y una serina en la posición 238; una cisteína en la posición 149, una cisteína en la posición 213 y una cisteína en la posición 238; una cisteína en la posición 149, una serina en la posición 213 y una asparagina en la posición 223; y una cisteína en la posición 149, una asparagina en la en la posición 223 y una arginina en la posición 256 de GGBP de E. coli.
7. El biosensor de la reivindicación 3 que comprende al menos cuatro sustituciones de aminoácidos seleccionadas entre una serina en la posición 1, una cisteína en la posición 149, una arginina en la posición 213 y una serina en la posición 238; una serina en la posición 1, una cisteína en la posición 149, una serina en la posición 213 y una serina en la posición 238; y una cisteína en la posición 149, una cisteína en la posición 182, una cisteína en la posición 213 y una serina en la posición 238 de GGBP de E. coli.
8. El biosensor de una cualquiera de las reivindicaciones 3 a 7, en el que dicha proteína de unión comprende además al menos un marcador de histidina.
9. El biosensor de la reivindicación 1, en el que
(i) dicho marcador luminiscente tiene una longitud de onda de excitación de más de aproximadamente 600 nanómetros; o
(ii) dicho marcador luminiscente tiene una longitud de onda de emisión de más de aproximadamente 600 nanómetros; o
(iii) dicho marcador luminiscente es acoplado covalentemente a dicha al menos una proteína de unión a glucosa/galactosa mediante la reacción con un miembro seleccionado entre fluoresceína, coumarinas, rodaminas, tetrametilrodamina-5-yodoacetamida (5-TMRIA) , R-ficoeritrina acoplada a sal (9- (2 ( ó 4) - (N- (2-maleimidiletil) -sulfonamidil) -4 (ó 2) -sulfofenil) -2, 3, 6, 7, 12, 13, 16, 17-octahidro- (1H, 5H, 11H, 15H- xanteno (2, 3, 4-ij:5, 6, 7-i'j') diquinolizin-18-io (Quantum Red®) , sal 9- (2 (ó 4) - (N- (2-maleimidiletil) - sulfonamidil) -4 (ó 2) -sulfofenil) -2, 3, 5, 7, 2, 13, 16, 17-octahidro- (1H, 5H, 11H, 15H-xanteno (2, 3, 4-ij:5, 6, 7-i'j') diquinolizin-18-io (Texas Red®) , sal 2- (5- (1- (6- (N- (2-maleimidiletil) -amino) -6- oxohexil) -1 , 3-dihidro-3, 3-dimetil-5-sulfo-2H-indol-2-iliden) -1, 3-prop-ildienil) -1-etil-3, 3-dimetil-5-sulfo-3H-indolio (Cy3®) , N- ( (2-yodo-acetoxi) etil) -N-metil) amino-7-nitrobenzoxadiazol (IANBD) , 6-acriloil-2-dimetilaminonaftaleno (acr y lodan) , pireno, sal de ácido 6-amino-2, 3-dihidro-2- (2- ( (yodoacetil) amino) etil) -1, 3-dioxo-1H-benz (de) isoquinolin-5, 8-disulfónico (amarillo Lucifer) , sal 2- (5- (1- (6- (N- (2-male- imidiletil) -amino) -6-oxohexil) -1, 3-dihidro-3, 3-dimetil-5-sulfo-3H-indol-2-iliden) -1, 3-pentadienil) -1-etil-3, 3- dimetil-5-sulfo-3H-indolio (Cy5®) , (2-bromoacetamidoetil) -sulfonamida) : 4- (5- (4-dimetilaminofenil) oxazol-2-il) fenil - N- (2-bromoacetamidoetil) sulfonamida (Dapoxyl®) (2-bromoacetamido-etil) fulsfonamida, (N- (4, 4-difluoro-1, 3, 5, 7-tetrametil-4-bora-3a, 4a-diaza-s-indaceno-2-iliodoacetamida (BODIPY® 507/545 IA) , N- (4, 4-difluoro-5, 7-difenil-4-bora-3a, 4a-diaza-s-indaceno-3-propionil) -N-yodoacetil-etilen-diamina (BODIPY® 530/ 550 IA) , ácido 5- ( ( ( (2-yodacetil) -amino) etil) amino) naftalen-1-sulfónico (1, 5-IAEDANS) y carboxi-X-rodamina, 5/6-yodoacetamida (XRIA 5, 6) .
10. El biosensor de la reivindicación 1, en el que dicho analito es glucosa.
11. El biosensor de la reivindicación 1, en el que dicha matriz permeable al analito comprende un hidrogel reticulado covalentemente, siendo preferiblemente dicho hidrogel reticulado covalentemente seleccionado entre polipéptidos, polisacáridos, derivados de polisacárido, alcoholes polivinílicos, ácidos poliacrílicos, poliacrilamida, polietilenglicoles, copolímeros de estireno y anhídrido maleico, copolímeros de olefina y anhídrido maleico y copolímeros de viniléter y anhídrido maleico; siendo el hidrogel reticulado más preferible un alcohol polivinílico que comprende alcohol polivinílico y sales acetal de N-metil-4 (4'-formil-estiril) piridinio.
12. El biosensor de la reivindicación 1, en el que dicha matriz permeable al analito comprende sol-gel aumentado con silicatos modificados orgánicamente, preferiblemente, dicha matriz permeable al analito es sol-gel derivado de condensado de silicato modificado con glicerol (GMSC) .
13. El biosensor de una cualquiera de las reivindicaciones 1 a 12, en el que la GGBP encapsulada en la matriz está contenida en una matriz adicional, siendo, preferiblemente, dicha matriz adicional un hidrogel, sol-gel o una membrana de diálisis.
14. El biosensor de cualquiera de las reivindicaciones 1 a 13 que es un dispositivo implantable.
15. El biosensor de cualquier reivindicación 1 a 14 para su uso como dispositivo de diagnóstico.
16. Un procedimiento para la detección de la glucosa que comprende:
a) proporcionar al menos una proteína de unión a glucosa/galactosa mutada (GGBP) que tenga al menos una sustitución de aminoácido de un aminoácido no reactivo con un aminoácido reactivo que se pueda modificar con un agente de marcaje análogo al marcaje de la cisteína con un colorante reactivo al tiol y al menos un grupo indicador luminiscente unido covalentemente a la misma, en el que dicho al menos un grupo indicador luminiscente es capaz de proporcionar una señal detectable y reversible como respuesta a la unión a dicha GGBP mutante, y en el que dicha GGBP mutada se une a glucosa;
b) atrapar o encapsular dicha proteína de unión a glucosa/galactosa mutada en una matriz permeable al analito que comprende hidrogeles reticulados covalentemente, sol-geles aumentados con silicatos modificados orgánicamente o una combinación de los mismos;
c) exponer dicha proteína de unión a glucosa/galactosa mutada a concentraciones de glucosa variables; y d) detectar una señal procedente de dicho grupo indicador luminiscente.
17. El procedimiento de la reivindicación 16
(i) que comprende además la etapa de exponer dicho grupo indicador luminiscente a una fuente de energía capaz de excitar dicho grupo indicador para que emita dicha señal; o
(ii) en el que dicha detección comprende la detección de la señal reversible correspondiente a dichas concentraciones variables de glucosa; o
(iii) en el que dicha detección es continua, programada, episódica o combinaciones de las mismas.
18. El procedimiento de la reivindicación 16 ó 17, en el que dicha proteína de unión a glucosa/galactosa y/o dicho indicador son como se definen en las reivindicaciones 3 a 9.
19. Una composición que comprende una mezcla de:
a) una proteína de unión a glucosa/galactosa mutada (GGBP) que tiene al menos una sustitución de aminoácido seleccionada entre una cisteína en la posición 1, una serina en la posición 1, una cisteína en la posición 11, una cisteína en la posición 14, una cisteína en la posición 19, una cisteína en la posición 43, una cisteína en la posición 74, una cisteína en la posición 107, una cisteína en la posición 110, una cisteína en la posición 112, una cisteína en la posición 113, una cisteína en la posición 137, una cisteína en la posición 149, una cisteína en la posición 213, una cisteína en la posición 216, una cisteína en la posición 238, una cisteína en la posición 287, una cisteína en la posición 292, una cisteína en la posición 152, una cisteína en la posición 182, una cisteína en la posición 236 y una cisteína en la posición 296 correspondiente a las posiciones de aminoácidos de la GGBP de E. coli, en la que dicha GGBP mutada se une a la glucosa;
b) una matriz permeable al analito que comprende hidrogeles reticulados covalentemente, sol-geles aumentados con silicatos modificados orgánicamente o una combinación de los mismos; y c) al menos un grupo indicador luminiscente (marcador luminiscente) en el que dicho al menos un grupo indicador luminiscente es capaz de proporcionar una señal detectable y reversible como respuesta a la unión a dicha GGBP mutante.
20. La composición de la reivindicación 19, en la que dicha proteína de unión comprende al menos una combinación de sustituciones de aminoácidos seleccionada entre una cisteína en la posición 112 y una serina en la posición 238; una cisteína en la posición 149 y una serina en la posición 238; una cisteína en la posición 152 y una cisteína en la posición 182; una cisteína en la posición 152 y una serina en la posición 213; una cisteína en la posición 213 y una cisteína en la posición 238; una cisteína en la posición 149 y an arginina en la posición 213; una cisteína en la posición 149 y una cisteína en la posición 213; una cisteína en la posición 149 y una treonina en la posición 213; una cisteína en la posición 149 y una leucina en la posición 213; una cisteína en la posición 149 y una tirosina en la posición 213; una cisteína en la posición 149 y una asparagina en la posición 223; una cisteína en la posición 149 y una cisteína en la posición 238; una cisteína en la posición 149 y una serina en la posición 256; una cisteína en la posición 149 y una arginina en la posición 256; una cisteína en la posición 152 y una arginina en la posición 213; una cisteína en la posición 152 y una asparagina en la posición 223; una cisteína en la posición 213 y una cisteína en la posición 255; una cisteína en la posición 149, una serina en la posición 213 y una serina en la posición 238; una cisteína en la posición 149, una arginina en la posición 213 y una serina en la posición 238; una cisteína en la posición 149, una cisteína en la posición 213 y una cisteína en la posición 238; una cisteína en la posición 149, una serina en la posición 213 y una asparagina en la posición 223; una cisteína en la posición 149, una asparagina en la posición 223 y una arginina en la posición 256; una serina en la posición 1, una cisteína en la posición 149, una arginina en la posición 213 y una serina en la posición 238; una serina en la posición 1, una cisteína en la posición 149, una serina en la posición 213 y una serina en la posición 238; y una cisteína en la posición 149, una cisteína en la posición 182, una cisteína en la posición 213 y una serina en la posición 238 correspondientes a las posiciones de aminoácidos de la GGBP de E. coli.
21. El biosensor de la reivindicación 19 ó 20, en el que dicha proteína de unión a glucosa/galactosa mutada comprende además al menos un marcador de histidina.
22. La composición de la reivindicación 19 ó 20, en la que
(i) dicho marcador luminiscente tiene una longitud de onda de excitación de más de aproximadamente 600 nanómetros; o
(ii) dicho marcador luminiscente tiene una longitud de onda de emisión de más de aproximadamente 600 nanómetros; o
(iii) dicho marcador luminiscente es acoplado covalentemente a dicha al menos una proteína de unión a glucosa/galactosa mediante la reacción con un miembro seleccionado entre fluoresceína, cumarinas, rodaminas, tetrametilrodamina-5-yodoacetamida (5-TMRIA) , R-ficoeritrina acoplada a sal (9- (2 ( ó 4) - (N- (2-maleimidiletil) -sulfonamidil) -4 (ó 2) -sulfofenil) -2, 3, 6, 7, 12, 13, 16, 17-octahidro- (1H, 5H, 11H, 15H -xanteno (2, 3, 4-ij:5, 6, 7-i'j') diquinolizin-18-io (Quantum Red®) , sal 9- (2 (ó 4) - (N- (2-maleimidiletil) -s ulfonamidil) -4 (ó 2) -sulfofenil) -2, 3, 5, 7, 12, 13, 16, 17-octahidro- (1H, 5H, 11H, 15H-xanteno (2, 3, 4-ij:5, 6, 7-i'j') diquinolizin-18-io (Texas Red®) , sal 2- (5- (1- (6- (N- (2-maleimidiletil) - amino) -6-oxohexil) - 1 , 3- dihidro - 3, 3-dimetil-5-sulfo-2H-indol-2-iliden) -1, 3-propildienil) -1-etil-3, 3-dimetil-5-sulfo-3H-indolio (Cy3®) N- ( (2-yodo- acetoxi) etil) - N - metil) amino - 7-nitrobenzoxadiazol (IANBD) , 6-acriloil-2-dimetilaminonaftaleno (acr y lodan) , pireno, sal de ácido 6- amino - 2, 3-dihidro-2- (2- ( (yodoacetil) amino) etil) -1, 3-dioxo-1H-benz (de) isoquinolin-5, 8-disulfónico (amarillo Lucifer) , sal 2- (5- (1- (6- (N- (2-male- imidiletil) -amino) -6-oxohexil) -1, 3-dihidro-3, 3-dimetil-5-sulfo-3H-indol-2-iliden) -1, 3-pentadienil) -1-etil-3, 3- dimetil-5-sulfo - 3H - indolio (Cy5®) , (2 - bromoacetamidoetil - sulfonamida) 4- (5- (4-dimetilaminofenil) oxazol-2-il) fenil-N- (2-bromoacetamidoetil) sulfonamida (Dapoxyl®) (2-bromoacetamido-etil) fulsfonamida, (N- (4, 4-difluoro-1, 3, 5, 7-tetrametil-4-bora-3a, 4a-diaza-s-indaceno-2-iliodo-acetamida (BODIPY® 507/545 IA) , N- (4, 4-difluoro-5, 7-difenil-4-bora-3a, 4a-diaza-s-indaceno-3-propionil) -N-yodoacetil-etilen-diamina (BODIPY® 530/ 550 IA) , ácido 5- ( ( ( (2-yodacetil) -amino) etil) amino) naftalen-1-sulfónico (1, 5-IAEDANS) y carboxi-X-rodamina, 5/6-yodoacetamida (XRIA 5, 6) .
23. La composición de la reivindicación 19, en la que dicha matriz permeable al analito es como se define en las reivindicaciones 11 a 12.
Patentes similares o relacionadas:
Métodos para purificar una proteína objetivo de una o más impurezas en una muestra, del 17 de Junio de 2020, de EMD Millipore Corporation: Un metodo para purificar una proteina objetivo que contiene una region Fc de una o mas impurezas en una muestra, el metodo comprende las etapas de: a) poner en contacto […]
Arginina desiminasa con reactividad cruzada reducida hacia anticuerpos para ADI - PEG 20 para el tratamiento del cáncer, del 6 de Mayo de 2020, de TDW Group: Una composición terapéutica que comprende una arginina desiminasa (ADI) aislada y un vehículo farmacéuticamente aceptable, en donde la ADI aislada comprende la secuencia de […]
Redes moleculares, del 29 de Abril de 2020, de Sienna Cancer Diagnostics Inc: Una estructura de red molecular multipolimérica estratificada que tiene múltiples capas con múltiples especies de agentes de captura unidas […]
Composiciones y métodos para la distribución de moléculas en células vivas, del 25 de Marzo de 2020, de THE TEXAS A & M UNIVERSITY SYSTEM: Un compuesto que tiene la fórmula: **(Ver fórmula)** En donde X es un resto de unión, Y es un residuo de aminoácido acoplado de forma covalente […]
Vacuna peptídica para la prevención e inmunoterapia de demencia del tipo Alzheimer, del 4 de Marzo de 2020, de UNITED BIOMEDICAL, INC.: Una composición que comprende una combinación de construcciones de inmunógenos de péptidos Aβ que consiste en las secuencias de aminoácidos de SEQ […]
Péptidos, dispositivos y procedimientos para la detección de anticuerpos de Anaplasma, del 20 de Noviembre de 2019, de Abaxis, Inc: Una composición que comprende una población de péptidos aislados, comprendiendo dicha población tres o más péptidos diferentes, en la que cada péptido en la población […]
Anticuerpos humanos que se unen al gen de activación de linfocitos 3 (LAG-3) y sus usos, del 6 de Noviembre de 2019, de E. R. Squibb & Sons, L.L.C: Un anticuerpo monoclonal humano aislado, o una porción de unión a antígeno del mismo, que se une al gen de activación de linfocitos humanos 3 (LAG-3) y se une […]
Métodos y composiciones para el tratamiento de la esclerosis múltiple y trastornos asociados, del 14 de Mayo de 2019, de UTI Limited Partnership: Un complejo de nanopartícula que comprende: un núcleo de nanopartícula; una serie de complejos de antígeno- MHC (pMHC) que comprenden una serie de proteínas […]