PROCEDIMIENTO PARA LA SINTESIS DE FRAGMENTOS COLEADOS DE DNA MONOCATENARIO A PARTIR DE UN VECTOR DE EXPRESION Y UN CEBADOR UNIVERSAL, Y SU USO EN HIBRIDACION IN SITU.
Procedimiento para la síntesis de fragmentos coleados de DNA monocatenario a partir de un vector de expresión y un cebador universal,
y su uso en hibridación in situ.
El objeto de la presente invención refiere a un procedimiento para la obtención de sondas de DNA monocatenario según un primer paso de amplificación por PCR del fragmento escogido, seguido de su clonación en un vector de expresión. Una vez obtenido el vector puede almacenarse en una bacteria hospedadora.
A partir del vector se amplifica el inserto utilizando dos cebadores complementarios a los genes del propio vector que lo flanquean. Esta PCR produce un segmento de dsDNA que contiene el inserto flanqueado a ambos extremos por secuencias adicionales de la longitud deseada.
Empleando este producto como molde se realiza una segunda PCR empleando uno solo de los cebadores anteriores y oligonucleótidos marcados. Como resultado se obtienen sondas de DNA monocatenario con una cola en cada extremo; esta cola no será capaz de hibridar con la secuencia diana pero sí permitirá una mayor señal al aumentar la cantidad total de nucleótidos marcados
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P200702523.
Solicitante: CENTRO DE BIOLOGIA MOLECULAR CENBIMO, S.L.
Nacionalidad solicitante: España.
Provincia: LUGO.
Inventor/es: ALBA LOSADA,JESUS.
Fecha de Solicitud: 26 de Septiembre de 2007.
Fecha de Publicación: .
Fecha de Concesión: 23 de Junio de 2010.
Clasificación Internacional de Patentes:
- C12P19/34 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
- C12Q1/68M10
Clasificación PCT:
- C12P19/34 C12P 19/00 […] › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
Procedimiento para la síntesis de fragmentos coleados de DNA monocatenario a partir de un vector de expresión y un cebador universal, y su uso en hibridación in situ.
Campo técnico de la invención
La presente invención se engloba dentro del campo de la síntesis de fragmentos de DNA monocatenario y sus aplicaciones.
Estado de la técnica
Desde los primeros experimentos de hibridación (Pardue ML and Gall JG. Proc. Natl. Acad. Sci USA 64: 600-604 [1969]) se observó que la hibridación in situ era una de las técnicas que más hartan avanzar el conocimiento científico de todas las ciencias biológicas.
Mediante esta técnica se puede conseguir visualizar la presencia de genes y secuencias sobre el DNA tanto en cromosomas extendidos como durante la interfase. También mediante esta técnica es posible visualizar los genes que se expresan en una célula determinada y durante una fase concreta de su ciclo biológico; lo cual establece una ventaja sobre las técnicas de procesamiento de "arrays" de nucleótidos ya que éstas no permiten determinar con exactitud qué genes del cultivo expresan en cada momento.
La técnica de hibridación consiste en la obtención o fabricación de una cadena de ácido nucleico de determinada longitud que sea complementaria a la secuencia que se desea estudiar, bien sea DNA o RNA.
Las dos cadenas hibridan mediante la ley de pares de bases de Watson y Crick, es decir, que tanto la cadena diana como la cadena fabricada o "sonda" tienen que ser monocatenarias.
El ácido ribonucleico (RNA) en condiciones habituales se encuentra en forma monocatenaria. Existen diversas técnicas para la fabricación de sondas de RNA (Sambrook and Russell. Molecular Cloning, vol 2:9.29. 3ª Edición. CSHL PRESS), aunque la tecnología es compleja, consume tiempo y el rendimiento en cuanto a cantidad no es muy alto.
En cuanto a la producción de sondas de DNA monocatenario (ssDNA), varios autores han reportado el sistema utilizado en el presente estudio y que permite obtener la mayor eficiencia en el estado actual de la técnica.
Kitazawa (In situ hybridization with polymerase chain reaction-derived single-stranded DNA probe and S 1 nuclease. Histochemistry Cell Biology, 1998, Vol 111), describe una preparación del fragmento de DNA molde de doble cadena (dsDNA) a partir de una reacción de PCR convencional seguida de otra PCR lineal. Esta segunda PCR está alimentada por un único cebador específico que dirige la amplificación unidireccional de una sola de las hebras del DNA molde amplificado. El uso en esta segunda etapa de desoxinucleótidos marcados (digoxigenina-11-dUTP) permite la obtención de sondas de hibridación ssDNA que pueden ser utilizadas para la detección in situ de mRNA específico en muestras de tejidos biológicos mantenidos en parafina.
Antes, Konat G.W. ("Generation of high efficiency ssDNA hybridization probes by linear polymerase chain reaction". Scam-ling Micros. Suppl., 1996, Vol.10), ya había puesto a punto el método de obtención de sondas con nucleótidos marcados con radiactividad o con digoxigenina (dig-11-dUTP) para poder ser utilizadas en muestras de tejidos biológicos.
An S.F. et al. ("Generation of digoxigenin-labelled double-stranded and single-stranded probes using the polymerase chain reaction", Mol. Cell. Probes, 1992, Vol. 6. Nr. 3), obtiene sondas específicas marcadas con biotina o digoxigenina para hibridar con el virus de la hepatitis B (HBV), y su utilización en tejido de biopsia hepática.
El procedimiento había sido originalmente descrito por Finchk U. et al. ("Producing single-stranded DNA probes with the Taq DNA polymerase: a high yield protocol". Biotechniques, 1991, Vol. 10, No 1), en el que documentan el método de la doble PCR y el marcaje de la segunda con biotina-11-dUTP y biotina-21-dUTP.
El sistema más extendido para la conservación de cultivos de tejidos es mantenerlos en parafina. Pero este sistema plantea el problema de que destruye la mayor parte de los ácidos nucleicos; el mRNA queda reducido a una cantidad aproximada de la décima parte y suele estar además deteriorado. Esto dificulta la hibridación mediante sondas e impide que éstas puedan ser más resolutivas.
La presente invención aporta una solución a la potenciación de señal utilizando sondas cromáticas para la detección de secuencias de mRNA.
Descripción de la invención
La presente invención trata de un procedimiento para la síntesis de fragmentos coleados de DNA monocatenario marcados con un hapteno, obtenidos con PCR asimétrica utilizando un cebador universal. Este cebador reconoce una secuencia plasmídica vecina a un inserto y hace factible su utilización "in situ" como sonda sobre tejidos fijados en formol e incluidos en parafina.
Así, la presente invención se refiere a un procedimiento para la síntesis de sondas coleadas de DNA monocatenario, caracterizado porque comprende:
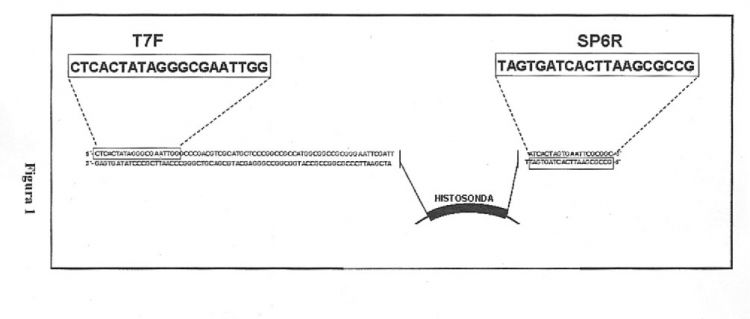
En una realización concreta de la invención, el vector de expresión del paso 1 es el plásmido PG&T. En una realización preferida de la invención, el inserto se sitúa entre las secuencias T7F y SP6R del plásmido. En una realización más preferida de la invención, los cebadores del paso 2 son dos oligos complementarios a dichas secuencias T7F y SP6R.
Tras la amplificación del fragmento comprendido entre los dos cebadores según el paso 2 se obtiene un DNA de doble cadena (dsDNA). Dicho dsDNA comprende la secuencia problema junto con una secuencia adicional de nucleótidos que corresponde originalmente al vector.
En una realización concreta de la invención, el producto de esta primera amplificación sirve de molde para una segunda amplificación por PCR. En una realización preferida de la invención, dicha segunda amplificación es una PCR asimétrica en la que sólo se utiliza uno de los cebadores utilizados en la amplificación anterior.
En una realización concreta de la invención, en la segunda PCR se introducen nucleótidos marcados. En otra realización concreta, el nucleótido marcado es la desoxiuridina (dUTP). En una realización concreta de la invención, se marca con un hapteno. En otra realización concreta el hapteno está seleccionado entre digoxigenina, biotina o fluoresceína.
En una realización preferente de la invención, en la segunda amplificación se introduce dUTP marcada con digoxigenina.
El resultado de esa segunda amplificación asimétrica es un DNA monocatenario que contiene la secuencia problema junto con una cola de secuencia propia del plásmido. Esa cola de secuencia no es hibridante pero sí está marcada con nucleótidos de uridina como lo está todo el producto de esta amplificación, de forma que actúa como potenciadora de la señal de la secuencia que hibrida.
En una realización preferente de la invención, los fragmentos de DNA marcados se utilizan como sonda para la detección de expresión génica; entendiéndose como "sonda" un fragmento de DNA empleado para detectar e identificar secuencias correspondientes en ácidos nucleicos mediante su hibridación selectiva con ellas.
Así, la presente invención se refiere también a una sonda de DNA monocatenario obtenida por el procedimiento descrito. En una realización concreta de la presente invención, dicha sonda de DNA monocatenario reconoce mRNA que se expresa en una célula diana, tal como se enuncia en el "Estado de la Técnica" de la presente descripción.
En un aspecto concreto, la presente invención se refiere al uso de la sonda de DNA monocatenario previamente definida en un método para la detección de la expresión génica mediante hibridación in situ. En una realización preferente, dicha hibridación se realiza sobre cortes de tejidos fijados en formol e incluidos en parafina. En una realización concreta de la invención, dicho método para la hibridación in situ emplea como tampón PBS a pH...
Reivindicaciones:
1. Un procedimiento para la síntesis de sondas coleadas de DNA monocatenario caracterizado porque comprende:
2. Un procedimiento para la síntesis de fragmentos de DNA monocatenario según la reivindicación 1, caracterizado porque el vector de expresión empleado corresponde al plásmido PG&T.
3. Un procedimiento para la síntesis de fragmentos de DNA monocatenario según las reivindicaciones 1 y 2, caracterizado porque los cebadores que se utilizan en el vector de expresión son oligos complementarios a los genes T7F y SP6R.
4. Un procedimiento para la síntesis de fragmentos de DNA monocatenario según la reivindicación 1, caracterizado porque el oligonucleótido del paso 3 contiene desoxiuridina (dUTP) marcada.
5. Un procedimiento para la síntesis de fragmentos de DNA monocatenario según las reivindicaciones 1 y 4, caracterizado porque dicho desoxinucleótido se encuentra marcado con un hapteno.
6. Un procedimiento para la síntesis de fragmentos de DNA monocatenario según la reivindicación 5 porque dicho hapteno está seleccionado entre digoxigenina, biotina o fluoresceína.
7. Un fragmento de DNA monocatenario caracterizado porque se obtiene por el procedimiento descrito en una de las reivindicaciones 1 a 6.
8. Un fragmento de DNA monocatenario según la reivindicación 7, caracterizado porque contiene una cola de nucleótidos no hibridante.
9. Un fragmento de DNA monocatenario según la reivindicación 8, caracterizado porque contiene una cola de nucleótidos no hibridante marcada.
10. Uso de un fragmento de DNA monocatenario descrito en una de las reivindicaciones 7 a 9, como sonda para la detección de expresión génica.
11. Una sonda de DNA monocatenario obtenida por el procedimiento descrito en las reivindicaciones 1 a 10.
12. Uso de una sonda definida en la reivindicación 11 en un método para la detección de la expresión génica mediante hibridación in situ.
13. Uso de una sonda según la reivindicación 12, caracterizado porque dicha hibridación se realiza sobre cortes de tejidos fijados en formol e incluidos en parafina.
14. Uso de una sonda según una de las reivindicaciones 12 ó 13, caracterizado porque dicho método para la hibridación in situ emplea un tampón de hibridación con los siguientes componentes:
Patentes similares o relacionadas:
Fitasa, del 6 de Mayo de 2020, de BASF Enzymes LLC: Un ácido nucleico aislado, recombinante o sintético que codifica un polipéptido que tiene actividad fitasa, en donde el ácido nucleico se selecciona del grupo que […]
Métodos de preparación de polinucleótidos usando composiciones de sales de catiónicas multivalentes, del 6 de Mayo de 2020, de GERON CORPORATION: Un método para preparar un polinucleótido, comprendiendo el método: a) poner en contacto una primera composición polinucleotídica con una sal catiónica […]
Preparación de muestras para la amplificación de ácidos nucleicos, del 8 de Abril de 2020, de ILLUMINA CAMBRIDGE LIMITED: Un método para preparar una muestra para la amplificación de bibliotecas y la amplificación posterior que comprende las siguientes etapas: (a) en una […]
Método para producir y purificar ARN, que comprende al menos una etapa de filtración de flujo tangencial, del 1 de Abril de 2020, de CureVac Real Estate GmbH: Método para producir y purificar ARN, que comprende las etapas de A) proporcionar ADN que codifica para el ARN; B) realizar la transcripción in vitro del […]
Método y composiciones para reducir productos de amplificación no específicos, del 25 de Marzo de 2020, de Paragon Genomics, Inc: Un método para reducir productos de amplificación no específicos de una reacción de extensión de cebadores dependiente de plantilla, comprendiendo el método: […]
Partículas de transducción no replicativas y sistemas indicadores basados en partículas de transducción, del 22 de Enero de 2020, de Geneweave Biosciences Inc: Un sistema de empaquetamiento de célula bacteriana para empaquetar una molécula de ácido nucleico indicadora en una partícula de transducción no […]
Composiciones terapéuticas y procedimientos que implican la transfección de ARNm, del 21 de Enero de 2020, de Herzberg, Mark C: ARNm que codifica un polipéptido implicado en la inmunidad innata, en el que el polipéptido es proteína antimicrobiana de catelicina (CAMP), calprotectina, […]
Fabricación y uso de ARN monocatenario sintetizado in vitro para introducción en células de mamífero para inducir un efecto biológico o bioquímico, del 4 de Diciembre de 2019, de CELLSCRIPT, LLC: Una composición de ARN tratado que comprende ARN monocatenario o ARNm sintetizado in vitro, en la que menos del 0,01 %, preferentemente menos del 0,001 […]