PROCEDIMIENTO PARA LA SECUENCIACION DE MOLECULAS DE ACIDO NUCLEICO.
Procedimiento de secuenciación de una sola molécula de ácido nucleico diana que presenta una pluralidad de bases nucleotídicas que comprende:
proporcionar un complejo de una enzima que polimeriza el ácido nucleico y la molécula de ácido nucleico diana para permitir la formación de una cadena de ácido nucleico en crecimiento complementario al ácido nucleico diana;
proporcionar una pluralidad de tipos de análogos nucleotídicos marcados, en el que cada tipo de análogo nucleotídico es complementario a un nucleótido diferente en la secuencia de ácido nucleico diana;
polimerizar la pluralidad de tipos de análogos nucleotídicos marcados incorporando la pluralidad de tipos de análogos nucleotídicos marcados en la cadena de ácido nucleico en crecimiento, en el que cada uno de los análogos incorporados puede polimerizarse con un análogo nucleotídico posterior para permitir la polimerización continua de una pluralidad de análogos nucleotídicos;
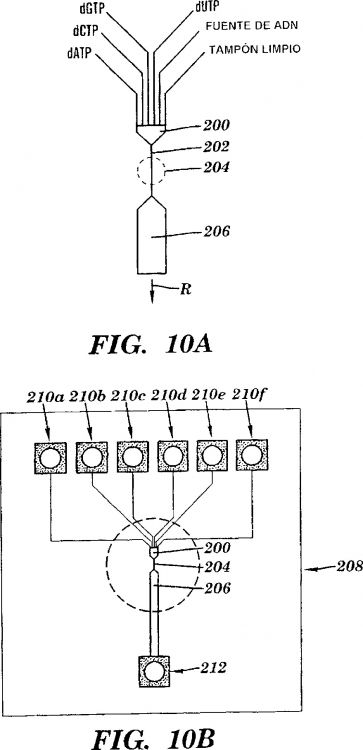
identificar la secuencia de tiempo de la incorporación de dicha pluralidad de tipos de análogos nucleotídicos marcados en la cadena de ácido nucleico en crecimiento detectando los tiempos de residencia de los análogos nucleotídicos marcados próximos a la enzima que polimeriza el ácido nucleico en tiempo real mientras que la extensión del cebador dirigida por la plantilla tiene lugar, y eliminándose el marcador tras la detección
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US00/13677.
Solicitante: CORNELL RESEARCH FOUNDATION, INC..
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 20 THORNWOOD DRIVE, SUITE 105,ITHACA, NY 14850.
Inventor/es: WEBB,WATT W., TURNER, STEPHEN, KORLACH,JONAS, LEVENE,MICHAEL, CRAIGHEAD,HAROLD,G, FOQUET,MATHIEU.
Fecha de Publicación: .
Fecha Concesión Europea: 5 de Mayo de 2010.
Clasificación PCT:
- C12M1/34 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12M EQUIPOS PARA ENZIMOLOGIA O MICROBIOLOGIA (instalaciones para la fermentación de estiércoles A01C 3/02; conservación de partes vivas de cuerpos humanos o animales A01N 1/02; aparatos de cervecería C12C; equipos para la fermentación del vino C12G; aparatos para preparar el vinagre C12J 1/10). › C12M 1/00 Equipos para enzimología o microbiología. › Medida o ensayo de detección de las condiciones del medio, p. ej. por contadores de colonias.
- C12P19/34 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
Fragmento de la descripción:
Procedimiento para la secuenciación de moléculas de ácido nucleico.
Campo de la invención
La presente invención se refiere a un procedimiento para determinar la secuencia de moléculas de ácido nucleico.
Antecedentes de la invención
El objetivo que consiste en aclarar el genoma humano completo ha creado un interés en las tecnologías para la secuenciación rápida del ADN, tanto en aplicaciones a pequeña como a gran escala. Los parámetros importantes son la velocidad de secuenciación, la longitud de la secuencia que puede leerse durante una sola serie de secuenciación y la cantidad de plantilla de ácido nucleico requerida. Estos retos en investigación sugieren aspirar a secuenciar la información genética de células aisladas sin ampliación previa, y sin necesidad previa de clonar el material genético en vectores de secuenciación. Los proyectos del genoma a gran escala son actualmente demasiado costosos para ser realizados de modo realista para un gran número de organismos o pacientes. Además, a medida que aumenta el conocimiento de las bases genéticas para las enfermedades humanas, existirá una necesidad siempre creciente de secuenciación de ADN preciso y de alto rendimiento que sea asequible para aplicaciones clínicas. Los procedimientos prácticos para determinar las secuencias de pares de bases de moléculas individuales de ácidos nucleicos, preferentemente con alta velocidad y longitudes largas de lectura, proporcionarían la capacidad de medición necesaria.
Dos técnicas tradicionales para secuenciar el ADN son el método de la terminación didesoxi de Sanger (Sanger et al., Proc. Natl. Acad. Sci. U.S.A. 74: 563-5467 (1977)) y el método de degradación química de Maxam-Gilbert (Maxam y Gilbert, Proc. Natl. Acad. Sci. U.S.A. 74: 560-564 (1977)). Ambos procedimientos suministran cuatro muestras que contiene cada una, una familia de cadenas de ADN en las que todas las cadenas terminan en el mismo nucleótido. La electroforesis en gel de bloque ultrafino, o más recientemente la electroforesis en matriz capilar se utilizan para resolver las cadenas de diferente longitud y determinar la secuencia de nucleótidos, ya sea etiquetando de manera diferente las cadenas de cada muestra antes de la electroforesis para indicar el nucleótido terminal o introduciendo las muestras en diferentes bandas del gel o en diferentes capilares. Tanto el procedimiento de Sanger como el de Maxam-Gilbert son laboriosos y ocupan tiempo, y requieren un pretratamiento dilatado de la fuente de ADN. Se han hecho intentos para hacer uso de la espectroscopia de masas para reemplazar la etapa de electroforesis intensiva. Para el estudio de tecnologías de secuenciación existentes, véase Cheng "High-Speed-Sequence Analysis", Progr. Biochem. Biophys. 22: 223-227(1995).
Se han desarrollado procedimientos afines que utilizan marcadores con colorantes o fluorescentes asociadas al nucleótido terminal, donde la determinación de la secuencia se realiza también por electroforesis en gel y detectores fluorescentes automáticos. Por ejemplo, el procedimiento de ampliación de Sanger ha sido modificado recientemente para su utilización en un sistema de microsecuenciación automático que requiere solamente volúmenes de submicrolitros de reactivos y trifosfatos de didesoxiribonucleótido marcados con colorante. En la patente US nº 5.846.727 de Soper et al., se lleva a cabo la detección por fluorescencia en chip con fibra óptica de un solo modo llevando la luz de excitación al canal capilar, y una segunda fibra óptica de un solo modo que recoge los fotones fluorescentes. Se estiman lecturas de las secuencias en el intervalo de 400 a 500 bases lo que no es una mejora significativa sobre la cantidad de información de secuencias obtenidas con los procedimientos de Sanger o de Maxam-Gilbert. Además, el procedimiento de Soper requiere la ampliación por RCP de la plantilla de ADN, y la purificación y la electroforesis en gel de los "escalones" de secuenciación de oligonucleótidos, antes del inicio de la reacción de separación. Estos sistemas requieren todos cantidades significativas de ADN diana. Incluso el procedimiento descrito en la patente US nº 5.302.509 de Cheeseman, que no utiliza electroforesis en gel para la determinación de secuencias, requiere por lo menos un millón de moléculas de ADN.
En una mejora reciente de una metodología de secuenciación por síntesis originalmente inventada hace diez años, la secuencias de ADN se deducen midiendo la liberación de pirofosfato en las pruebas de complejos de ADN/polimerasa con cada trifosfato de desoxirribonucleótido (dNTP) por separado y sucesivamente. Véase Ronaghi et al., "A Sequencing Method Based on Real-Time Pyrophosphate", Science 281: 363-365 (1998) y Hyman, "A New Method of Sequencing DNA", Anal. Biochem. 174: 423-436(1988). Aunque utilizando nucleótidos naturales, el procedimiento necesita la sincronización de las polimerasas en las cadenas de ADN, que limitan en gran medida las longitudes de lectura de las secuencias. Se consiguieron solo aproximadamente lecturas de 40 nucleótidos y no cabe esperar que el procedimiento de detección pueda aproximarse a la sensibilidad de una sola molécula debido a la eficacia cuántica limitada de la producción de luz por la luciferasa en el procedimiento presentado por Ronaghi et al., "A Sequencing Method Based on Real-Time Pyrophosphate", Science 281: 363-365(1998). Además, la velocidad de secuenciación global está limitada por las etapas necesarias de lavado, posteriores a las etapas químicas con objeto de identificar la presencia de pirofosfato, y por el tiempo intrínseco requerido para determinar cada par de bases que ha de secuenciarse con las cuatro bases sucesivamente. Además, se reconocieron dificultades para determinar con precisión los segmentos de homonucleótidos en las secuencias.
Los intentos previos para secuenciar moléculas aisladas (en general sin éxito pero originales) han utilizado exonucleasas para liberar sucesivamente bases individuales marcadas con fluorescencia como una segunda etapa una vez la ADN polimerasa ha formado una cadena complementaria completa. Véase Goodwin et al., "Application of Single Molecule Detection to DNA Sequencing", Nucleos. Nucleot. 16: 543-550 (1997). Consiste en sintetizar una cadena de ADN marcada con cuatro análogos diferentes de dNTP fluorescentes, después de la degradación de la cadena marcada por la acción de una exonucleasa, y la detección de las bases individuales liberadas en un detector de flujo hidrodinámico. Sin embargo, tanto la polimerasa como la exonucleasa tienen que mostrar actividad en una cadena de ADN muy modificada, y la generación de una cadena de ADN sustituida con cuatro análogos dNTP fluorescentes diferentes no se ha conseguido todavía. Véase Dapprich et al., "DNA Attachment to Optically Trapped Beads in Microstructures Monitored by Bead Displacement", Bioimaging 6: 25-32 (1998). Además, se conoce información poco precisa acerca de la relación entre el grado de marcado del ADN y la inhibición de la actividad de la exonucleasa. Véase Dörre et al., "Techniques for Single Molecule Sequencing", Bioimaging 5: 139-152 (1997).
En una segunda estrategia que utiliza exonucleasas, se digiere ADN natural mientras está siendo impulsado hacia una película líquida delgada con objeto de separar espacialmente los nucleótidos escindidos. Véase Dapprich et al., "DNA Attachment to Optically Trapped Beads in Microstructures Monitored by Bead Displacement", Bioimaging 6:25-33 (1998). Éstas a continuación se difunden a corta distancia antes de llegar a inmovilizarse en una superficie para la detección. Sin embargo, la mayoría de las exonucleasas presentan tasas de escisión dependientes de la secuencia y de la estructura, dando como resultado dificultades en el análisis de los datos e igualando conjuntos de secuencias parciales. Además, las formas de identificar las bases en la superficie de detección tienen que desarrollarse o mejorarse todavía.
Independientemente del sistema de detección, los procedimientos que utilizan exonucleasas no han sido desarrollados en los procedimientos que satisfacen la demanda actual de secuenciación rápido y de alto rendimiento. Además, la mayoría de las exonucleasas presentan velocidades de renovación relativamente lentas, y los procedimientos propuestos requieren pretratamiento dilatado, marcado y posterior inmovilización de la plantilla de ADN sobre el lecho en la corriente en circulación del fluido, todo lo cual convierte una realización más complicada en un sistema sencillo de alto rendimiento.
Reivindicaciones:
1. Procedimiento de secuenciación de una sola molécula de ácido nucleico diana que presenta una pluralidad de bases nucleotídicas que comprende:
proporcionar un complejo de una enzima que polimeriza el ácido nucleico y la molécula de ácido nucleico diana para permitir la formación de una cadena de ácido nucleico en crecimiento complementario al ácido nucleico diana;
proporcionar una pluralidad de tipos de análogos nucleotídicos marcados, en el que cada tipo de análogo nucleotídico es complementario a un nucleótido diferente en la secuencia de ácido nucleico diana;
polimerizar la pluralidad de tipos de análogos nucleotídicos marcados incorporando la pluralidad de tipos de análogos nucleotídicos marcados en la cadena de ácido nucleico en crecimiento, en el que cada uno de los análogos incorporados puede polimerizarse con un análogo nucleotídico posterior para permitir la polimerización continua de una pluralidad de análogos nucleotídicos;
identificar la secuencia de tiempo de la incorporación de dicha pluralidad de tipos de análogos nucleotídicos marcados en la cadena de ácido nucleico en crecimiento detectando los tiempos de residencia de los análogos nucleotídicos marcados próximos a la enzima que polimeriza el ácido nucleico en tiempo real mientras que la extensión del cebador dirigida por la plantilla tiene lugar, y eliminándose el marcador tras la detección.
2. Procedimiento según la reivindicación 1, en el que se identifica una pluralidad de análogos nucleotídicos incorporados por segundo.
3. Procedimiento según la reivindicación 2, en el que se identifican hasta 100 análogos incorporados por segundo.
4. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico se selecciona de entre el grupo constituido por una ADN polimerasa, una ARN polimerasa, transcriptasa inversa y mezclas de las mismas.
5. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico es una polimerasa termoestable.
6. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico es una polimerasa termodegradable seleccionada de entre el grupo constituido por ADN polimerasa de E. coli, el fragmento de Klenow de la ADN polimerasa de E. coli, T4 ADN polimerasa y T7 ADN polimerasa.
7. Procedimiento según la reivindicación 1, en el que la molécula de ácido nucleico diana se selecciona de entre el grupo constituido por ADN bicatenario, ADN monocatenario, horquillas de ADN monocatenario, híbridos de ADN/ARN, ARN con una zona de reconocimiento para la unión de una ARN polimerasa y horquillas de ARN.
8. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico está unida al complejo con la molécula del ácido nucleico diana en un origen de replicación, en una hendidura o separación en un ácido nucleico diana bicatenario, una estructura secundaria en un ácido nucleico diana monocaternario, una zona de unión creada por un proteína accesoria seleccionada de entre una proteína de enlace monocatenaria, una primasa, y una helicasa o a un ácido nucleico monocatenario cebado.
9. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico está provista de una o más proteínas accesorias seleccionadas de entre una proteína de enlace monocatenaria, una primasa, y una helicasa para modificar su actividad.
10. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico es procesiva.
11. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico es no procesiva.
12. Procedimiento según la reivindicación 1, en el que los análogos nucleotídicos se seleccionan de entre el grupo constituido por un ribonucleótido, un desoxirribonucleótido, un ribonucleótido modificado, un desoxirribonucleótido modificado, un nucleótido peptídico, un nucleótido peptídico modificado y un nucleótido de esqueleto central de fosfato-azúcar modificado.
13. Procedimiento según la reivindicación 1 que comprende además:
hibridar un cebador oligonucleotídico a la molécula de ácido nucleico diana antes de o durante dicha provisión de una pluralidad de análogos nucleotídicos.
14. Procedimiento según la reivindicación 13, en el que el cebador oligonucleotídico comprende nucleótidos seleccionados de entre el grupo constituido por ribonucleótidos, desoxirribonucleótidos, ribonucleótidos modificados, desoxirribonucleótidos modificados, ácidos nucleicos peptídicos, ácidos nucleicos peptídicos modificados y nucleótidos de esqueleto de fosfato-azúcar modificado.
15. Procedimiento según la reivindicación 1, en el que cada uno de los análogos nucleotídicos incorporados esta provisto de un marcador.
16. Procedimiento según la reivindicación 1, en el que el marcador se selecciona de entre el grupo constituido por cromóforos, restos fluorescentes, enzimas, antígenos, metales pesados, sondas magnéticas, colorantes, grupos fosforescentes, materiales radioactivos, restos quimioluminiscentes, nanopartículas dispersantes o fluorescentes, restos generadores de la señal Raman, y restos de detección electroquímica.
17. Procedimiento según la reivindicación 1, en el que el marcador se acopla al análogo nucleotídico en su base, resto de azúcar, alfa fosfato, beta fosfato o gamma fosfato.
18. Procedimiento según la reivindicación 1, en el que el marcador se acopla al análogo nucleotídico con un enlazador.
19. Procedimiento según la reivindicación 1, en el que el marcador se acopla al análogo nucleotídico en su terminal fosfato.
20. Procedimiento según la reivindicación 1, que comprende además:
retirar el marcador del análogo nucleotídico durante o después de dicha identificación y antes de dicha polimerización de un análogo nucleotídico posterior en una zona activa en la que el análogo nucleotídico se está añadiendo a la cadena cadena de ácido nucleico en crecimiento.
21. Procedimiento según la reivindicación 20, en el que dicha retirada se lleva a cabo blanqueando el marcador.
22. Procedimiento según la reivindicación 21, en el que dicho blanqueo se lleva a cabo por fotoblanqueo con radiación que está ajustada para provocar una retirada del marcador de referencia.
23. Procedimiento según la reivindicación 20, en el que dicha retirada se lleva a cabo por escisión del marcador del análogo nucleotídico.
24. Procedimiento según la reivindicación 23, en el que los análogos nucleotídicos marcados con beta-fosfato o con gamma-fosfato se escinden con enzimas.
25. Procedimiento según la reivindicación 1, en el que cada uno de la pluralidad de tipos de análogos nucleotídicos presentan diferentes marcadores que se distinguen entre sí durante dicha identificación.
26. Procedimiento según la reivindicación 1, en el que tres o menos de la pluralidad de tipos de análogos nucleotídicos presentan un marcador diferente.
27. Procedimiento según la reivindicación 1, en el que los diferentes tipos de análogos nucleotídicos presentan el mismo marcador pero se distinguen por las propiedades diferentes debido a la presencia de fluoróforos de base, fluoróforos extinguidos o análogos nucleotídicos fluorógenos.
28. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico es portadora de un marcador y dicha identificación se lleva a cabo detectando la interacción entre el marcador y el análogo nucleotídico.
29. Procedimiento según la reivindicación 28, en el que el marcador es un donador o un aceptor de transferencia de energía por resonancia de fluorescencia.
30. Procedimiento según la reivindicación 1, en el que dicha identificación se lleva a cabo por procedimientos ópticos seleccionados de entre el grupo constituido por microespectroscopia en campo lejano, microespectroscopia en campo próximo, iluminación guiada por ondas evanescentes o por ondas, reforzamiento de nanoestructuras y las combinaciones de los mismos.
31. Procedimiento según la reivindicación 1, en el que dicha identificación se lleva a cabo utilizando la excitación monofotónica o multifotónica, la transferencia de energía por resonancia de fluorescencia o la fotoconversión.
32. Procedimiento según la reivindicación 1, en el que dicha identificación se consigue mediante discriminación de las longitudes de onda del espectro, medición y separación de los tiempos de permanencia de la fluorescencia, identificación de fluoróforos y/o supresión del fondo.
33. Procedimiento según la reivindicación 30, en el que la identificación del fluoróforo y/o la supresión del fondo utilizan la conexión rápida entre los modos de excitación y las fuentes de iluminación y las combinaciones de los mismos.
34. Procedimiento según la reivindicación 1, en el que dicha provisión de un complejo comprende:
disponer (1) un primer cebador oligonucleotídico o (2) la molécula de ácido nucleico diana en un soporte;
hibridar (1) la molécula de ácido nucleico diana con el primer cebador oligonucleotídico o (2) el primer cebador oligonucleotídico con la molécula de ácido nucleico diana dispuesta, para formar un complejo con la molécula de ácido nucleico diana cebada; y
proporcionar la enzima que polimeriza el ácido nucleico en el complejo con la molécula de ácido nucleico diana cebada.
35. Procedimiento según la reivindicación 34, en el que un segundo cebador oligonucleotídico se hibrida con la molécula de ácido nucleico diana en la zona opuesta a la hibridada por el primer cebador oligonucleotídico.
36. Procedimiento según la reivindicación 34, en el que el soporte y el cebador oligonucleotídico o la molécula de ácido nucleico diana se unen de manera reversible o irreversible con los componentes correspondientes de un par de enlace covalente o no covalente seleccionado de entre el grupo constituido por un par de enlace antígeno-anticuerpo, un par de enlace estreptavidina-biotina, moléculas de acoplamiento fotoactivadas, y un par de ácidos nucleicos complementarios.
37. Procedimiento según la reivindicación 34, en el que el cebador oligonucleotídico está dispuesto sobre el soporte y la molécula de ácido nucleico diana está hibridada con el cebador oligonucleotídico dispuesto.
38. Procedimiento según la reivindicación 34, en el que la molécula de ácido nucleico diana está dispuesta sobre el soporte y el cebador oligonucleotídico está hibridado con la molécula de ácido nucleico diana dispuesta.
39. Procedimiento según la reivindicación 1, en el que dicha provisión de un complejo comprende:
disponer, sobre un soporte, una molécula de ácido nucleico bicatenario que comprende el ácido nucleico diana y que presenta una zona de reconocimiento próxima a una zona activa en la que se está añadiendo el análogo nucleotídico a la cadena de ácido nucleico en crecimiento, y
proporcionar la enzima que polimeriza ácido nucleico sobre la molécula de ácido nucleico diana.
40. Procedimiento según la reivindicación 1, en el que dicha provisión de un complejo comprende:
disponer una enzima que polimeriza ácido nucleico sobre un soporte en una posición adecuada para que el complejo con ácido nucleico diana se desplace en relación a la enzima que polimeriza ácido nucleico.
41. Procedimiento según la reivindicación 40, en el que el soporte y la enzima que polimeriza el ácido nucleico se unen de manera reversible o irreversible con los componentes correspondientes de un par de enlace covalente o no covalente seleccionado de entre el grupo constituido por un par de enlace antígeno-anticuerpo, un par de enlace estreptavidina-biotina, moléculas de acoplamiento fotoactivadas y un par de ácidos nucleicos complementarios.
42. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico o el ácido nucleico diana se dispone sobre un soporte.
43. Procedimiento según la reivindicación 1, en el que la enzima que polimeriza el ácido nucleico o el ácido nucleico diana se dispone en un gel con poros.
44. Procedimiento según la reivindicación 1, en el que el ácido nucleico diana y la enzima que polimeriza el ácido nucleico se disponen sobre un soporte sólido.
45. Procedimiento según la reivindicación 1, en el que dicha identificación se lleva a cabo reduciendo el ruido de fondo resultante de los análogos nucleotídicos libres.
46. Procedimiento según la reivindicación 45, en el que dicha identificación comprende:
dirigir la radiación de activación a una zona que corresponde sustancialmente a una zona activa en la que se está añadiendo análogo nucleotídico a la cadena de ácido nucleico en crecimiento; y
detectar el análogo nucleotídico polimerizado en la zona activa.
47. Procedimiento según la reivindicación 45, en el que dicha identificación distingue análogos nucleotídicos polimerizados en la zona activa en la que se está añadiendo análogo nucleotídico a la cadena de ácido nucleico en crecimiento procedente de los análogos nucleotídicos libres.
48. Procedimiento según la reivindicación 45, en el que dicha identificación se lleva a cabo en una zona confinada correspondiente a la zona activa en la que se está añadiendo análogo nucleotídico a la cadena de ácido nucleico en crecimiento.
49. Procedimiento según la reivindicación 48, en el que dicha identificación se lleva a cabo en una nanoestructura.
50. Procedimiento según la reivindicación 49, en el que dicha nanoestructura es una nanoestructura punteada, acicular o resonante que aumenta dicha detección.
51. Procedimiento según la reivindicación 48, en el que los análogos nucleotídicos que no se polimerizan en la zona activa se desplazan rápidamente por una microestructura hasta y desde la zona confinada correspondiente a la zona activa.
52. Procedimiento según la reivindicación 51, en el que la microestructura comprende:
una pluralidad de canales para dirigir diferentes análogos nucleotídicos a la zona confinada y
un canal de descarga para permitir que los materiales se retiren de la zona confinada, y la nanoestructura comprende:
un alojamiento que define la zona confinada y se construye para facilitar dicha identificación.
53. Procedimiento según la reivindicación 45, en el que dicha identificación se lleva a cabo por intensificación del campo electromagnético con radiación electromagnética intensificándose en la proximidad a un objeto con un radio de curvatura pequeño adyacente a la zona activa.
54. Procedimiento según la reivindicación 45, en el que dicha identificación se lleva a cabo por iluminación en campo próximo de las cavidades en las que está dispuesta la molécula de ácido nucleico diana cebada.
55. Procedimiento según la reivindicación 45, en el que dicha identificación se lleva a cabo con fibras ópticas próximas al complejo.
56. Procedimiento según la reivindicación 45, en el que dicha identificación y dicha reducción del fondo se llevan a cabo por retardo controlado por tiempo de la detección de fotones.
57. Procedimiento según la reivindicación 1, en el que dicho procedimiento se lleva a cabo secuenciando diferentes moléculas de ácido nucleico diana en una variedad de ubicaciones diferentes en una matriz.
58. Procedimiento según la reivindicación 1, en el que dicho procedimiento se lleva a cabo secuenciando simultánea o secuencialmente el mismo ácido nucleico diana y combinando la salida de dicha secuenciación.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]