Procedimiento de preparación de proteínas triméricas.
Un procedimiento de preparación de una proteína trimérica que comprende cultivar una célula huésped transformada otransfectada con un vector de expresión que codifica una proteína de fusión que comprende:
(i) un dominio ZymoZipper (ZZ) en el que el dominio ZymoZipper (ZZ) comprende una secuencia de repetición dehéptada viral;
(ii) una proteína heteróloga; y
(iii) una secuencia de adaptador, en el que el adaptador comprende una secuencia seleccionada del grupoconstituido por la SEC ID Nº: 33, SEC ID Nº: 34, SEC ID Nº: 35, SEC ID Nº: 36, SEC ID Nº: 37 y SEC ID Nº: 38.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2006/035225.
Solicitante: ZYMOGENETICS, INC..
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 1201 EASTLAKE AVENUE EAST SEATTLE, WA 98102 ESTADOS UNIDOS DE AMERICA.
Inventor/es: FOX,BRIAN,A, MOORE,MARGARET,D.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
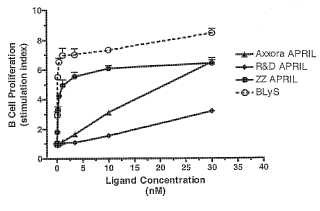
- C12N15/63 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Introducción de material genético extraño utilizando vectores; Vectores; Utilización de huéspedes para ello; Regulación de la expresión.
PDF original: ES-2415890_T3.pdf
Fragmento de la descripción:
Procedimiento de preparación de proteínas triméricas.
La presente memoria descriptiva describe polipéptidos capaces de formar multímeros, particularmente trímeros y la fabricación y uso de tales polipéptidos.
A. Bobinas en espiral
Un componente básico de la estructura cuaternaria de los presentes polipéptidos multimerizantes es la bobina en espiral (revisado en Müller y col., (2000) Meth. Enzymol. 328: 261-283) . Las bobinas en espiral son dominios de proteína que toman la forma de haces similares a sogas suavemente retorcidas. Los haces contienen dos a cinco hélices en orientación paralela o antiparalela. La característica esencial de las secuencias de bobina en espiral es un repetición de siete residuos, o héptada (abcdefg) n con la primera (a) y cuarta (d) posiciones comúnmente ocupadas por aminoácidos hidrófobos. Los restantes aminoácidos de la estructura de bobina en espiral son generalmente polares, excluyéndose normalmente la prolina debido a su efecto perturbador sobre la arquitectura helicoidal.
Esta repetición de héptada característica (también conocida como una repetición 3, 4 hidrófoba) es lo que forma la estructura del dominio de la bobina en espiral, barriendo cada residuo aproximadamente 100º. Esto hace que los siete residuos de la repetición de héptada no alcancen dos giros completos de 20º. El retraso forma un ligera tira hidrófoba hacia la izquierda de residuos que transcurren hacia abajo en una hélice y la bobina en espiral se forma cuando se asocian estas tiras hidrófobas. Las desviaciones de la separación 3, 4 regular de residuos apolares cambia el ángulo de la tira hidrófoba con respecto al eje de la hélice, alterando el ángulo de cruce de las hélices y desestabilizando la estructura cuaternaria. Un ejemplo de un medio común de representar en diagrama las repeticiones de héptada de bobina en espiral se ilustra en la Figura 1.
Los dímeros y trímeros paralelos son las estructuras de bobina en espiral observadas más comunes. Las características que distinguen dímeros de trímeros de formaciones de oligomerización de mayor orden son relativamente bien entendidas. Los residuos centrales de la repetición de héptada (residuos a y d) determinan en gran medida el estado de oligomerización, mientras que los residuos en el borde de la hélice (e y g) desempeñan funciones secundarias. Los trímeros son la organización por defecto para una distribución al azar de residuos centrales, ya que otros estados de oligomerización no pueden tolerar aminoácidos º-ramificados (Ile, Val y Thr) en la posición d para dímeros o la posición a para tetrámeros. A diferencia, los trímeros generalmente permiten la presencia de aminoácidos º-ramificados y otros hidrófobos en las posiciones centrales.
Se han usado fusiones de bobinas en espiral para lograr diversos fines experimentales. Un uso común es la sustitución de dominios de oligomerización naturales con una secuencia heteróloga para alterar el estado de oligomerización, estabilidad y/o avidez. Los monómeros de baja afinidad que no se asocian naturalmente pueden oligomerizarse con el fin de unirse efectivamente a otras dianas multiméricas. Adicionalmente, la fusión de dominios de oligomerización puede usarse para imitar el estado activado de la proteína nativa, que es difícil de lograr con la producción de proteínas recombinantes (véase, por ejemplo, Pullen y col. (1999) Biochem. 94:6032) . Este enfoque ha sido particularmente eficaz cuando solo se producen dominios específicos tales como la porción extracelular (citoplásmica) o intracelular de una proteína de interés. Comúnmente, las bobinas en espiral se fusionan genéticamente a la proteína de interés mediante un adaptador flexible que proporcionará acceso para la fusión a un gran espacio tridimensional. Se usan fusiones directas para fines experimentales que requieren más moléculas rígidas, tales como aquellas usadas para la cristalización.
Se han desarrollado varios sistemas modelo de bobina en espiral basándose en la información estructural de grandes proteínas estructural tales como miosina y tropomiosina (TM43, Lau y col. J Biol Chem; 259: 13253-13261) , un grupo de proteínas conocido como colectinas (Hoppe y col. (1994) Protein Sci; 3:1143-1158) , o de la región de dimerización de proteínas reguladoras de ADN tales como la proteína del activador transcripcional de levadura GCN4-p1 (Landschulz y col. (1988) Science; 240:1759-1764) . Esta última estructura se denomina frecuentemente una “cremallera de leucina” o LZ. Se han preparado sistemas modelo derivados de TM43, específicamente en los que una leucina por héptada se ha cambiado a fenilalanina. Esta estructura se conoce como una “cremallera de fenilalanina” o FZ (Thomas y col. Prog Colloid Polymer Sci; 99: 24-30) . Un tercer tipo de derivado muy conocido de LZ es la cremallera de isoleucina (IZ) (Harbur y y col. (1994) Nature 371:80-83) .
Un concepto importante de bobinas en espiral modelo es la capacidad para producirse en el huésped de expresión. La falta de enlaces disulfuro en las estructuras de bobina en espiral ayuda en su producción en sistemas de expresión heterólogos. Sin embargo, las secuencias diseñadas de novo tienden a ser sensibles a la proteólisis. Aunque se expresan eficazmente, la falta relativa de eficacia con respecto a secuencias naturales refleja las lagunas en el presente conocimiento sobre todas las variables que participan en la interacción de proteínas (Arndt y col. (2002) Structure 10: 1235-1248) . Adicionalmente, el uso de secuencias modelo es problemático cuando el fin de la proteína de fusión producida es una proteína biológicamente funcional.
B. Repeticiones de héptada virales Muchos virus producen una forma fusogénica de glicoproteínas de la envoltura viral. Entre los géneros o familias virales que presentan este tipo de proteínas de fusión están Orthomyxovirus (por ejemplo, virus de la gripe) , Filovirus (virus delÉbola) , Betaretrovirus (virus Mason-Pfizer del mono (VMMP) ) , Gammaretrovirus (virus de la leucemia murina de Friend (FRMLV) ; virus de la leucemia murina de Moloney (MoMLV) ) , Deltaretrovirus (virus de la leucemia de linfocitos T humana tipo 1 (HTLV-1) ) y Lentivirus (virus de la inmunodeficiencia humana tipo 1 (VIH-1) y de la inmunodeficiencia simia (VIS) ) (revisado en Cheynet y col. (2005) J Virol; 79; 5585-5593) . En estos virus, la subunidad de transmembrana se produce como una proteína de fusión, codificada por el gen env. Este producto génico se escinde en dos proteínas, la proteína de la superficie que participa en el reconocimiento de receptores y la subunidad de transmembrana que ancla el complejo env entero a la membrana y participa en la entrada del virus por la fusión de membranas. Estas proteínas se caracterizan por la presencia de repeticiones de héptada dentro de la región TM que forman fuertes interacciones entre oligómeros de la proteína mediante la formación de una estructura de bobina en espiral con tres subunidades (Li y col. (1996) J Virol; 70: 1266-1270; Tucker y col. (1991) Virol; 185:710-720) .
Se han determinado estructuras cristalinas de varias proteínas TM: MoMLV (Fass y col. (1996) Nat Struc Biol; 3:465-469) ; VIH-1 (Chan y col. (1997) Cell; 89: 263-273) ; HTLV-1 (Kobe y col. (1999) PNAS; 93:4319-4324) . También se encontraron estructuras similares en el virus de la gripe (Wilson y col. (1981) Nature; 289: 366-373) y el virus del Ébola (Malashkevich y col. (1999) PNAS; 96:2662-2667) y se ha supuesto que esta estructura refleja un mecanismo común para el proceso de fusión y entrada viral (Chambers y col. J Gen Virol; 71: 3075-3080) . Otros virus que incluyen repeticiones de héptada dentro de su genoma son Cornavirus (cornavirus asociado al síndrome respiratorio agudo grave (SARS-CoV) ) (Bosch y col. J Virol 77: 8801-8811) ; virus del herpes (virus del herpes simple 1 (VHS-1) (Gianni y col. (2005) J Virol 79:7042-7049) y citomegalovirus humano (CMV) ) (Lopper y col. J Virol (2004) 78: 8333-8341) ; y Paramyxovirus (virus del sarampión) (Buckland y col. (1992) J Gen Virol 73:1703-1707) .
La homología de secuencias en la región TM también ha permitido la identificación de retrovirus endógenos en bases de datos de secuencias. Se han realizado estas búsquedas y han identificado satisfactoriamente retrovirus endógenos en muchos genomas de organismos, que incluyen ser humano, rata y ratón. Un ejemplo muy conocido de una familia de retrovirus endógenos humanos (RVEH) es RVEH-W. Se ha mostrado que un sitio de esta familia, ERVWE1, codifica un marco de lectura abierto de env de longitud completa y produce una proteína también conocida como sincitina (Cheynet y col. (2005) J Virol; 79:5585-5593) . Al igual que la proteína viral,... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento de preparación de una proteína trimérica que comprende cultivar una célula huésped transformada o transfectada con un vector de expresión que codifica una proteína de fusión que comprende:
(i) un dominio ZymoZipper (ZZ) en el que el dominio ZymoZipper (ZZ) comprende una secuencia de repetición de héptada viral;
(ii) una proteína heteróloga; y
(iii) una secuencia de adaptador, en el que el adaptador comprende una secuencia seleccionada del grupo constituido por la SEC ID Nº: 33, SEC ID Nº: 34, SEC ID Nº: 35, SEC ID Nº: 36, SEC ID Nº: 37 y SEC ID Nº: 38.
2. El procedimiento de la reivindicación 1, en el que la proteína heteróloga comprende el dominio extracelular de dicha proteína.
3. El procedimiento de la reivindicación 1, en el que dicha proteína de fusión es soluble.
4. El procedimiento de la reivindicación 1, en el que el dominio ZZ se deriva de la subunidad transmembrana (TM) de un virus clasificado dentro del género o de la familia seleccionados del grupo constituido por Orthomyxovirus, Filovirus, Betaretrovirus, Gammaretrovirus, Deltaretrovirus, Lentivirus, Cornavirus y Paramyxovirus, preferentemente en el que el virus es gripe A o un Betaretrovirus tal como el virus Mason-Pfizer del mono (VMMP) .
5. El procedimiento de cualquier reivindicación anterior, en el que el dominio ZZ comprende una secuencia seleccionada del grupo constituido por la SEC ID Nº: 27, SEC ID Nº: 28, SEC ID Nº: 29, SEC ID Nº: 30, SEC ID Nº: 31 y SEC ID Nº: 32.
6. El procedimiento de cualquier reivindicación anterior, en el que el adaptador comprende la SEC ID Nº: 35 y el dominio ZZ comprende SEC ID Nº: 32.
7. El procedimiento de la reivindicación 1, en el que dicho dominio ZZ se deriva de la subunidad transmembrana (TM) de un retrovirus endógeno humano (RVEH) , preferentemente en el que dicho RVEH es ERVWE1.
8. El procedimiento de cualquier reivindicación anterior que comprende además eliminar el dominio ZZ de la proteína trimérica usando digestión con proteasa, preferentemente en el que dicha proteasa es tripsina.
9. Un procedimiento de preparación de una proteína hetero-trimérica soluble:
cultivando una primera célula huésped transformada o transfectada con un primer vector de expresión que codifica una primera proteína de fusión que comprende un primer dominio ZZ y una primera proteína heteróloga,
cultivando una segunda célula huésped transformada o transfectada con un segundo vector de expresión que codifica una segunda proteína de fusión que comprende un segundo dominio ZZ y una segunda proteína heteróloga, y
cultivando una tercera célula huésped transformada o transfectada con un tercer vector de expresión que codifica una tercera proteína de fusión que comprende un tercer dominio ZZ y una tercera proteína heteróloga;
en el que dicha primera, segunda y tercera proteínas de fusión comprenden además una secuencia de adaptador, en el que el adaptador comprende una secuencia seleccionada del grupo constituido por la SEC ID Nº: 33, SEC ID Nº: 34, SEC ID Nº: 35, SEC ID Nº: 36, SEC ID Nº: 37 y SEC ID Nº: 38;
en el que el primer, segundo y tercer dominios ZZ comprenden una secuencia de repetición de héptada viral y en el que el primer, segundo y tercer dominios ZZ forman preferencialmente un hetero-trímero;
y combinando la primera, segunda y tercera proteínas de fusión en condiciones que promuevan un hetero-trímero.
10. El procedimiento de la reivindicación 9, en el que dicha primera y segunda células huésped son la misma célula.
11. El procedimiento de la reivindicación 9, en el que dicha primera y segunda proteínas heterólogas son las mismas.
12. El procedimiento de la reivindicación 11, en el que dicha primera y segunda proteínas heterólogas son el dominio extracelular de BLyS y dicha tercera proteína heteróloga es el dominio extracelular de APRIL.
13. El procedimiento de la reivindicación 9 que comprende además eliminar el dominio ZZ de la proteína hetero-trimérica usando digestión con proteasa, preferentemente en el que dicha proteasa es tripsina.
Patentes similares o relacionadas:
 Métodos para producir proteínas bicatenarias en bacterias, del 29 de Julio de 2020, de GENENTECH, INC.: Un método para producir un receptor de linfocitos T monoclonal de movilización inmunitaria contra el cáncer (ImmTAC) que comprende una cadena alfa del receptor de linfocitos […]
Métodos para producir proteínas bicatenarias en bacterias, del 29 de Julio de 2020, de GENENTECH, INC.: Un método para producir un receptor de linfocitos T monoclonal de movilización inmunitaria contra el cáncer (ImmTAC) que comprende una cadena alfa del receptor de linfocitos […]
Anticuerpo biespecífico o mezcla de anticuerpos con cadenas ligeras comunes, del 15 de Julio de 2020, de Jiangsu Alphamab Biopharmaceuticals Co., Ltd: Anticuerpo biespecífico o parte de unión a antígeno del mismo, en el que el anticuerpo biespecífico o la parte de unión a antígeno del mismo tiene una cadena […]
Vacunas y diagnóstico de torque teno virus porcino, del 18 de Junio de 2020, de VIRGINIA TECH INTELLECTUAL PROPERTIES, INC.: Composición inmunogénica que comprende una proteína según SEQ ID NO. 16.
Producción de FDCA catalizada por deshidrogenasa, del 17 de Junio de 2020, de PURAC BIOCHEM B.V.: Proceso para oxidar ácido 5-hidroximetil-2-furancarboxílico (HMFCA) a ácido 5-formil-2-furoico (FFA), donde el proceso comprende la etapa de incubar una […]
Vectores de AAV dirigidos a oligodendrocitos, del 10 de Junio de 2020, de THE UNIVERSITY OF NORTH CAROLINA AT CHAPEL HILL: Un ácido nucleico que codifica una cápside de AAV, comprendiendo el ácido nucleico una secuencia codificante de la cápside de AAV que es al menos el 96 % idéntica […]
Ácido nucleico que comprende o codifica para un tallo-bucle de histona y una secuencia poli(A) o una señal de poliadenilación para aumentar la expresión de un antígeno patogénico codificado, del 3 de Junio de 2020, de CureVac AG: Secuencia de ácido nucleico que comprende o codifica en la dirección 5' → 3' para: i) • una región codificante, que codifica para al menos […]
Microorganismo con productividad de l-lisina aumentada y procedimiento para producir l-lisina utilizando el mismo, del 27 de Mayo de 2020, de CJ CHEILJEDANG CORPORATION: Una subunidad beta prima (subunidad-β') mutante de la ARN polimerasa, en la que la subunidad beta prima (subunidad-β') mutante de la ARN polimerasa tiene […]
Agrupación de genes de biosíntesis de carrimicina, del 27 de Mayo de 2020, de Shenyang Fuyang Pharmaceutical Technology Co., Ltd: Agrupación de genes de biosíntesis de carrimicina, que consiste en 44 genes que comprende: 1) cinco genes de policétido sintasa, incluyendo los residuos […]