Polimerización activada por pirofosforólisis relativa a un extremo en 2''.
Método para retirar un nucleótido extremo en 2' de un oligonucleótido que comprende:
incubar al menos un ácido nucleico diana con:
pirofosfato al menos un primer biocatalizador que comprende una actividad de pirofosforólisis, y al menos un oligonucleótido que comprende un nucleótido extremo en 2', siendo dicho oligonucleótido al menos parcialmente complementario a al menos una primera subsecuencia del ácido nucleico diana, bajo condiciones en las que el primer biocatalizador retira al menos el nucleótido extremo en 2' del oligonucleótido produciendo un nucleótido extremo en 2' retirado y un oligonucleótido acortado, retirando de este modo el nucleótido del oligonucleótido
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2007/008997.
Solicitante: F. HOFFMANN-LA ROCHE AG.
Nacionalidad solicitante: Suiza.
Dirección: GRENZACHERSTRASSE 124 4070 BASEL SUIZA.
Inventor/es: GELFAND, DAVID HARROW, Gupta,Amar P, BAUER,Keith A, BODEPUDI,Veeraiah, NIEMIEC,John.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2381505_T3.pdf
Fragmento de la descripción:
Polimerización activada por pirofosforólisis relativa a un extremo en 2'
SECTOR TÉCNICO DE LA INVENCIÓN
La presente invención se refiere de forma general a la biología molecular y la química de ácidos nucleicos. De forma más específica, la presente invención se refiere a la polimerización y amplificación de ácidos nucleicos en las que intervienen extremos en 2'.
ANTECEDENTES DE LA INVENCIÓN
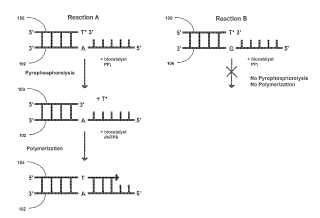
La polimerización activada por pirofosforólisis (PAP) es un proceso en el que interviene la activación mediada por pirofosforólisis de cebadores activables por pirofosforólisis hibridados con ácidos nucleico diana o molde seguida de la extensión de los cebadores activados. Los cebadores activables por pirofosforólisis incluyen típicamente nucleótidos extremos, tales como los dideoxinucleótidos (ddNMPs) en su extremo 3'. Estos nucleótidos extremos deben, de forma general, retirarse enzimáticamente de dichos cebadores hibridados y bloqueados mediante reacciones de pirofosforólisis que consumen pirofosfato produciendo los cebadores activados para que pueda proceder la extensión de los cebadores. Dado que la activación de los cebadores requiere típicamente de cebadores y ácido nucleicos diana que se correspondan perfectamente, estas reacciones generan generalmente una cantidad limitada, si es que generan alguna, de productos no específicos, por ejemplo, debido a la formación de dímeros de cebador u otros artefactos del cebado. A modo de ejemplo entre las aplicaciones de la PAP se incluyen, entre otras, la provisión de métodos alternativos para la amplificación de ácidos nucleicos (por ejemplo como parte de procesos de detección de alelos raros, ensayos de detección de mutaciones somáticas, etc.) .
Tal como se ha mencionado más arriba, algunas estrategias relacionadas con la PAP utilizan cebadores terminados con ddNMP y ADN polimerasas termoestables capaces de incorporar ddNMP (por ejemplo, enzimas mutados que contienen una mutación "F a Y" en la "Hélice O") para llevar a cabo la pirofosforólisis de los cebadores con un grupo ddNMP 3-terminal en presencia de pirofosfato (PPi) tras la primera unión/hibridación al molde. Estas estrategias presentan en general diversas desventajas. Por ejemplo, estas técnicas utilizan típicamente concentraciones limitantes muy bajas de dNTPs, lo cual reduce (enlentece) la velocidad de extensión (número de nucleótidos polimerizados por segundo) . Más específicamente, cuando en estas reacciones se libera un ddNMP en posición terminal 3' en forma de ddNTP, la concentración muy baja de dNTPs (es decir "el tamaño del pool") aumenta la posibilidad de reincorporar la concentración siempre en aumento de ddNTPs (resultante de los ciclos sucesivos de PAP) antes de la extensión completa de los cebadores (necesaria para una PCR eficiente) . Esta reincorporación es en general no deseable, requiriéndose PAP adicionales para retirar los ddNMP incorporados y además contribuye a la extensión incompleta de los cebadores y compromete la eficiencia de los productos de extensión completa de los cebadores de cada ciclo. Además, varias de estas estrategias previas PAP son capaces de generar solamente amplicones muy cortos, por ejemplo en algunos casi incluso más cortos que los dímeros de cebadores. Por lo tanto, la eficiencia de los ciclos de PCR de estas estrategias anteriores está generalmente alterada, requiriéndose típicamente muchos ciclos para detectar dianas poco abundantes. Zhang y otros (2005) , Trends in Biotechnology vol. 23/nº 2: 92-96, describen, por ejemplo, la utilización de la actividad exonucleasa 3' de las polimerasas para retirar los nucleótidos discrepantes del extremo 3' de un cebador. La actividad de corrección de errores de la actividad exonucleasa 3' de las polimerasas no retira nucleótidos extremos en 2' de los oligonucleótidos.
De lo anteriormente expuesto, es evidente que son deseables métodos adicionales en relación con la PAP. La presente invención da a conocer métodos en relación con la PAP que utilizan nucleótidos extremos en 2', así como una serie de características adicionales que serán evidentes tras la revisión completa de la siguiente descripción.
RESUMEN DE LA INVENCIÓN
La presente invención se refiere a la polimerización y amplificación de ácidos nucleicos. La presente invención en concreto da a conocer métodos relacionados con la PAP que implican el acoplamiento seriado de la pirofosforólisis y la polimerización. Estos métodos pueden utilizarse, por ejemplo, para el análisis de SNP y la detección de mutaciones somáticas raras, entre otras aplicaciones.
A modo de ejemplo, los métodos y otros aspectos descritos en la presente invención incrementan la especificidad general de las reacciones de síntesis mediadas por oligonucleótidos. Por ejemplo, de forma análoga a otros métodos "de inicio en caliente" (por ejemplo, "inicio en caliente" mediada por anticuerpos, aptámeros o enzimas modificados químicamente de forma reversible) , se reduce o elimina la "extensión del ciclo número cero" (pre-PCR) . Contrariamente a estos otros métodos, la activación de los cebadores se realiza en todas y cada una de las nuevas etapas de síntesis mediada por oligonucleótidos. Ello mejora la especificidad global de la reacción, minimizando la generación de productos secundarios no deseados. Por consiguiente, se mejora la detección de secuencias con un bajo número de copias o incluso con una sola copia. Además, también se mejora el rendimiento en reacciones de amplificación múltiplex (en el que se amplifican algunas o muchas dianas diferentes) al disminuir o eliminar la generación de productos de síntesis no específicos y no deseados, por ejemplo dímeros de cebadores en el caso de la PCR.
La invención en concreto da a conocer un método para retirar un nucleótido extremo en 2' de un oligonucleótido. El método incluye la incubación de al menos un ácido nucleico diana con; pirofosfato en al menos un primer biocatalizador que contiene actividad pirofosforólisis y al menos un oligonucleótido (por ejemplo un ácido nucleico cebador, un ácido nucleico sonda, etc.) que comprende un nucleótido extremo en 2' (por ejemplo en el extremo 3') , 5 siendo dicho oligonucleótido al menos parcialmente complementario a al menos una primera subsecuencia del ácido nucleico diana, bajo condiciones en las que el primer biocatalizador retira al menos el nucleótido extremo en 2' del oligonucleótido produciendo un nucleótido con el extremo en 2' retirado y un oligonucleótido acortado, retirando de este modo el nucleótido del oligonucleótido. En algunas realizaciones, el método incluye la incubación del ácido nucleico diana con el primer biocatalizador, el oligonucleótido y pirofosfato, añadiéndose el pirofosfato para retirar el nucleótido extremo en 2'.
Los nucleótidos extremos en 2' utilizados en los oligonucleótidos descritos en la presente invención incluyen diversas realizaciones. En algunas realizaciones, por ejemplo, el nucleótido extremo en 2' comprende un nucleótido 2'-monofosfato-3'-hidroxilo. Para ejemplificarlo más, en algunas realizaciones, el nucleótido extremo en 2' comprende la fórmula:
en la que R1 es OH; B es al menos un anillo homocíclico, al menos un anillo heterocíclico, al menos un grupo arilo o combinaciones de los mismos; BG es un grupo bloqueador seleccionado del grupo formado por: CN, NO2, un grupo fosfato, un grupo aldehído o combinaciones de los mismos; Z es O ó CH2; y ----- representa un enlace sencillo o doble. Además, el nucleótido extremo en 2' es típicamente no extensible gracias a uno o más nucleótidos que 20 incorporan biocatalizadores seleccionados del grupo formado por, por ejemplo, una ADN polimerasa G46E E678G CS5, una ADN polimerasa G46E L329A E678G CS5, una ADN polimerasa G46E L329A D640G S671F CS5, una ADN polimerasa G46E L329A D640G S671 F E678 CS5, una ADN polimerasa G46E E678G CS6, una polimerasa'ZO5R, ADN Taq E615G, una polimerasa de Thermus flavus, una polimerasa TMA-25, una polimerasa E678G TMA-25, una polimerasa TMA-30, una polimerasa E678G TMA-30, una ADN polimerasa Tth, una polimerasa 25 de la especie Thermus, una polimerasa Taq E615G, una polimerasa Thermus ZO5R, una ADN polimerasa T7, una ADN polimerasa I Kornberg, una ADN polimerasa Klenow, una ADN polimerasa Taq, una ADN polimerasa microcócica, una ADN polimerasa alfa, una transcriptasa inversa, una transcriptasa inversa AMV, una transcriptasa inversa M-MuLV, una ADN polimerasa, una ARN polimerasa, una ARN polimerasa de E. coli, una ARN polimerasa SP6, una ARN polimerasa T3, una ADN polimerasa T4, una ARN polimerasa T7, una ARN polimerasa II, una transferasa terminal,... [Seguir leyendo]
Reivindicaciones:
1. Método para retirar un nucleótido extremo en 2' de un oligonucleótido que comprende:
incubar al menos un ácido nucleico diana con:
pirofosfato al menos un primer biocatalizador que comprende una actividad de pirofosforólisis, y al menos un oligonucleótido que comprende un nucleótido extremo en 2', siendo dicho oligonucleótido al menos parcialmente complementario a al menos una primera subsecuencia del ácido nucleico diana, bajo condiciones en las que el primer biocatalizador retira al menos el nucleótido extremo en 2' del oligonucleótido produciendo un nucleótido extremo en 2' retirado y un oligonucleótido acortado, retirando de este modo el nucleótido del oligonucleótido 2. Método, según la reivindicación 1, en el que el oligonucleótido comprende el nucleótido extremo en 2' en el extremo 3' del oligonucleótido.
3. Método, según la reivindicación 1, en el que el nucleótido extremo en 2' es no extensible por uno o más biocatalizadores que incorporan de nucleótidos del grupo formado por: una ADN polimerasa G46E E678G 15 CS5, una ADN polimerasa G46E L329A E678G CS5, una ADN polimerasa G46E L329A D640G S671F CS5, una ADN polimerasa G46E L329A D640G S671 F E678G CS5, una ADN polimerasa G46E E678G CS6, una polimerasa 'ZO5R, una ADN polimerasa E615G Taq, una polimerasa Thermus flavus, una polimerasa TMA-25, una polimerasa E678G TMA-25, una polimerasa TMA-30, una polimerasa E678G TMA-30, una ADN polimerasa Tth, una polimerasa de la especie Thermus species SPS-17, una polimerasa 20 E615G Taq, una polimerasa Thermus ZO5R, una ADN polimerasa T7, una ADN polimerasa Kornberg I, una ADN polimerasa Klenow, una ADN polimerasa Taq, una ADN polimerasa microcócica, una ADN polimerasa alfa, una transcriptasa inversa, una transcriptasa inversa AMV, una transcriptasa inversa M-MuLV, una ADN polimerasa, una ARN polimerasa, una ARN polimerasa de E. coli, una ARN polimerasa SP6, una ARN polimerasa T3, una ADN polimerasa T4, una ARN polimerasa T7, una ARN polimerasa II, una transferasa terminal, una polinucleótido fosforilasa, una ADN polimerasa incorporadora de análogos de nucleótidos y una ADN polimerasa incorporadora de nucleótidos.
4. Método, según la reivindicación 1, en el que el primer biocatalizador comprende una actividad incorporadora de nucleótidos extremos en 2', y en el que el método comprende incubar el ácido nucleico diana con el primer biocatalizador, el oligonucleótido acortado y al menos un nucleótido adicional bajo condiciones en las que el primer biocatalizador incorpora el nucleótido adicional en el extremo del oligonucleótido acortado dando lugar a un oligonucleótido extendido; o en el que el método comprende incubar el ácido nucleico diana con al menos un segundo biocatalizador que comprende una actividad incorporadora de nucleótidos, el oligonucleótido acortado, y al menos un nucleótido adicional bajo condiciones en las que el segundo biocatalizador incorpora el nucleótido adicional en el extremo del oligonucleótido acortado dando lugar a un oligonucleótido extendido.
5. Método, según la reivindicación 4, en el que el primer y/o el segundo biocatalizador comprende un enzima seleccionado del grupo formado por; una polimerasa y una transcriptasa inversa.
6. Método, según la reivindicación 1, en el que el nucleótido extremo en 2' comprende la fórmula: 40 en la que Z es O o CH2; B es al menos un anillo homocíclico, al menos un anillo heterocíclico, al menos un grupo arilo o combinaciones de los mismos; BG es un grupo bloqueador; 45 R1 es OH;
y, - - - - - representa un enlace sencillo o doble; y en el que BG se selecciona del grupo formado por: CN, NO2, un grupo fosfato, un grupo aldehído y combinaciones de los mismos.
7. Método, según la reivindicación 6, en el que el BG comprende un grupo fosfato que comprende la fórmula:
o, en las que Q es O, S o NH; X es H, OH, CH3, BH3, F, o SeH; y Z es O, S o Se; o en la que Q es O, S o NH;
X es H, OH o NH;
Z es O, S o Se; y R es un grupo alquilo, un grupo alquenilo o un grupo alquinilo; o 20
en la que Q es O, S o NH; X es H, OH o NH; Z es O, S o Se; L es -CONH (CH2) nNH-, -CO (CH2) nNH-, o -CONH (CH2CH2O) nCH2CH2NH-; n es un número entero mayor a 0; y, R es NH2, SH, COOH, una fracción silenciadora, una fracción reportera, biotina o una fracción de afinidad.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]