Plantas de algodón resistentes a los insectos y métodos para identificación de las mismas.
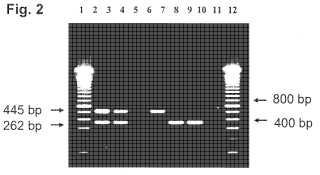
Una planta transgénicas de algodón resistente a los insectos, o células, partes, semilla o progenie de lamisma, cada una de las cuales comprende un evento de élite, comprendiendo dicho evento de élite un DNA extrañoque comprende la secuencia codificante de un gen cry1ab modificado bajo el control de un promotor S7 del virus dela atrofia subterránea del trébol, en donde dicho evento de élite puede obtenerse a partir de la semilla de referenciaque comprende dicho evento que ha sido depositada en la ATCC bajo el número de depósito PTA-8171, el DNAgenómico de dicha planta de algodón, o células, partes, semilla, o progenie de la misma cuando se analiza utilizandoel protocolo de identificación de eventos élite para dicho evento de élite con dos cebadores que comprenden lasecuencia de nucleótidos de SEQ ID No.
10 y SEQ ID No. 11 respectivamente, produce un fragmento de DNA de262 pb o cuando se analiza utilizando el protocolo de identificación de eventos élite para dicho evento de élite condos cebadores que comprenden la secuencia de nucleótidos de SEQ ID No. 3 y SEQ ID No. 6 respectivamente,produce un fragmento de DNA de 369 pb.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2008/002667.
Solicitante: Bayer CropScience NV .
Nacionalidad solicitante: Bélgica.
Dirección: J.E. Mommaertslaan 14 1831 Diegem BELGICA.
Inventor/es: TROLINDER,Linda, MOENS,SOFIE, PAELINCK,DIMITRI, HABEX,VEERLE, VAN HERCK,HANS.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- A01H5/00 NECESIDADES CORRIENTES DE LA VIDA. › A01 AGRICULTURA; SILVICULTURA; CRIA; CAZA; CAPTURA; PESCA. › A01H NOVEDADES VEGETALES O PROCEDIMIENTOS PARA SU OBTENCION; REPRODUCCION DE PLANTAS POR TECNICAS DE CULTIVO DE TEJIDOS. › Angiospermas,es decir, plantas con flores, caracterizadas por sus partes vegetales; Angiospermas caracterizadas de forma distinta que por su taxonomía botánica.
- C12N15/82 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › para células vegetales.
PDF original: ES-2432406_T3.pdf
Fragmento de la descripción:
Plantas de algodón resistentes a los insectos y métodos para identificación de las mismas.
Campo de la Invención Esta invención se refiere a plantas transgénicas de algodón, material y semillas de plantas, caracterizadas por
albergar un evento de transformación específico, particularmente por la presencia de un gen que codifica una proteína que confiere resistencia a los insectos, en una localización específica en el genoma del algodón. Las plantas de algodón de la invención combinan el fenotipo de resistencia a los insectos con una eficiencia agronómica, estabilidad genética y adaptabilidad a diferentes sustratos genéticos equivalentes a la línea de algodón no transformada en ausencia de insectos. Esta invención proporciona adicionalmente métodos y kits para identificar la presencia de material de plantas que comprende específicamente el evento de transformación EE-GH5 en muestras biológicas.
Antecedentes de la Invención La expresión fenotípica de un transgén en una planta está determinada a la vez por la estructura del gen propiamente dicha y por su localización en el genoma de planta. Al mismo tiempo, la presencia del transgén (en un 15 DNA extraño) en diferentes localizaciones en el genoma influirá en el fenotipo global de planta de diferentes maneras. La introducción con éxito agronómica o industrialmente de un rasgo comercialmente interesante en una planta por manipulación genética puede ser un procedimiento laborioso que depende de diferentes factores. La transformación y regeneración real de plantas transformadas genéticamente son sólo el primero en una serie de pasos de selección, que incluyen caracterización genética extensa, mejora genética, y evaluación en pruebas de campo, conduciendo finalmente a la selección de un evento de élite.
La fibra de algodón es el artículo textil individual más importante mundialmente. Aproximadamente 80 millones de acres (32 millones de hectáreas) de algodón se recolectan anualmente a lo largo del globo terráqueo. El algodón es la quinta cosecha más importante en los Estados Unidos en términos de producción en hectáreas, con más de 15 millones de acres (6 millones de hectáreas) plantadas en 2000.
Las especies de insectos más dañinas que se alimentan de algodón son Helicoverpa zea (oruga de las mazorcas del maíz u oruga de las cápsulas del algodón) , Helicoverpa armigera (oruga de las cápsulas del algodón americano) , Heliothis virescens (oruga de los brotes del tabaco) y Helicoverpa punctigera.
La identificación inequívoca de un evento de élite está adquiriendo importancia creciente teniendo en cuenta las discusiones en Novel Food/Feed, segregación de productos GMO y no-GMO y la identificación de material
patentado. Idealmente, dicho método de identificación es a la vez rápido y sencillo, sin necesidad de un montaje extenso de laboratorio. Adicionalmente, el método debería proporcionar resultados que permitan la determinación inequívoca del evento de élite sin interpretación de expertos, pero que se cumplan bajo escrutinio de expertos en caso necesario. Instrumentos específicos para uso en la identificación del evento de élite EE-GH5 en muestras biológicas se describen en esta memoria.
EE-GH5 ha sido identificado como un evento de élite de una población de plantas transgénicas de algodón en el desarrollo de algodón resistente a los insectos (Gossypium hirsutum) que comprende un gen que codifica una proteína cristalina insecticida de Bacillus thuringiensis. Las plantas transgénicas de algodón contienen un gen quimérico que codifica una proteína cristalina insecticida Bt (como se describe en WO 03/093484) bajo control de un promotor expresable en plantas.
Plantas de algodón que comprenden un gen de resistencia a los insectos han sido descritas en la técnica. Sin embargo, ninguna de las descripciones de la técnica anterior propone o sugiere la presente invención.
Sumario de la Invención La presente invención se refiere a una planta transgénicas de algodón que comprende, integrada de manera estable en su genoma, una casete de expresión que comprende un gen de resistencia a los insectos que comprende la 45 secuencia codificante del gen cr y 1Ab (como se describe en el Ejemplo 1.1 en esta memoria) , que es resistente a los insectos y, en ausencia de presión de insectos, tiene una eficiencia agronómica que es sustancialmente equivalente a la línea isógena no transgénica como se caracteriza en las reivindicaciones. Bajo presión de insectos, la planta tendrá un fenotipo agronómico superior comparada con la planta no transgénica.
De acuerdo con la presente invención, la planta de algodón comprende el evento de élite EE-GH5.
Más específicamente, la presente invención se refiere a una planta transgénica de algodón, cuyo DNA genómico se caracteriza por el hecho de que, cuando se utiliza en un protocolo de identificación PCR como se describe en esta memoria, utilizando dos cebadores dirigidos a la región flanqueante 5' o 3' de EE-GH5 y el DNA extraño, respectivamente, produce un fragmento que es específico para EE-GH5. Los cebadores pueden estar dirigidos contra la región flanqueante 5' dentro de SEQ ID No. 1 y el DNA extraño respectivamente, de tal modo que los cebadores comprenden o están constituidos por la secuencia de nucleótidos de SEQ ID No. 7 y SEQ ID No. 8
respectivamente, y producen un fragmento de DNA que tiene entre 100 y 700 pb, con preferencia aproximadamente 129 pb. Los cebadores pueden estar dirigidos también contra la región flanqueante 3' dentro de SEQ ID No. 2 y el DNA extraño respectivamente, de tal modo que los cebadores comprenden o están constituidos por la secuencia de nucleótidos de SEQ ID No. 3 y SEQ ID No. 6 respectivamente, y producen un fragmento de DNA que contiene entre 300 y 700 pb, con preferencia aproximadamente 369 pb.
La semilla de referencia que comprende el evento de élite de la invención ha sido depositada en la ATCC bajo el número de acceso PTA-8171, Una realización de la invención es la semilla que comprende el evento de élite EE-GH5 depositada con el número de acceso ATTC PTA-8171, que se desarrollarán en una planta de algodón resistente a los insectos, particularmente Helicoverpa sp. o Heliothis sp. La semilla del depósito ATCC número PTA8171, es un lote de siembra constituido por al menos aproximadamente 95% de almendras transgénicas homocigóticas para el transgén, que comprenden el evento de élite de la invención, que se desarrollarán en plantas resistentes a los insectos, siendo las plantas también plantas tolerantes a glufosinato. La semilla puede sembrarse y las plantas en crecimiento pueden tratarse con glufosinato como se describe en esta memoria para obtener 100% de plantas tolerantes a glufosinato, que comprenden el evento de élite de la invención. La invención se refiere adicionalmente a células, tejidos, progenie, y descendientes procedentes de una planta que comprende el evento de élite de la invención desarrollada a partir de la semilla depositada en la ATCC que tiene el número de acceso PTA8171. Se describen también en esta memoria plantas que pueden obtenerse por propagación de y/o mejora genética de una planta de algodón que comprende el evento de élite de la invención desarrollado a partir de la semilla depositada en la ATCC que tiene el número de acceso PTA-8171. La invención se refiere también a plantas de algodón que comprenden el evento de élite EE-GH5.
La invención se refiere adicionalmente a un método para identificación de una planta transgénica, o células o tejidos de la misma, que comprenden el evento de élite EE-GH5, método que está basado en la identificación de la presencia de secuencias caracterizadoras de DNA o aminoácidos codificados por dichas secuencias de DNA en la planta transgénica, células o tejidos como se caracterizan en las reivindicaciones. De acuerdo con una realización preferida de la invención, tales secuencias caracterizadoras de DNA son secuencias de 15 pares de bases (pb) ,
preferiblemente 20 pb, muy preferiblemente 30 pb o más que comprenden el sitio de inserción del evento, es decir a la vez una parte de DNA extraño y una parte del genoma de algodón (sea en la región flanqueante 5' o 3') contigua al mismo, que permiten la identificación específica del evento de élite.
La presente invención se refiere adicionalmente a métodos para identificar el evento de élite EE-GH5 en muestras biológicas, métodos que están basados en cebadores que reconocen específicamente la secuencias flanqueantes 5'
y/o 3' de EE-GH5.
Más específicamente, la invención se refiere a un método que comprende amplificar una secuencia de un ácido nucleico... [Seguir leyendo]
Reivindicaciones:
1. Una planta transgénicas de algodón resistente a los insectos, o células, partes, semilla o progenie de la misma, cada una de las cuales comprende un evento de élite, comprendiendo dicho evento de élite un DNA extraño que comprende la secuencia codificante de un gen cr y 1ab modificado bajo el control de un promotor S7 del virus de 5 la atrofia subterránea del trébol, en donde dicho evento de élite puede obtenerse a partir de la semilla de referencia que comprende dicho evento que ha sido depositada en la ATCC bajo el número de depósito PTA-8171, el DNA genómico de dicha planta de algodón, o células, partes, semilla, o progenie de la misma cuando se analiza utilizando el protocolo de identificación de eventos élite para dicho evento de élite con dos cebadores que comprenden la secuencia de nucleótidos de SEQ ID No. 10 y SEQ ID No. 11 respectivamente, produce un fragmento de DNA de 262 pb o cuando se analiza utilizando el protocolo de identificación de eventos élite para dicho evento de élite con dos cebadores que comprenden la secuencia de nucleótidos de SEQ ID No. 3 y SEQ ID No. 6 respectivamente, produce un fragmento de DNA de 369 pb.
2. Un método para identificación de una planta transgénicas de algodón resistente a los insectos, o células, partes, semilla o progenie de la misma especificada en la reivindicación 1 en muestras biológicas, comprendiendo 15 dicho método amplificar un fragmento de DNA de entre 100 y 500 pb de un ácido nucleico presente en dichas muestras biológicas utilizando una reacción en cadena de polimerasa con al menos dos cebadores, reconociendo uno de dichos cebadores la secuencia de nucleótidos de SEQ ID No. 1 desde el nucleótido 1 al nucleótido 98 o la secuencia de nucleótidos de SEQ ID No. 16 desde el nucleótido 1 al nucleótido 563 o la secuencia de nucleótidos del complemento de SEQ ID No. 2 desde el nucleótido 41 al nucleótido 452, reconociendo el otro cebador de dichos cebadores la secuencia de nucleótidos del complemento de SEQ ID No. 1 desde el nucleótido 99 al nucleótido 334 o la secuencia de nucleótidos de SEQ ID No. 2 desde el nucleótido 1 al nucleótido 40.
3. El método de la reivindicación 2, en donde un cebador está constituido por una secuencia de nucleótidos de 17 a 200 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos de SEQ ID No. 1 desde el nucleótido 1 al nucleótido 98 o la secuencia de nucleótidos de SEQ ID No. 16 desde el nucleótido 1 al nucleótido 563
o dicho cebador está constituido por una secuencia de nucleótidos de 17 a 200 nucleótidos consecutivos, seleccionada de la secuencia de nucleótidos del complemento de SEQ ID No. 2 desde el nucleótido 41 al nucleótido 452, y el otro cebador está constituido por 17 a 200 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos del complemento de SEQ ID No. 1 desde el nucleótido 99 al nucleótido 334 o la secuencia de nucleótidos de SEQ ID No. 2 desde el nucleótido 1 al nucleótido 40.
4. El método de la reivindicación 2, en donde un cebador comprende en su extremo 3’ último una secuencia de nucleótidos de al menos 17 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos de SEQ ID No. 1 desde el nucleótido 1 al nucleótido 98 o la secuencia de nucleótidos de SEQ ID No. 16 desde el nucleótido 1 al nucleótido 563 o dicho cebador comprende en su extremo 3’ último una secuencia de nucleótidos de al menos 17 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos del complemento de SEQ ID No. 2 desde el
nucleótido 41 al nucleótido 452, y el otro cebador comprende en su extremo 3’ último al menos 17 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos del complemento de SEQ ID No. 1 desde el nucleótido 99 al nucleótido 334 o la secuencia de nucleótidos de SEQ ID No. 2 desde el nucleótido 1 al nucleótido 40.
5. El método de la reivindicación 4, en donde dichos cebadores comprenden la secuencia de SEQ ID No. 3 y SEQ ID No. 6, respectivamente.
6. El método de la reivindicación 5, método que comprende amplificar un fragmento de 369 pares de bases utilizando el protocolo de identificación de eventos élite.
7. Un kit para identificación de una planta transgénicas de algodón resistente a los insectos, o células, partes, semilla o progenie de la misma especificadas en la reivindicación 1 en muestras biológicas, comprendiendo dicho kit un cebador que reconoce la secuencia de nucleótidos de SEQ ID No. 1 desde el nucleótido 1 al nucleótido 98 o la 45 secuencia de nucleótidos de SEQ ID No. 16 desde el nucleótido 1 al nucleótido 563 o un cebador reconoce la secuencia de nucleótidos del complemento de SEQ ID No. 2 desde el nucleótido 41 al nucleótido 452, y un cebador que reconoce la secuencia de nucleótidos del complemento de SEQ ID No. 1 desde el nucleótido 99 al nucleótido 334 o la secuencia de nucleótidos de SEQ ID No. 2 desde el nucleótido 1 al nucleótido 40.
8. El kit de la reivindicación 7, en donde un cebador está constituido por una secuencia de nucleótidos de 17 a 50 200 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos de SEQ ID No. 1 desde el nucleótido 1 al nucleótido 98 o la secuencia de nucleótidos de SEQ ID No. 16 desde el nucleótido 1 al nucleótido 563, o dicho cebador está constituido por una secuencia de nucleótidos de 17 a 200 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos del complemento de SEQ ID No. 2 desde el nucleótido 41 al nucleótido 452, y un cebador está constituido por 17 a 200 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos del
complemento de SEQ ID No. 1 desde el nucleótido 99 al nucleótido 334 o la secuencia de nucleótidos de SEQ ID No. 2 desde el nucleótido 1 al nucleótido 40.
9. El kit de la reivindicación 7, en donde un cebador comprende en su extremo 3’ último una secuencia de nucleótidos de al menos 17 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos de SEQ ID No. 1 desde el nucleótido 1 al nucleótido 98 o la secuencia de nucleótidos de SEQ ID No. 16 desde el nucleótido 1 al nucleótido 563, o un cebador comprende en su extremo 3’ último una secuencia de nucleótidos de al menos 17
nucleótidos consecutivos seleccionados de la secuencia de nucleótidos del complemento de SEQ ID No. 2 desde el nucleótido 41 al nucleótido 452, y uno comprende en su extremo 3’ último al menos 17 nucleótidos consecutivos seleccionados de la secuencia de nucleótidos del complemento de SEQ ID No. 1 desde el nucleótido 99 al nucleótido 334 o la secuencia de nucleótidos de SEQ ID No. 2 desde el nucleótido 1 al nucleótido 40.
10. El kit de la reivindicación 7, que comprende un cebador constituido por la secuencia de SEQ ID No. 3 y un 10 cebador constituido por la secuencia de SEQ ID No. 6.
11. Una molécula de ácido nucleico aislada que comprende una secuencia de nucleótidos de SEQ ID No. 1 desde el nucleótido 78 al nucleótido 119 o SEQ ID No. 2 desde el nucleótido 20 al 60, o el complemento de dichas secuencias.
800 pb
445 pb
400 pb
262 pb 600 pb517 pb369 pb 300 pb
Patentes similares o relacionadas:
Producción de partículas similares al virus de la gripe en plantas, del 6 de Mayo de 2020, de MEDICAGO INC.: Un ácido nucleico que comprende una región reguladora activa en una planta y un potenciador de la expresión activo en una planta, la región […]
Combinación de dos elementos genéticos para el control del desarrollo del tipo floral de una planta dicotiledónea, y utilización en procedimientos de detección y selección, del 1 de Abril de 2020, de Institut national de recherche pour l'agriculture, l'alimentation et l'environnement: Utilización de una combinación de dos elementos genéticos para el control del desarrollo del tipo floral de una planta dicotiledónea, comprendiendo dicha combinación, respectivamente: […]
Proteínas insecticidas y métodos para su uso, del 12 de Febrero de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Una construcción de ADN que comprende una molécula de ácido nucleico heteróloga que codifica un polipéptido de PIP-72 que tiene actividad insecticida contra el gusano de […]
Polinucleótidos y polipéptidos aislados, y métodos para usar los mismos para incrementar la eficiencia en el uso de nitrógeno, rendimiento, tasa de crecimiento, vigor, biomasa, contenido de aceite, y/o tolerancia al estrés abiótico, del 13 de Noviembre de 2019, de Evogene Ltd: Un método para incrementar la eficiencia en el uso de nitrógeno, biomasa en condiciones limitantes de nitrógeno, y/o tasa de crecimiento de una planta que comprende: […]
Moléculas pequeñas de ARN que median en la interferencia de ARN, del 22 de Octubre de 2019, de MAX-PLANCK-GESELLSCHAFT ZUR FORDERUNG DER WISSENSCHAFTEN E.V.: Molécula de ARN de doble hebra aislada, en la que cada hebra de ARN tiene una longitud de 19-25 nucleótidos y al menos una hebra tiene un saliente […]
Planta Brassica que comprende un alelo indehiscente mutante, del 11 de Septiembre de 2019, de BASF Agricultural Solutions Seed US LLC: Un alelo mutante defectivo parcial de un gen IND, en el que el gen IND comprende una molécula de ácido nucleico seleccionada del grupo que […]
Receptor de reconocimiento de patrones de plantas y quimeras del mismo para su uso contra infecciones bacterianas, del 11 de Septiembre de 2019, de EBERHARD-KARLS-UNIVERSITAT TUBINGEN: Un receptor de reconocimiento de patrones quiméricos (PRR) para reconocer patrones moleculares asociados a patógenos de plantas, que comprende al menos un ectodominio […]
Plantas de arroz que tienen tolerancia incrementada frente a herbicidas de imidazolinona, del 31 de Julio de 2019, de INSTITUTO NACIONAL DE TECNOLOGIA AGROPECUARIA: Un método para la caracterización molecular de una planta de arroz que comprende amplificar, mediante PCR, un gen AHAS de una planta de arroz, en donde la […]