Nucleótidos modificados para secuenciación de polinucleótidos.
Un método para convertir un compuesto de fórmula R-O-alilo a un compuesto correspondiente en el cual el grupo alilo es eliminado y reemplazado por hidrógeno,
comprendiendo dicho método la etapa de hacer reaccionar un compuesto de fórmula R-O-alilo en solución acuosa con un metal de transición y una fosfina soluble en agua, en donde R es una molécula de nucleósido, nucleótido o polinucleótido de fórmula PN-O-alilo, en donde PN es dicho nucleósido o nucleótido o es un nucleótido con terminal 3' de dicho polinucleótido.
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E12180077.
Solicitante: ILLUMINA CAMBRIDGE LIMITED.
Nacionalidad solicitante: Reino Unido.
Dirección: Chesterford Research Park Little ChesterfordSaffron WaldenEssex CB10 1XL REINO UNIDO.
Inventor/es: SMITH, MARK, LIU,XIAOHAI, MILTON,JOHN, BARNES,COLIN, WU,XIAOLIN, BRENNAN,JOSEPH, RUEDIGER,SILKE.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07H19/06 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07H AZUCARES; SUS DERIVADOS; NUCLEOSIDOS; NUCLEOTIDOS; ACIDOS NUCLEICOS (derivados de ácidos aldónicos o sacáricos C07C, C07D; ácidos aldónicos, ácidos sacáricos C07C 59/105, C07C 59/285; cianohidrinas C07C 255/16; glicales C07D; compuestos de constitución indeterminada C07G; polisacáridos, sus derivados C08B; ADN o ARN concerniente a la ingeniería genética, vectores, p. ej. plásmidos o su aislamiento, preparación o purificación C12N 15/00; industria del azúcar C13). › C07H 19/00 Compuestos que contienen un heterociclo que comparten un heteroátomo del ciclo con un radical sacárido; Nucleósidos; Mononucleótidos; Sus anhidro-derivados. › Radicales de pirimidina.
- C07H19/10 C07H 19/00 […] › con el radical sacárido esterificado por ácidos fosfóricos o polifosfóricos.
- C07H19/16 C07H 19/00 […] › Radicales de purina.
- C07H19/20 C07H 19/00 […] › con el radical sacárido esterificado por ácidos fosfóricos o polifosfóricos.
- C07H21/00 C07H […] › Compuestos que contienen al menos dos unidades mononucleótido que tienen cada una grupos fosfato o polifosfato distintos unidos a los radicales sacárido de los grupos nucleósido, p. ej. ácidos nucleicos.
- C12Q1/68 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2550513_T3.pdf


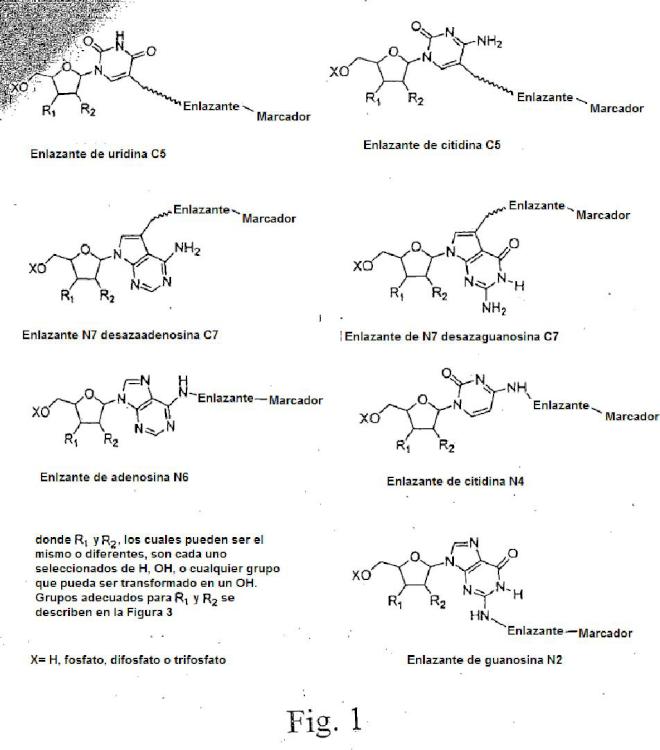
Fragmento de la descripción:
Nucleótidos modificados para secuenciación de polinucleótidos
La invención se relaciona con nucleótidos modificados. En particular, está invención divulga nucleótidos que tienen un grupo protector removible, su uso en métodos de secuenciación de polinucleótidos y un método para la 5 desprotección química del grupo protector.
Los avances en el estudio de las moléculas han sido guiados, en parte, por la mejora en tecnologías utilizadas para caracterizar las moléculas o sus reacciones biológicas. En particular, el estudio de los ácidos nucleicos ADN y ARN se ha beneficiado de las técnicas en desarrollo utilizadas para el análisis de secuencias y el estudio de eventos de hibridación.
10 Un ejemplo de las tecnologías que han mejorado el estudio de los ácidos nucleicos es el desarrollo de disposiciones fabricadas de ácidos nucleicos inmovilizados. Estas disposiciones consisten típicamente de una matriz de alta densidad de polinucleótidos inmovilizados sobre un material de soporte sólido. Véase, por ejemplo, Fodor et al., Trends Biotech. 12:19-26, 1994, la cual describe maneras de ensamblar los ácidos nucleicos utilizando una superficie de vidrio sensibilizada químicamente protegida por una máscara, pero expuesta en áreas definidas para 15 permitir la unión de fosforamiditas de nucleótidos modificadas adecuadamente. Las disposiciones fabricadas también pueden ser manufacturadas por la técnica de "manchar" polinucleótidos conocidos sobre un soporte sólido en posiciones predeterminadas (por ejemplo, Stimpson et al., Proc. Nati. Acad. Sci. USA 92: 6379-6383, 1995).
La secuenciación por síntesis de ADN requiere idealmente la incorporación controlada (esto es, uno a la vez) del nucleótido complementario correcto opuesto al oligonucleótido que está siendo secuenciado. Esto permite una 20 secuenciación precisa agregando nucleótidos en ciclos múltiples a medida que cada residuo de nucleótido es secuenciado uno a la vez, evitando así la ocurrencia de una serie no controlada de incorporaciones. El nucleótido incorporado es leído utilizando un marcador apropiado unido al mismo antes de la eliminación de la unidad estructural marcada y la subsecuente ronda siguiente de secuenciación. Con el fin de asegurar que ocurra solamente una incorporación individual, se requiere una modificación estructural ("grupo de bloqueo") de los 25 nucleótidos en secuenciación para asegurar una incorporación individual de nucleótido pero que evite cualquier incorporación de nucleótidos adicional en la cadena de polinucleótidos. El grupo bloqueador debe ser removible, bajo condiciones de reacción que no interfieran con la integridad del ADN que está siendo secuenciado. El ciclo de secuenciación puede continuar entonces con la incorporación del siguiente nucleótidos bloqueado, marcado. Con el fin de ser de uso práctico, el proceso completo debería consistir de etapas químicas y enzlmátlcas altamente 30 específicas, de alto rendimiento para facilitar múltiples ciclos de secuenciación.
Para ser útiles en la secuenciación de ADN, el nucleótido, y más usualmente los trifosfatos de nucleótido, requieren generalmente un grupo bloqueador de 3OH de tal manera que evite que la polimerasa utilizada para Incorporarlo en una cadena de polinucleótidos continúe replicando una vez que la base sobre el nucleótido es agregada. Hay muchas limitaciones sobre la adecuabilidad de una molécula como grupo bloqueador. Debe ser tal que evite que 35 moléculas adicionales de nucleótido se agreguen a la cadena de polinucleótidos mientras que simultáneamente sea fácil de retirar de la unidad estructural azúcar sin producir un daño a la cadena de polinucleótidos. Adlclonalmente, el nucleótido modificado debe ser tolerado por la polimerasa u otras enzimas apropiadas utilizadas para Incorporarlo en la cadena de polinucleótidos. El grupo bloqueador ideal por lo tanto exhibirá una estabilidad a largo plazo, será incorporado eficientemente por la enzima polimerasa, producirá un bloqueo total de Incorporaciones secundarias o 40 adicionales y tendrá la capacidad de ser retirado bajo condiciones moderadas que no produzcan daño a la estructura del polinucleótido, preferiblemente bajo condiciones acuosas. Estos requerimientos restrictivos son obstáculos formidables para el diseño y síntesis de los nucleótidos modificados requeridos.
Grupos bloqueadores reversibles para este propósito han sido descritos previamente pero ninguno de ellos satisface en general los criterios anteriores para la química de polinucleótidos, por ejemplo, que sean compatibles con ADN.
45 Metzker et al., (Nucleic Aclds Research, 22 (20): 4259-4267, 1994) divulga la síntesis y uso de ocho 5-trifosfatos de 2-desoxirribonucleósldo modificados en 3' (dNTPs modificados en 3) y su prueba en dos ensayos de plantilla de ADN para la actividad de Incorporación. Los dNTPs modificados en 3 incluyeron 3'alll desoxlrhboadenosina 5'- trifosfato (3-alil de ATP). Sin embargo, el compuesto bloqueado por 3alilo no fue utilizado para demostrar un ciclo completo de terminación, desprotección y relnlclaclón de la síntesis de ADN: los únicos resultados de la prueba 50 presentados fueron aquellos que mostraron la capacidad de este compuesto para terminar la síntesis de ADN en un ensayo de terminación sencillo, y de los ocho tales ensayos llevados a cabo, cada uno fue llevado a cabo con una ADN polimerasa diferente.
La WO 02/29003 (The Trustees of Columbia University en la ciudad de Nueva York) describe un método de secuenciación el cual, puede Incluir el uso de un grupo protector aillo para tapar el grupo 3'-OH en una cadena 55 creciente de ADN en una reacción con polimerasa. El grupo alilo es Introducido de acuerdo con el procedimiento de
Metzker (infra) y se dice que es retirado utilizando la metodología reportada por Kamal et al (Tet. Let, 40, 371-372, 1999).
La metodología de desprotección de Kamal emplea yoduro de sodio y el o rot ri m eti I si I a n o de tal manera que se genere in situ el yodotrimetilsilano, en acetonitrilo como solvente, deteniéndola con tiosulfato de sodio. Después de la extracción en acetato de etilo y secado (sulfato de sodio), la concentración posterior bajo presión reducida y cromatografía de columna (acetato de etilo: hexano; 2:3 como eluyente), se obtuvieron los alcoholes libres en un rendimiento de 90-98%.
En la WO 02/29003, se sugiere la desprotección de alilos según Kamal como directamente aplicable en el secuencialmente de ADN sin modificación, siendo las condiciones de Kamal moderadas y específicas.
Mientras que Metzker reporta la preparación de un nucleótido o nucleósido bloqueado con 3alilo y la WO 02/29003 sugiere el uso de la funcionalidad alilo como una tapa para 3-OH durante la secuenciación, ninguno de estos documentos enseña realmente la desprotección de un grupo hidroxilo con 3-alilo en el contexto de un protocolo de secuenciación. Mientras que el uso de un grupo alilo como grupo protector de hidroxilo es bien conocido - es fácil de introducir y es estable a lo largo del todo el rango de pH y a temperaturas elevadas - no hay hasta la fecha una realización concreta de una escisión exitosa de un grupo 3-alilo bajo condiciones compatibles con el ADN, esto es, condiciones bajo las cuales la integridad del ADN no es destruida total o parcialmente. En otras palabras, no ha sido posible hasta el momento llevar a cabo una secuenciación de ADN utilizando nucleótidos bloqueados con alilo en 3'OH.
La metodología de Kamal es inapropiada para ser llevada a cabo en medio acuoso puesto que el cloruro de TMS hidrolizará evitando la generación in situ de yoduro de TMS. Los intentos para llevar a cabo la desprotección de Kamal (en acetonitrilo) en secuenciación se han mostrado no exitosos en nuestras manos.
La presente invención está basada en el desarrollo sorprendente de un cierto número de grupos bloqueadores reversibles y métodos para desprotegerlos bajo condiciones compatibles con al ADN. Algunos de estos grupos bloqueadores son novedosos per se; otros han sido divulgados en la técnica anterior pero, como se anotó más arriba, no ha sido demostrada la posibilidad de utilizar estos grupos bloqueadores en la secuenciación de ADN.
Una característica de la invención se deriva del desarrollo de un método completamente nuevo de desprotección del alilo. Nuestro procedimiento es de amplia aplicabilidad en la desprotección de virtualmente todas las funcionalidades hidroxilo protegidas con alilo y puede efectuarse en solución acuosa, en contraste con la metodología de Kamal et al (la cual se efectúa en acetonitrilo) y con otros métodos conocidos generalmente en la técnica anterior los cuales son altamente sensibles al oxígeno y la humedad. Una característica... [Seguir leyendo]
Reivindicaciones:
1. Un método para convertir un compuesto de fórmula R-O-alilo a un compuesto correspondiente en el cual el grupo alilo es eliminado y reemplazado por hidrógeno, comprendiendo dicho método la etapa de hacer reaccionar un compuesto de fórmula R-O-alilo en solución acuosa con un metal de transición y una fosfina soluble en agua, en donde R es una molécula de nucleósido, nucleótido o polinucleótido de fórmula PN-O-alllo, en donde PN es dicho nucleósido o nucleótido o es un nucleótido con terminal 3 de dicho polinucleótido.
2. Una composición acuosa que comprende un metal de transición, una fosfina soluble en agua y un compuesto de fórmula R-O-alilo en donde R es una molécula de nucleósido, nucleótido o polinucleótido de fórmula PN-O-alllo, en donde PN es dicho nucleósido o nucleótido o es un nucleótido con terminal 3 de dicho polinucleótido.
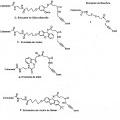
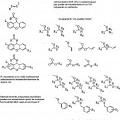
3. El método de la reivindicación 1 o la composición de la reivindicación 2 en donde dicho nucleósido, nucleótido o polinucleótido comprende adicionalmente un marcador detectable enlazado a la base de los mismos mediante un enlazante escindible o no escindible.
4. El método o composición de la reivindicación 3 en donde dicho marcador detectable es un fluoróforo.
5. El método o composición de la reivindicación 3 en donde dicho enlazante es lábil a ácidos, fotolábil o contiene un enlace disulfuro.
6. El método o composición de la reivindicación 3 en donde dicho grupo alilo y dicho marcador lábil son eliminados en una etapa sencilla.
7. El método de la reivindicación 1 o la composición de la reivindicación 2 en donde dicho metal de transición es seleccionado del grupo que comprende platino, paladio, rodio, rutenio, osmio e iridio.
8. El método de la reivindicación 1 o la composición de la reivindicación 2 en donde dicho metal de transición es paladio.
9. El método de la reivindicación 1 o la composición de la reivindicación 2 en donde dicha fosfina es una triaril fosfina derivada o trialquil fosfina derivada.
10. El método o composición de la reivindicación 9 en donde dicha fosfina es derivada con una o más funcionalidades seleccionadas del grupo que comprende grupos amino, hidroxilo, carboxilo y sulfonato.
11. El método o composición de la reivindicación 10 en donde la fosfina se selecciona de 3,3,3"-fosfidin tris (ácido bencenosulfónico) y tris(2-carboxietil)fosfinas y sus sales.
12. Un método para determinar la secuencia de un polinucleótido de cadena sencilla objetivo, que comprende monitorlzar la Incorporación secuencial de nucleótldos complementarios, en donde al menos una Incorporación es de un nucleótido que tiene un grupo 3-0-alilo y un marcador detectable enlazado a la base de nucleótido, en donde la identidad del nucleótido Incorporado es determinada detectando el marcador enlazado a la base, y el grupo bloqueador aillo es eliminado utilizando el método de las reivindicaciones 1-11.
13. El método de la reivindicación 12 en donde el marcador del nucleótido y el grupo bloqueador aillo son eliminados en una etapa de tratamiento químico sencilla.
14. El método de la reivindicación 12 en donde dicha etapa de incorporación es lograda mediante una Thermococcus pollmerasa modificada.
15. El método de las reivindicaciones 12-14 en donde cuatro nucleótldos son puestos en contacto con el objetivo simultáneamente, y los nucleótldos no incorporados son eliminados antes de la detección y subsecuentemente a la eliminación del marcador y del grupo bloqueador.
Patentes similares o relacionadas:
Proteínas de unión a interleuquina-13, del 15 de Julio de 2020, de AbbVie Bahamas Ltd: Un anticuerpo anti-IL-13 recombinante, o fragmento de unión a antígeno del mismo, en donde dicho anticuerpo anti-IL-13 recombinante, o fragmento de unión a antígeno del mismo, […]
Polinucleótidos aislados y métodos y plantas que usan los mismos para regular la acidez de las plantas, del 10 de Junio de 2020, de The State of Israel, Ministry of Agriculture and Rural Development, Agricultural Research Organization, (A.R.O.), Volcani Cent: Una célula de planta o una planta que comprende una construcción de ácido nucleico que comprende una secuencia de ácido nucleico que codifica un polipéptido […]
Métodos y composiciones para modular PD1, del 13 de Mayo de 2020, de Sangamo Therapeutics, Inc: Célula aislada que comprende una inserción o una deleción en un gen de PD1 endógeno dentro de, o entre, las secuencias mostradas en SEQ ID NO: 56 y SEQ ID NO: 60 del gen de PD1 […]
Compuesto de tioéter para la protección del grupo 2''-hidroxi en nucleósidos que van a ser utilizados en la síntesis de oligonucleótidos, del 6 de Mayo de 2020, de Bonac Corporation: Un éter representado por la siguiente fórmula química : **(Ver fórmula)** en dicha fórmula química , R4 es un grupo alquilo de cadena lineal o ramificada […]
Métodos de preparación de polinucleótidos usando composiciones de sales de catiónicas multivalentes, del 6 de Mayo de 2020, de GERON CORPORATION: Un método para preparar un polinucleótido, comprendiendo el método: a) poner en contacto una primera composición polinucleotídica con una sal catiónica […]
Nucleótidos modificados para secuenciación de polinucleótidos, del 8 de Abril de 2020, de ILLUMINA CAMBRIDGE LIMITED: Una molécula de nucleótido que tiene una unidad estructural de azúcar ribosa o desoxirribosa y una base enlazada a un marcador detectable a través de […]
Análogos de oligonucleótidos que incorporan 5-aza-citosina en los mismos, del 8 de Enero de 2020, de Astex Pharmaceuticals, Inc: Un análogo de oligonucleótido aislado o sintético, o una sal o éster del mismo, de fórmula general 5'-DpG-3' o 5'-GpD-3', en los que D es decitabina; p es un […]
Nucleótidos modificados, del 1 de Enero de 2020, de ILLUMINA CAMBRIDGE LIMITED: Un kit que comprende cuatro moléculas de nucleótido trifosfato modificadas, cada una de las cuales comprende una base de purina o pirimidina y una unidad estructural […]