Métodos para medir in vivo biomoléculas procedentes del sistema nervioso.
Un método in vitro para medir el metabolismo de una proteína in vivo en un sujeto que ha recibido una porción marcada que es capaz de cruzar la barrera hematoencefálica e incorporarse a dicha proteína cuando dicha proteína se sintetiza en el sistema nervioso central del sujeto,
comprendiendo el método:
a. detectar la cantidad de proteína marcada con la porción y la cantidad de proteína no marcada en una muestra de líquido cefalorraquídeo (LCR) obtenida del sujeto, comprendiendo la muestra de LCR una fracción proteica sintetizada en el SNC y marcada detectablemente con la porción, y una fracción proteica no marcada con la porción, donde la cantidad de proteína marcada y la cantidad de proteína no marcada se detectan usando espectrometría de masas;
b. determinar la relación de proteína marcada respecto a la proteína no marcada;
c. calcular la relación de síntesis fraccional (TSF) de proteína marcada y/o la relación de aclaramiento fraccional (TAF) de proteína marcada a partir de la relación determinada mediante el paso (b) en las muestras a lo largo del tiempo;
en el que la tasa de síntesis fraccional (FSR) de proteína marcada se calcula a partir de las relaciones determinadas mediante el paso (b) en múltiples muestras desde el momento en el que es detectable la proteína marcada y mientras se incrementa la relación de proteína marcada respecto a la no marcada en las múltiples muestras, determinada mediante el paso (b); y/o la tasa de aclaramiento fraccional (TAF) de la proteína marcada se calcula a partir de las relaciones determinadas en el paso (b) en múltiples muestras después de que la relación de proteína marcada respecto a la no marcada disminuye en el LCR del sujeto;
en el que la TSF y/o TAF son directamente proporcionales al metabolismo en el SNC de la proteína.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2006/012200.
Solicitante: Washington University in St. Louis.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: ONE BROOKINGS DRIVE ST. LOUIS, MO 63110 ESTADOS UNIDOS DE AMERICA.
Inventor/es: BATEMAN,RANDALL JOHN, HOLTZMAN,DAVID MICHAEL.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- G01M15/00 FISICA. › G01 METROLOGIA; ENSAYOS. › G01M ENSAYO DEL EQUILIBRADO ESTATICO O DINAMICO DE MAQUINAS O ESTRUCTURAS; ENSAYO DE ESTRUCTURAS O APARATOS, NO PREVISTOS EN OTRO LUGAR. › Ensayos de motores.
PDF original: ES-2498973_T3.pdf


Fragmento de la descripción:
Métodos para medir in vivo biomoléculas procedentes del sistema nervioso 5 Campo de la invención
[1] La invención describe métodos para el diagnóstico y el tratamiento de enfermedades y trastornos neurológicos y neurodegenerativos, y procesos asociados. La invención se refiere a un método para medir el metabolismo de biomoléculas procedentes del sistema nervioso central en un sujeto in vivo.
Antecedentes de la invención
Enfermedad de Alzheimer
[2] La enfermedad de Alzheimer (EA) es la causa más común de demencia y es un problema de salud pública creciente. Actualmente se estima que hay 5 millones de personas afectadas en los Estados Unidos, con un incremento esperado de 13 millones para el año 25 (Herbert et al 21, Alzheimer Dis. Assoc. Disord. 15(4): 169- 173). La EA, como otras enfermedades neurodegenerativas del sistema nervioso central (SNC), se caracteriza por alteraciones de la producción, acumulación y aclaramiento de proteínas. En la EA, la desregulación del metabolismo 2 de la protema beta-amiloide (AB), está indicada por una acumulación masiva de esta proteína en los cerebros de aquellos que tienen la enfermedad. La EA conduce a una pérdida de la memoria, de la función cognitiva y, finalmente, de la independencia. Esto acarrea una pesada carga personal y financiera al paciente y a la familia. Debido a la severidad y al incremento de la prevalencia de esta enfermedad en la población, es urgente que se desarrollen mejores tratamientos.
[3] Actualmente existen algunos medicamentos que modifican los síntomas, sin embargo no existen tratamientos que modifiquen la enfermedad. Los tratamientos que modifican la enfermedad probablemente serán los más efectivos cuando se den antes de la aparición del daño cerebral permanente. Sin embargo, en el momento que se hace el diagnóstico clínico de la EA, ya ha ocurrido una pérdida neuronal extensa (Price et al. 21, Archiv. Neurol.
58(9): 1395-142). Por lo tanto, un modo de identificar a aquellos que tienen riesgo de desarrollar EA sería lo que más ayudaría a prevenir o retrasar la aparición de la EA. Actualmente, no existen medios para identificar los cambios fisiopatológicos que ocurren en la EA antes de la aparición de síntomas clínicos o de medir eficazmente los efectos de los tratamientos que podrían prevenir la aparición o ralentizar la progresión de la enfermedad. En el documento US 23/228259 se mide el metabolismo de AB en orina.
[4] Existe la necesidad, por lo tanto, de un método sensible, exacto y reproducible para medir el metabolismo in vivo de las biomoléculas en el SNC. En particular, es necesario un método para medir la tasa de síntesis y la tasa de aclaramiento fraccional in vivo de las proteínas asociadas con una enfermedad neurodegenerativa, por ejemplo, el metabolismo de AB en la EA.
Sumario de la invención
[5] La presente solicitud se define en su más amplio sentido en las reivindicaciones anexas. Un aspecto de la invención es la provisión de un método in vitro para medir el metabolismo in vivo de una proteína en un sujeto que
ha recibido un resto marcado que es capaz de cruzar la barrera hametoencefálica y de incorporarse en dicha proteína cuando dicha proteína se sintetiza en el sistema nervioso central del sujeto, comprendiendo el método:
a. detectar la cantidad de proteína marcada con el resto y la cantidad de proteína no marcada en una muestra de líquido cefalorraquídeo (LCR) obtenida del sujeto, comprendiendo la muestra de LCR una fracción proteica
sintetizada en el SNC y marcada detectablemente con el resto, y una fracción proteica no marcada con el resto, donde la cantidad de proteína marcada y la cantidad de proteína no marcada se detecta usando espectrometría de masas;
b. determinar la proporción de proteína marcada respecto a la proteína no marcada;
c. calcular la tasa de síntesis fraccional (TSF) de proteína marcada y/o la tasa de aclaramiento fraccional (TAF) de proteína marcada a partir de la proporción determinada mediante el paso (b) en las muestras a lo largo del tiempo;
en el que la tasa de síntesis fraccional (FSR) de proteína marcada se calcula a partir de las proporciones determinadas mediante el paso (b) en múltiples muestras desde el momento en el que la proteína marcada es detectable y mientras se incrementa la tasa de proteína marcada respecto a la no marcada en las muestras, determinada mediante el paso (b); y/o la tasa de aclaramiento fraccional (TAF) de la proteína marcada se calcula a partir de las proporciones determinadas en el paso (b) en múltiples muestras después de que la proporción de proteínas marcadas respecto a las proteínas no marcadas disminuya en el LCR del sujeto;
en el que la TSF y/o la TAF es directamente proporcional al metabolismo en el SNC de la proteína.
Breve descripción de las figuras
[6]
La Figura 1 representa un esquema que ilustra el procesamiento de la proteína precursora de amiloide (APP) en beta-amllolde (AB) dentro de una célula. Las leucinas (L), uno de los posibles sitios de mareaje, se indican en negro. La secuencia de aminoácidos de AB se muestra en la parte Inferior, con los sitios de digestión por tripsina indicados para demostrar los fragmentos que se analizaron por espectrometría de masas.
La Figura 2 representa una gráfica de espectrometría de masas que muestra la separación de los péptidos beta-amiloides. Los péptidos AB se inmunoprecipitaron de LCR humano con el anticuerpo anti-AB de dominio central, m266, y la AB eluida se sometió a espectrometría de masas. Los picos de la espectrometría de masas están marcados con sus variantes peptídicas correspondientes: AB38, AB39, AB4 y AB42.
La Figura 3 presenta las gráficas de espectrometría de masas que ilustran la variación en el peso molecular del fragmento AB17-28 marcado con 13C. En el panel A, se recogieron y se inmunoprecipitaron medios no marcados de una línea celular de neuroglioma humano que produce AB in vitro. Después, los péptidos beta-amiloides se escindieron con tripsina en los sitios 5, 16 y 28 (véase la Figura 1) produciendo las dos envueltas de fragmentos que se muestran a las masas de 1325 y 1336. Obsérvese que las dos masas de las envueltas de los fragmentos AB, A&17-28 (1325) y AB6-16 (1336) muestran la distribución estadística de los isótopos naturales en AB no marcado. También, obsérvese que no hay señal a una masa de 1331, donde debería estar la señal del mareaje. En el panel B se recogieron medios de células humanas de neuroglioma cultivadas durante 24 horas en presencia de 13C6-leucina y se inmunoprecipitó y se escindió AB con tripsina para producir las envueltas de los fragmentos que se muestran a las masas de 1325, 1331 y 1336. Obsérvese la variación de la masa (flecha) de AI3.17-28 de 1325 a 1331, que demuestra el mareaje con 13C6-leucina (AB*i7-2s). AB6-16 no contiene leucina, así que no está marcado ni su masa se modifica. Una cantidad minoritaria de of AB17-28 permanece sin marcar.
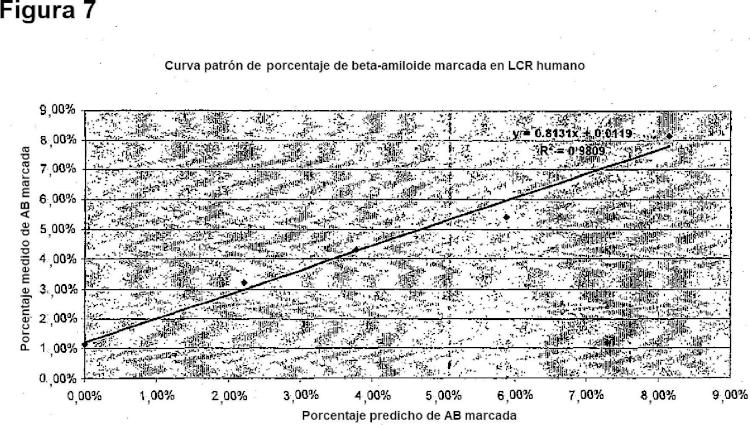
La Figura 4 representa una gráfica que muestra una curva patrón del mareaje de AB in vitro. Se hizo una dilución seriada de una muestra de medio de cultivo marcado para generar una curva patrón para ensayar la linealidad y la variabilidad de la técnica de medición. La AB se precipitó del medio, se digirió con tripsina, y los fragmentos se analizaron en un espectrómetro de masas con cromatografía líquida de inyección por electronebulización (LC- ESI) y los Iones de los espectros de masa en tándem se cualificaron usando un programa informático diseñado de manera personalizada. El programa informático sumó tanto los iones tándem marcados como los no marcados y calculó la proporción de AB marcada respecto del total. El porcentaje de AB marcada frente al valor predicho se muestra con una línea de regresión lineal. Obsérvese el buen ajuste lineal, además de la baja desviación.
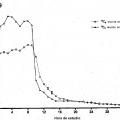
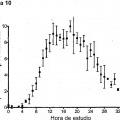
La Figura 5 representa un esquema que ilustra el protocolo de mareaje in vivo. Se muestra un diagrama de un participante con un catéter intravenoso en la vena antecubital y un catéter lumbar en el espacio intratecal entre L3-4. En un catéter IV, se infundió leucina marcada con 13C6 a una tasa de 1,8 a 2,5 mg/kg/h durante 9 o 12 horas, después de un bolo inicial de 2 mg/kg. Se obtuvieron 12 mi de plasma a través del otro catéter IV cada hora durante las primeras 16 horas y cada dos horas de allí en adelante, tal como se representa. Se obtuvieron seis mi de LCR a través del catéter lumbar cada hora. Cada muestra se procesó después mediante inmunoprecipitación de AB, digestión con tripsina, y análisis de LC-ESI-MS para determinar el porcentaje de AB marcada en cada punto de tiempo.
La Figura 6 representa gráficas de espectrometría... [Seguir leyendo]
Reivindicaciones:
1. Un método in vitro para medir el metabolismo de una proteína in vivo en un sujeto que ha recibido una porción marcada que es capaz de cruzar la barrera hematoencefálica e incorporarse a dicha proteína cuando dicha proteína 5 se sintetiza en el sistema nervioso central del sujeto, comprendiendo el método:
a. detectar la cantidad de proteína marcada con la porción y la cantidad de proteína no marcada en una muestra de líquido cefalorraquídeo (LCR) obtenida del sujeto, comprendiendo la muestra de LCR una fracción proteica sintetizada en el SNC y marcada detectablemente con la porción, y una fracción proteica no marcada con la
porción, donde la cantidad de proteína marcada y la cantidad de proteína no marcada se detectan usando espectrometría de masas;
b. determinar la relación de proteína marcada respecto a la proteína no marcada;
c. calcular la relación de síntesis fraccional (TSF) de proteína marcada y/o la relación de aclaramiento fraccional (TAF) de proteína marcada a partir de la relación determinada mediante el paso (b) en las muestras a lo largo del
tiempo;
en el que la tasa de síntesis fraccional (FSR) de proteína marcada se calcula a partir de las relaciones determinadas mediante el paso (b) en múltiples muestras desde el momento en el que es detectable la proteína marcada y mientras se incrementa la relación de proteína marcada respecto a la no marcada en las múltiples 2 muestras, determinada mediante el paso (b); y/o la tasa de aclaramiento fraccional (TAF) de la proteína marcada se calcula a partir de las relaciones determinadas en el paso (b) en múltiples muestras después de que la relación de proteína marcada respecto a la no marcada disminuye en el LCR del sujeto; en el que la TSF y/o TAF son directamente proporcionales al metabolismo en el SNC de la proteína.
2. El método de la reivindicación 1 en el que la proteína es beta-amiloide y en el que, para el cálculo de la TSF, la proteína marcada es detectable 4 ó 6 horas después de la administración de la porción marcada y la relación de proteína marcada respecto a la no marcada se incrementa durante 12, 13 6 15 después de la administración de la porción marcada;
y en el que, para el cálculo de la TAF, la relación de proteína marcada respecto a la no marcada disminuye 24-36 3 horas después de la administración de la porción marcada.
3. El método de la reivindicación 1, en el que la muestra de LCR se obtuvo mediante punción lumbar, catéter lumbar o una combinación de los mismos.
4. El método de cualquiera de las reivindicaciones precedentes, en el que la proteína se selecciona del grupo que consiste en beta-amiloide, apolipoproteína E, apolipoproteína J, sinucleína, precursor soluble de la proteína amiloide, Tau, alfa-2 macroglobulina, S1B, proteína básica de la mielina, una interleucina y TNF.
5. El método de cualquiera de las reivindicaciones precedentes, en el que la porción marcada es un átomo, o una 4 molécula con un átomo marcado.
6. El método según la reivindicación 5 en el que la molécula es un aminoácido.
7. El método de la reivindicación 5 ó 6, en el que el átomo es un isótopo no radiactivo seleccionado del grupo que
consiste en 2H, 13C, 15N, 17, 1sO, 33S, 34S y 36S.
8. El método según las reivindicaciones 5, 6 ó 7 en el que la porción marcada es 13C6-leucina.
9. El método de cualquiera de las reivindicaciones precedentes, que comprende además separar la fracción proteica 5 marcada y la fracción proteica no marcada de la muestra de LCR.
1. El método de la reivindicación 9 en el que la proteína se separa por inmunoprecipitación.
Patentes similares o relacionadas:
Medio de almacenamiento informático, producto de programa informático, y método y dispositivo para monitorizar el mal funcionamiento de una turbina eólica, del 1 de Enero de 2020, de BEIJING GOLDWIND SCIENCE & CREATION WINDPOWER EQUIPMENT CO., LTD: Método para monitorizar una falla de un grupo electrógeno eólico, que comprende: realizar identificación de falla en un componente en el grupo electrógeno […]
Determinación de la pérdida de rendimiento energético de una turbina eólica, del 14 de Noviembre de 2019, de Senvion GmbH: Procedimiento para determinar una pérdida de rendimiento energético de una primera turbina eólica de un parque eólico con varias turbinas eólicas […]
Método y sistema para su uso en una prueba dinamométrica de un vehículo motorizado, del 27 de Marzo de 2019, de Rototest International AB: Método para su uso en pruebas dinamométricas de un vehículo , en el que el vehículo incluye al menos un primer eje de ruedas y […]
Banco de pruebas de transmisión de vehículo y método de control del mismo, del 20 de Febrero de 2019, de Horiba Instruments, Inc: Un banco de pruebas que comprende: un embrague dispuesto operativamente con un dinamómetro y un volante de inercia , estando el banco de pruebas […]
 Método y un dispositivo para parametrizar un sensor, del 16 de Diciembre de 2015, de AVL LIST GMBH: Método para parametrizar un sensor que se encuentra colocado sobre un objeto bajo prueba en un banco de pruebas , caracterizado porque el […]
Método y un dispositivo para parametrizar un sensor, del 16 de Diciembre de 2015, de AVL LIST GMBH: Método para parametrizar un sensor que se encuentra colocado sobre un objeto bajo prueba en un banco de pruebas , caracterizado porque el […]
Método para la alimentación de un motor de combustión interna con gas de combustión acondicionado, dispositivo para la ejecución del método mencionado, método para la determinación de las cantidades de contaminante en el gas de escape de un motor de combustión interna, y dispositivo para la ejecución del método mencionado, del 1 de Abril de 2015, de AVL LIST GMBH: Método para la alimentación de un motor de combustión interna con gas de combustión acondicionado, particularmente aire, preferentemente en bancos […]
 MÉTODO Y DISPOSITIVO DE CONVERSION PARA ENSAYAR AEROGENERADORES EN CAMPO, del 2 de Julio de 2013, de GAMESA INNOVATION & TECHNOLOGY, S.L: Método y dispositivo de conversión para ensayar aerogeneradores en campo. El método comprende la conexión del aerogenerador a una red eléctrica , ambos configurados […]
MÉTODO Y DISPOSITIVO DE CONVERSION PARA ENSAYAR AEROGENERADORES EN CAMPO, del 2 de Julio de 2013, de GAMESA INNOVATION & TECHNOLOGY, S.L: Método y dispositivo de conversión para ensayar aerogeneradores en campo. El método comprende la conexión del aerogenerador a una red eléctrica , ambos configurados […]
PROCEDIMIENTO Y DISPOSICIÓN PARA PRUEBAS DE FUNCIONAMIENTO TOTALMENTE AUTOMÁTICAS DE MOTORES DE COMBUSTIÓN INTERNA, del 13 de Febrero de 2012, de AUDI AG: Procedimiento para la prueba de funcionamiento de los motores de combustión interna en vehículos de motor, en el cual se conecta un sistema externo […]