INTERNALIZACION.
Un procedimiento para recuperar una molécula de ácido nucleico que codifica un miembro de unión de un complejo internalizado en una célula eucariota,
que comprende las siguientes etapas de: (a) poner en contacto una célula eucariota con una colección diversa de partículas de bacteriófago, en la que cada una o sustancialmente todas dichas partículas de bacteriófago presentan un miembro de unión en su superficie, en la que dicho miembro de unión se presenta como un (poli)péptido que no es de fusión con una proteína de cubierta de fago de dicha partícula de bacteriófago y en la que cada una o sustancialmente todas dichas partículas de bacteriófago comprenden una molécula de ácido nucleico que codifica el miembro de unión presentado, (b) permitir la unión del miembro de unión presentado sobre la partícula de bacteriófago a su diana, permitiendo de esta manera la formación de al menos un complejo, comprendiendo cada uno de dichos complejos una partícula de bacteriófago con su miembro de unión presentado y su diana, (c) cultivar la célula en condiciones que permitan la internalización de al menos uno de dichos complejos en la célula, (d) eluir las moléculas de ácido nucleico que codifican un miembro de unión que no se internaliza en condiciones en las que sustancialmente no se produce lisis celular, (e) lisar la célula que comprende los complejos internalizados y (f) recuperar a partir de la célula lisada la molécula de ácido nucleico que codifica un miembro de unión derivado de al menos uno de los complejos internalizados
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2007/063843.
Solicitante: MORPHOSYS AG.
Nacionalidad solicitante: Alemania.
Dirección: LENA-CHRIST-STRASSE 48 82152 MARTINSRIED/MUNCHEN ALEMANIA.
Inventor/es: ENZELBERGER,MARKUS.
Fecha de Publicación: .
Fecha Solicitud PCT: 12 de Diciembre de 2007.
Fecha Concesión Europea: 21 de Julio de 2010.
Clasificación Internacional de Patentes:
- C12N15/10C1
Clasificación PCT:
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
Fragmento de la descripción:
ANTECEDENTES DE LA INVENCIÓN
Se han identificado nuevas dianas mediante análisis comparativos y estadísticos de pacientes sanos y enfermos, en particular mediante el análisis de tejidos y/o plasma procedente de sangre de dichos pacientes. Normalmente, el análisis comparativo puede realizarse sobre diferentes niveles, tales como sobre los niveles de ADN, ARN, proteínas y postraduccionales. Una técnica usada comúnmente se basa en el análisis de la expresión diferencial de genes. En resumen, se marca ARNm procedente de células enfermas y sanas y posteriormente se hibrida con un chip de genes y se cuantifica. La regulación positiva o negativa de diferentes ARNm, basada en las señales de cuantificación, revela nuevas dianas potenciales. Otra estrategia bien conocida en la técnica anterior se basa en la identificación y comparación de patrones de metilación de ADN de moléculas de ADN procedentes de pacientes sanos y enfermos.
Sin embargo, en el contexto anterior debe entenderse que ni la modificación de ADN (es decir, el patrón de metilación de ADN) ni el análisis de expresión diferencial de genes (es decir el nivel de ARNm expresado en una célula) necesariamente refleja si una proteína específica codificada por el ADN correspondiente o el ARNm correspondiente efectivamente se expresa. Por lo tanto, sigue siendo un desafío la identificación de niveles de expresión diferencial, es decir, el análisis cuantitativo y también cualitativo de patrones de expresión de proteínas de células sanas en comparación con células enfermas.
Un procedimiento para la identificación de niveles de expresión diferencial de proteínas se basa en el análisis diferencial en gel bidimensional de dichas proteínas con el análisis posterior por espectrometría de masas, una técnica bien conocida para el experto en la materia. Además, son bien conocidos para un experto en la materia procedimientos basados en la fraccionación de proteínas tales como, mencionando sólo algunos, técnicas basadas en el uso de chips de proteínas y técnicas relacionadas con HPLC y FPLC.
Las técnicas de presentación en fagos ofrecen, por ejemplo, la posibilidad de reducir una biblioteca grande, por ejemplo, una biblioteca de expresión de miembros de unión, en muestras, tales como tejidos o células que, por ejemplo, proceden de un donante sano, y usar la población residual de la biblioteca en muestras, tales como tejidos o células, que proceden, por ejemplo, de un donante enfermo. Los miembros de unión que se han rastreado mediante el análisis de reducción, es decir, que se unen a dianas (poli)peptídicas u homólogos de tejidos/células enfermas pero no de tejidos/células sanas, normalmente se considera que se unen a una diana que se expresa de forma única (o al menos en un nivel mucho mayor) en las células diana (por ejemplo, la célula enferma). Posteriormente, pueden identificarse complejos de miembro de unión/diana (poli)peptídica, por ejemplo, por espectrometría de masas o procedimientos para el análisis de proteínas bien conocidos para el experto en la materia.
Son de un interés particular miembros de unión que se internalizan tras la unión a su diana. Dichos miembros de unión de internalización se conocen en la técnica (véase, por ejemplo, el documento WO 99/55367 o Biochimica et Biophysica Acta 1591 (2002) 109-118). Sin embargo, dichos miembros de unión se descubrieron por procedimientos diferentes de los procedimientos superiores descritos por la presente invención. Un experto en la materia sabe que dichos miembros de unión pueden, por ejemplo, fusionarse a cualquier sustancia o cualquier molécula pequeña que podría ser tóxica para la célula, desencadenando de esta manera la destrucción de la célula, preferentemente enferma, que expresa dicha o dichas dianas, siendo preferentemente dicha célula una célula enferma. Como también es conocido en la técnica, una vez que una diana de interés que se internaliza se ha identificado como, por ejemplo, una célula enferma o cancerosa, es posible determinar miembros de unión adicionales con, por ejemplo, mayor afinidad por la diana y/o mayor potencial para desencadenar la internalización de dicha diana. Dichos miembros de unión mejorados entonces pueden considerarse, por ejemplo, fármacos para tratar, por ejemplo, células enfermas que expresan dicha o dichas dianas.
Para determinar eficazmente las dianas potenciales que se han internalizado en la célula tras la unión de su miembro de unión respectivo, sería deseable separar los complejos internalizados de los complejos no internalizados. Sin embargo, en la técnica anterior, dicha separación no se ha conseguido de una manera satisfactoria cualitativa y cuantitativa confrontando de esta manera al experto en la materia con técnicas que requieren mucho tiempo y complejas para determinar miembros de unión y dianas que se internalizan.
Por lo tanto, sigue existiendo la necesidad de desarrollar adicionalmente y también mejorar procedimientos y procesos que permitan una separación eficaz entre complejos internalizados y no internalizados.
La presente invención satisface esta necesidad presentando miembros de unión como polipéptidos que no son de fusión sobre proteínas de la cubierta de fagos bacterianos. Esto puede conseguirse por diversos sistemas de presentación, por ejemplo, los desvelados en el documento WO 01/05950. BREVE DESCRIPCIÓN DE LAS FIGURAS
La Figura 1 muestra la eficacia de la internalización de dianas tras la unión a Fab.
Se calculó el porcentaje (%) de internalización a partir de la relación de señal extracelular en la superficie celular a 4ºC frente a 37ºC; la recuperación de la fluorescencia se midió mediante la relación de tinción extracelular más intracelular a 4ºC frente a 37ºC, mostrando que no se perdía ninguna o sólo se perdían unas pocas partículas de fago durante el proceso de internalización, el tratamiento con saporina y/o la tinción. Fab A mostró una internalización del 80%, Fab B sólo se internalizó con un 20% y Fab C no mostró ninguna unión en absoluto.
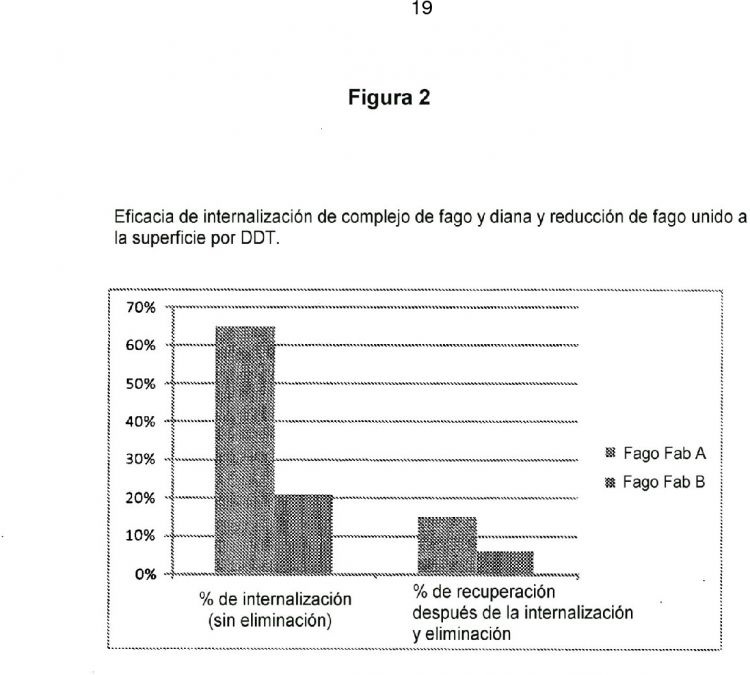
La Figura 2 muestra la eficacia de la internalización de complejos de fago y diana y la reducción del fago unido a la superficie por DDT.
Se muestra la internalización de fagos que presentan a través de un enlace disulfuro Fab A contra un antígeno que predominantemente se internaliza frente a fagos que presentan a través de un enlace disulfuro un Fab B contra un antígeno que predominantemente no se internaliza.
El porcentaje (%) de internalización se calculó a partir de la relación de señal extracelular sobre la superficie celular a 4ºC frente a 37ºC; la recuperación de la fluorescencia se midió mediante la relación de tinción extracelular más intracelular a 4ºC frente a 37ºC. DESCRIPCIÓN DETALLADA DE LA INVENCIÓN
La presente invención se refiere, en un aspecto, a un procedimiento para recuperar una molécula de ácido nucleico que codifica un miembro de unión de un complejo internalizado en una célula, que comprende las siguientes etapas de (a) poner en contacto una célula con una colección diversa de partículas de bacteriófago, en la que cada una o sustancialmente todas dichas partículas de bacteriófago presentan un miembro de unión en su superficie, en la que dicho miembro de unión se presenta como un (poli)péptido que no es de fusión con una proteína de cubierta de fago de dicha partícula de bacteriófago y en la que cada una o sustancialmente todas dichas partículas de bacteriófago comprenden una molécula de ácido nucleico que codifica el miembro de unión presentado, (b) permitir la unión del miembro de unión presentado sobre la partícula de bacteriófago a su diana, permitiendo de esta manera la formación de al menos un complejo, comprendiendo cada uno de dichos complejos una partícula de bacteriófago con su miembro de unión presentado y su diana, (c) cultivar la célula en condiciones que permitan la internalización de al menos uno de dichos complejos en la célula, (d) eluir las moléculas de ácido nucleico que codifican un miembro de unión que no se internaliza en condiciones en las que sustancialmente no se produce lisis celular, (e) lisar la célula que comprende los complejos internalizados y (f) recuperar a partir de la célula lisada la molécula de ácido nucleico que codifica un miembro de unión derivado de al menos uno de los complejos internalizados.
El término “célula” se refiere a cualquier célula eucariota o procariota. En relación con la presente invención se prefieren las células de mamífero....
Reivindicaciones:
1. Un procedimiento para recuperar una molécula de ácido nucleico que codifica un miembro de unión de un complejo internalizado en una célula eucariota, que comprende las siguientes etapas de:
(a) poner en contacto una célula eucariota con una colección diversa de partículas de bacteriófago, en la que cada una o sustancialmente todas dichas partículas de bacteriófago presentan un miembro de unión en su superficie, en la que dicho miembro de unión se presenta como un (poli)péptido que no es de fusión con una proteína de cubierta de fago de dicha partícula de bacteriófago y en la que cada una o sustancialmente todas dichas partículas de bacteriófago comprenden una molécula de ácido nucleico que codifica el miembro de unión presentado,
(b) permitir la unión del miembro de unión presentado sobre la partícula de bacteriófago a su diana, permitiendo de esta manera la formación de al menos un complejo, comprendiendo cada uno de dichos complejos una partícula de bacteriófago con su miembro de unión presentado y su diana,
(c) cultivar la célula en condiciones que permitan la internalización de al menos uno de dichos complejos en la célula,
(d) eluir las moléculas de ácido nucleico que codifican un miembro de unión que no se internaliza en condiciones en las que sustancialmente no se produce lisis celular,
(e) lisar la célula que comprende los complejos internalizados y
(f) recuperar a partir de la célula lisada la molécula de ácido nucleico que codifica un miembro de unión derivado de al menos uno de los complejos internalizados.
2. El procedimiento de la reivindicación 1, que comprende además la etapa de determinar la secuencia de la diana del complejo internalizado.
3. El procedimiento de la reivindicación 1, en el que dicha presentación como (poli)péptido de no fusión se caracteriza por un enlace no peptídico entre la proteína de cubierta de fago y el miembro de unión.
4. El procedimiento de la reivindicación 3, en el que dicho enlace no peptídico es un enlace disulfuro.
5. El procedimiento de la reivindicación 4, en el que dicho enlace disulfuro se genera entre
un primer resto de cisteína comprendido en dicha proteína de cubierta de fago y un segundo resto de cisteína comprendido en dicho miembro de unión.
6. El procedimiento de la reivindicación 4 ó 5, en el que dicha etapa de elución de dichas
5 moléculas de ácido nucleico que codifican un miembro de unión que no se internaliza se realiza en condiciones reductoras de tal forma que se escinde dicho enlace disulfuro.
7. El procedimiento de una cualquiera de las reivindicaciones 1 a 6, en el que dicha
recuperación de la célula lisada de dicha molécula de ácido nucleico que codifica el miembro 10 de unión se consigue por PCR.
8. El procedimiento de la reivindicación 2, en el que dicha etapa de determinación de la secuencia de la diana en el complejo internalizado se consigue por espectrometría de masas.
Patentes similares o relacionadas:
BIBLIOTECA DEL DOMINIO KUNITZ, del 5 de Julio de 2011, de DYAX CORPORATION: Una biblioteca que comprende una pluralidad de partículas de fago filamentoso, cada partícula de fago de la pluralidad incluyendo (i) una proteína […]
VECTOR PARA LA SELECCIÓN EFICAZ Y/O MADURACIÓN DE UN ANTICUERPO Y USOS DEL MISMO, del 28 de Junio de 2011, de SIGMA-TAU INDUSTRIE FARMACEUTICHE RIUNITE S.P.A. TECNOGEN S.P.A: Un vector adecuado para eficiente selección y/o maduración de un anticuerpo recombinante perteneciente al grupo de: ScFv, fragmentos activos de Abs, secuencias humanizadas […]
GENERACIÓN DE PROTEINAS DE UNIÓN SINTÉTICAS BASADAS EN PROTEINAS DE UBIQUITINA, del 16 de Junio de 2011, de SCIL PROTEINS GMBH: Procedimiento para la preparación de una proteína, presentando el procedimiento las siguientes etapas: a) seleccionar una proteína que se va […]
BIBLIOTECAS FOCALIZADAS DE PAQUETES GENÉTICOS, del 6 de Junio de 2011, de DYAX CORP.: Biblioteca de ADN focalizada que comprende secuencias de ADN variegadas que codifican: por lo menos una RDC1 de cadena pesada seleccionada de entre el grupo constituido […]
GENERACIÓN DE ELEMENTOS DE UNIÓN ESPECÍFICA QUE SE UNEN A (POLI)PÉPTIDOS CODIFICADOS POR FRAGMENTOS DE ADN GENÓMICO O EST, del 28 de Febrero de 2011, de MORPHOSYS AG: Un procedimiento para generar un elemento de unión específica a un (poli)péptido que está codificado por una secuencia de ácido nucleico comprendida en un fragmento […]
 FAGÉMIDOS PARA EL RASTREO DE ANTICUERPOS, del 23 de Febrero de 2011, de DEUTSCHES KREBSFORSCHUNGSZENTRUM STIFTUNG DES OFFENTLICHEN RECHTS: Uso de un fagémido que comprende ADN que codifica una proteína de fusión anticuerpo-proteína pIII de colifago en el que la proteína pIII de colifago comprende el dominio amino […]
FAGÉMIDOS PARA EL RASTREO DE ANTICUERPOS, del 23 de Febrero de 2011, de DEUTSCHES KREBSFORSCHUNGSZENTRUM STIFTUNG DES OFFENTLICHEN RECHTS: Uso de un fagémido que comprende ADN que codifica una proteína de fusión anticuerpo-proteína pIII de colifago en el que la proteína pIII de colifago comprende el dominio amino […]
 FAGOS QUE PRESENTAN EPÍTOPES MEJORADOS, del 9 de Febrero de 2011, de DYAX CORP.: Un método para identificar proteínas de fijación que se fijan a una diana, comprendiendo el método los pasos de (a) proporcionar una biblioteca […]
FAGOS QUE PRESENTAN EPÍTOPES MEJORADOS, del 9 de Febrero de 2011, de DYAX CORP.: Un método para identificar proteínas de fijación que se fijan a una diana, comprendiendo el método los pasos de (a) proporcionar una biblioteca […]
SECUENCIA LIDER UNIVERSAL DE GAS1, del 7 de Febrero de 2011, de DOMANTIS LIMITED: Un bacteriófago que comprende una molécula polinucleotídica que comprenden un promotor unido operablemente a una secuencia de ácido nucleico […]