DOMINIOS PROTEICOS DE AFINIDAD A COLINA PARA MEJORAR LA EXPRESION, INMOVILIZACION Y PURIFICACION DE POLIPETIDOS.
Son objetos de la presente invención una secuencia polipeptídica derivada de la del módulo de unión a colina de la amidasa LytA de Streptococcus pneumoniae,
así como un conjunto de vectores de expresión destinados a la obtención, de fusiones traduccionales a la secuencias codificante de dicho polipéptido en bacterias y en levaduras. Son también objetos de invención diversas mejoras estructurales y del procedimiento de purificación, que permiten mejorar la expresión y estabilidad de las fusiones, además de minimizar interferencias con el polipéptido fusionado y optimizar la separación proteolítica del citado módulo. Como ejemplos de realización se describe la construcción de un conjunto de vectores plasmídicos con las señales operativas necesarias para la expresión en bacterias o en levaduras de una de estas secuencias, y que permiten su fusión a genes heterólogos para obtener proteínas híbridas que pueden inmovilizarse en una variedad de soportes, y eventualmente purificarse mediante cromatografía de afinidad
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P200700281.
Solicitante: UNIVERSIDAD MIGUEL HERNANDEZ.
Nacionalidad solicitante: España.
Provincia: ALICANTE.
Inventor/es: CEBOLLA RAMIREZ,ANGEL, SANZ MORALES, JESUS MIGUEL, AREVALO RODRIGUEZ,MIGUEL, MAESTRO GARCIA-DONAS,BEATRIZ, HERNANDEZ ROCAMORA,VICTOR MANUEL.
Fecha de Solicitud: 29 de Enero de 2007.
Fecha de Publicación: .
Fecha de Concesión: 22 de Febrero de 2010.
Clasificación Internacional de Patentes:
- C07K1/22 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 1/00 Procedimientos generales de preparación de péptidos. › Cromatografía de afinidad o técnicas análogas basadas en procesos de absorción selectiva.
Clasificación PCT:
- C07K1/22 C07K 1/00 […] › Cromatografía de afinidad o técnicas análogas basadas en procesos de absorción selectiva.
- C12N15/55 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Hidrolasas (3).
- C12N15/72 C12N 15/00 […] › Sistemas de expresión que utilizan secuencias reguladoras derivadas del operón lac.
- C12N15/81 C12N 15/00 […] › para levaduras.
- C12N9/00 C12N […] › Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas.
Fragmento de la descripción:
Dominios proteicos de afinidad a colina para mejorar la expresión, inmovilización y purificación de polipéptidos.
Sector de la técnica
La presente invención se refiere a la utilización de una secuencia polipeptídica que, fusionada a una proteína de interés, permite su inmovilización en determinados soportes mediante afinidad específica por los mismos, y su posterior purificación a partir de éstos en un sólo paso. Se describe también un conjunto de vectores plasmídicos con los elementos genéticos adecuados para facilitar la expresión recombinante de proteínas fusionadas a la mencionada secuencia polipeptídica, tanto en bacterias como en levaduras, así como los procedimientos necesarios para llevar a cabo los procesos de inmovilización y purificación citados. El sector de la técnica al que puede aplicarse esta invención comprende por tanto la producción, inmovilización y purificación de polipéptidos y proteínas de interés biotecnológico, ya sea con fines industriales, agronómicos, terapéuticos o de diagnóstico en biomedicina, o como herramienta de investigación y análisis en el ámbito de las ciencias de la vida.
Estado de la técnica
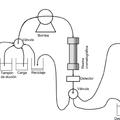
La inmovilización de enzimas en soportes sólidos, así como la purificación de proteínas en un sólo paso cromatográfico son dos procesos de esencial interés en Biotecnología que muchas veces comparten un procedimiento previo común: la fusión de la proteína de interés con un polipéptido o "tag" de afinidad que posibilita una interacción fuerte con un soporte sólido (Uhlén, M. et al (1992). Curr. Op. Biotechnol. 3, 363-369; Waugh. (2005). Trends Biotechnol. 23, 316-320). La adición a dicho soporte de un extracto celular de un organismo en el que se ha producido la expresión de la proteína híbrida posibilita la unión específica de la fusión al soporte, y la eliminación del resto de proteínas mediante el lavado exhaustivo del mismo. En este punto puede optarse, bien por utilizar la actividad enzimática así retenida, bien por eluir de manera selectiva la proteína híbrida mediante la adición de un ligando competidor (base de la cromatografia de afinidad), con el resultado de un alto grado de pureza en la preparación final de la proteína de interés.
La inmovilización de una enzima en una fase sólida facilita su recuperación y reutilización en un reactor (Trevan, M.D. et al (1990). En: Biotecnología Básica. Ed. Acribia, Zaragoza, España.). Además, las interacciones que el soporte mantiene con la enzima y su capacidad para resguardarla del disolvente pueden incrementar la estabilidad de la misma, y por tanto su rendimiento. Dentro de los métodos de inmovilización que se utilizan, los procedimientos de unión no covalente al soporte presentan varias ventajas frente a los procedimientos de unión covalente. En el primer caso, se pueden utilizar soportes muy variados que no es necesario activar, reduciéndose al mínimo las posibilidades de distorsión estructural de la proteína al no modificarla covalentemente. Además, la unión no covalente es en general un proceso reversible, permitiendo la regeneración de los soportes, de los cuales existe una amplia gama disponible comercialmente como puede comprobar cualquier experto en la materia.
En cuanto a la cromatografía de afinidad, es obvio que la reducción en el número de pasos de purificación de una proteína se traduce no sólo en un mayor rendimiento, sino además en un incremento de la pureza y un estimable ahorro de tiempo y esfuerzo.
Los módulos de unión a colina (ChBM, "choline-binding modules") constituyen una familia de polipéptidos que forman parte de las denominadas proteínas de unión a colina (ChBP, "choline-binding proteins"), presentes en una variedad de microorganismos (Swiatlo, E. et al (2004). En: The Pneumococcus. American Society for Microbiology Press, Washington DC). Los ChBMs constan a su vez de la repetición de secuencias muy conservadas de aproximadamente 20 aminoácidos (ChBR o "choline-binding repeats"; código Pfam PF01473: http://www.sanger.ac.uk//cgi-bin/Pfam/getacc?PF01473.), que forman estructuras del tipo bucle-horquilla-ß (Fernández-Tornero, C. et al (2001). Nat. Struct. Biol. 8, 1020-1024.). Dos ChBRs consecutivas configuran un sitio de unión a colina. La afinidad de los ChBMs por colina y análogos estructurales de la misma (Sanz, J.M. et al. (1988). FEBS Lett. 232, 308-312.), ha permitido poner a punto un sistema eficaz de purificación de proteínas de fusión con algunos de estos ChBMs (Sánchez-Puelles, J.M. et al. (1992) Eur. J. Biochem. 203, 153-159.). Básicamente, el procedimiento consiste en aplicar un extracto celular conteniendo la proteína de fusión sobre un soporte derivatizado con aminas terciarias o cuaternarias. La proteína así inmovilizada mantiene su funcionalidad, y puede ser eluída fácilmente mediante la adición de un ligando competidor, como la colina. El procedimiento así descrito es la base de una patente anterior (ES 2 032 717).
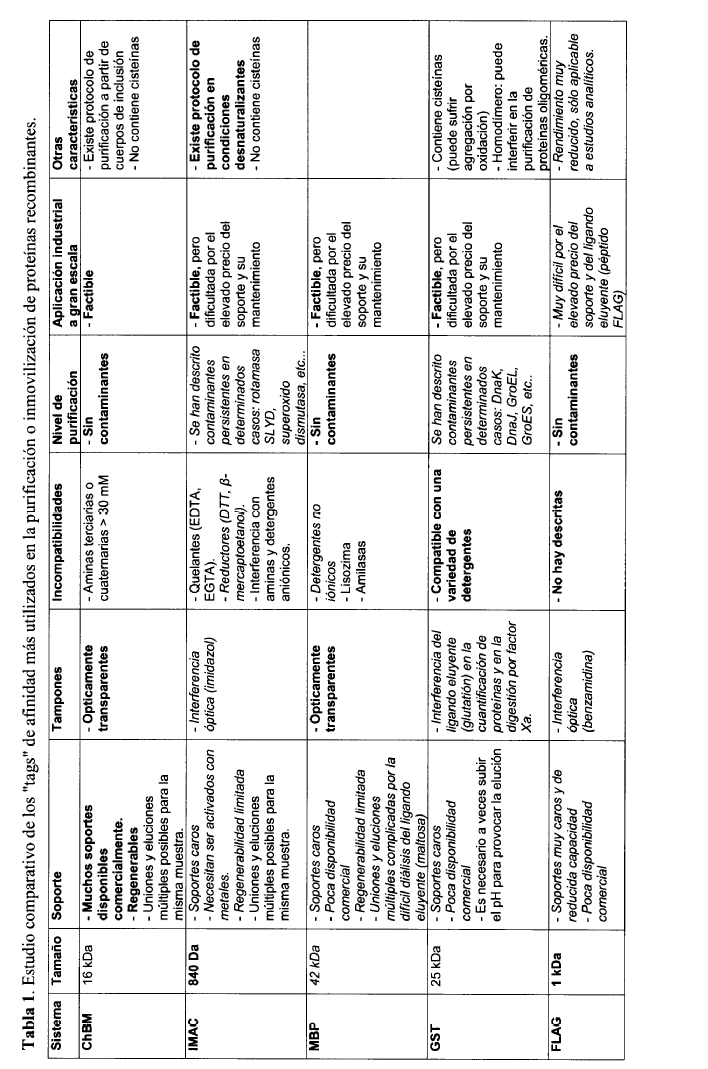
La Tabla 1 muestra un estudio comparativo de las características de los "tags" de afinidad más comúnmente utilizados junto con el sistema de los módulos de unión a colina (Sánchez-Puelles, J.M. et al. (1992) Eur. J. Biochem. 203, 153-159; Lichty, J.J. et al (2005) Protein Expression and Purification 41, 98-105; Waugh, D.S. (2005) Trends Biotechnol. 23, 316-320). Según se desprende del estudio, el procedimiento objeto de esta patente posee una serie de ventajas específicas que lo hace altamente competitivo, y que justifica su explotación para convertirse en un procedimiento extendido de inmovilización y purificación de proteínas. Fundamentalmente, el sistema es altamente específico, puede utilizar una gran variedad de soportes ya disponibles comercialmente (resinas, papel, placas multipocillo, etc...), muestra muy pocas incompatibilidades, y posee todas las atribuciones necesarias para pensar en un escalado eficiente para su empleo en biorreactores a nivel industrial.
Ámbito de la invención
La presente invención es útil para la expresión, inmovilización y purificación de proteínas de interés mediante fusión a las secuencias polipeptídicas descritas. Por tanto, puede servir para elaborar productos de utilidad para industrias de reactivos y herramientas de investigación en ciencias de la vida. Además, se puede aplicar a la industria química para la purificación de enzimas útiles, para la construcción de biorreactores enzimáticos para síntesis o análisis químicos, o procesos que requieran biotransformación, incluso dentro del ámbito agroalimentario. También es igualmente aplicable a la industria farmacéutica y sanitaria en general, para producir y purificar proteínas recombinantes aplicables al diagnóstico y terapias, así como para la elaboración de matrices de proteínas inmovilizadas de aplicación en proteómica.
(Tabla pasa a página siguiente)

Descripción detallada de la invención
El objeto de la presente invención comprende la inmovilización por unión no covalente y la purificación de proteínas recombinantes basándose en su fusión a unas secuencias polipeptídicas derivadas de la del dominio de unión a colina de la amidasa lítica LytA de S. pneumoniae (C-LytA), mediante sistemas genéticos que permiten su expresión en bacterias y en levaduras. La presente invención describe mejoras del método general descrito en una patente anterior (ES 2 032 717), ya que define el uso de una región concreta del dominio C-LytA y abre un abanico de posibilidades de utilización de este sistema en condiciones no estándar de temperatura y pH.
La presente invención comprende el uso de una versión recortada de una secuencia de DNA (SEQ ID Nº 1, codificante de C-LytA, SEQ ID Nº 2), así como de una variante mutante de la misma, que codifican polipéptidos más pequeños y con niveles de expresión superiores a los del polipéptido original C-LytA completo, manteniendo o mejorando sus propiedades para la inmovilización y purificación de polipéptidos recombinantes de fusión en soportes sólidos derivatizados con aminas terciarias y cuaternarias.
El polipéptido recortado derivado de C-LytA, LYTAG2 (SEQ ID Nº 3) posee 131 aminoácidos y un peso molecular de 14.62 kDa. Los aminoácidos 1-104...
Reivindicaciones:
1. Secuencia nucleotídica aislada, derivada de la SEQ1 ID Nº 1, que comprende una secuencia codificante de un polipéptido derivado del dominio de afinidad a colina de la proteína LytA de S. pneumoniae, caracterizado porque:
- (a) tiene afinidad por soportes sólidos con aminas terciarias o cuaternarias en su superficie.
- (b) la interacción con los soportes puede interrumpirse por adición de colina eq. solución a una concentración superior a 30 mM,
- (c) tiene un máximo de 162 aminoácidos, y un máximo de 5 de las repeticiones de unión a colina (ChBRs) presentes en el polipéptido de la secuencia SEQ1 ID Nº 2:- codificado en la secuencia SEQ1 ID Nº 1.
- (d) fusionado a otro polipéptido puede permitir su purificación por afinidad a 10 soportes descritos en (a) en condiciones que incluyen la (b).
- (e) codifica un polipéptido cuyos niveles de expresión, solubilidad y/o estabilidad, y afinidad por los soportes descritos en (a) en condiciones extremas son superiores:. a los de la secuencia SEQ ID Nº 2 completa.
2. Secuencia nucleotídica según la reivindicación 1 caracterizada porque, fusionada a la: secuencia codificante de otro polipéptido, permite la expresión de éste como un, polipéptido híbrido, a un nivel superior al del polipéptido híbrido resultante de una, fusión equivalente con la secuencia codificante de SEQ ID Nº 2 completa.
3. Secuencia nucleotídica según las reivindicaciones 1 y 2 caracterizada porque contiene una región, añadida a uno de sus extremos, y que codifica para una secuencia polipeptídica con estructura en hélice a.
4. Secuencia nucleotídica según las reivindicaciones 1 a 3 caracterizada porque comprende una secuencia codificante del polipéptido de secuencia SEQ ID Nº 3.
5. Mutantes de la secuencia nucleotídica que codifica el polipéptido SEQ ID Nº 2, y que se caracterizan porque:
- (a) mejoran las propiedades del polipéptido SEQ ID Nº 2 descritas en la reivindicación 1, a temperaturas elevadas, y/o
- (b) mejoran las propiedades del polipéptido SEQ ID Nº 2 descritas en la reivindicación 1, a valores extremos de pH.
6. Secuencia nucleotídica de las reivindicación 5 con mutaciones que determinan una, algunas o todas las sustituciones siguientes con respecto a la secuencia SEQ ID Nº 2: Asn5l a Lys, Ser52 a Lys, Thr85 a Lys y Thr108 a Lys.
7. Secuencia nucleotídica de la reivindicación 6 caracterizada porque comprende una secuencia codificante del polipéptido de secuencia SEQ ID Nº 51.
8. Secuencia nucleotídica derivada de la de la reivindicación 4 caracterizada porque comprende una secuencia codificante del polipéptido de secuencia SEQ ID Nº 4.
9. Secuencia nucleotídica de las reivindicaciones 1 a 3 que es al menos un 50% idéntica a las secuencias que codifican los polipéptidos SEQ ID Nº 3 o SEQ ID Nº 4.
10. Secuencia nucleotídica derivada de la de la reivindicación 4, caracterizada porque incorpora una región codificante para una secuencia de aminoácidos reconocible por una endopeptidasa de corte específico, y que se localiza en uno de los extremos, el correspondiente a la secuencia codificante del polipéptido con estructura en hélice a.
11. Vectores de DNA que contienen una de las secuencias de las reivindicaciones 1 a 10.
12. Vectores según la reivindicación 11, caracterizados porque la secuencia de las reivindicaciones 1 a 10 se encuentra situada bajo el control de un promotor de la transcripción, y adyacente a una región con dianas para enzimas de restricción, o de un sitio de recombinación específica, que permite la fusión traduccional de dicha secuencia a la del polipéptido que se quiere inmovilizar o purificar, a sus extremos amino o carboxilo.
13. Vectores según la reivindicación 12, caracterizados porque el promotor es activo en células de organismos procariotas o eucarióticas como levaduras, hongos filamentosos, plantas, o animales.
14. Vectores según la reivindicación 13, caracterizados porque el promotor es el promotor Pm de Pseudomonas putida, o derivados del mismo.
15. Vectores según la reivindicación 14, caracterizados porque el promotor es derivado de la región promotora localizada entre los genes GAL12 y GAL10 de S. cerevisiae.
16. Células transformadas que contengan vectores según las reivindicaciones 11 a 15, en forma episómica o integrados en DNA genómico de dichas células.
17. Procedimiento de inmovilización de polipéptidos por fusión a una secuencia:. polipeptídica según las reivindicaciones 1 a 10 que comprende:
- (a) el uso de un sustrato sólido con la superficie cubierta por aminas terciarias o cuaternarias al que se une por afinidad el polipéptido de las reivindicaciones 1 a 10.
- (b) una solución donde está solubilizado el polipéptido de fusión al polipéptido anterior, que puede tener una concentración de 0 mM a 1500 mM de NaCl, hasta 3 mM de colina, un detergente como SDS (hasta 2 mM) o Triton X-100 (hasta 1.5%) y un pH de 3 a 9.
- (c) una solución de lavado con una concentración de NaCl igual o más concentrada que en (b) y que puede aplicarse a la resina con el polipéptido de fusión a una temperatura de 4ºC a 95ºC.
- (d) una solución para eluir eventualmente la proteína de fusión que contiene colina a una concentración superior a 30 mM.
18. Procedimiento según la reivindicación 17 donde la amina terciaria o cuaternaria es dietilaminoetanol (DEAE), trimetilamonio (QAE), colina, o cualquier compuesto similar.
19. Procedimiento según las reivindicaciones 17 y 18 donde el sustrato es un polímero del tipo de las agarosas, los dextranos, las celulosas, las siliconas, el metacrilato, los poliestirenos, u otros tipos de polímeros que puedan tener en su superficie aminas terciarias o cuaternarias de las que unen la secuencia polipeptídica de las reivindicaciones de 1 a 10.
20. Procedimiento de sobre-expresión y purificación de polipéptidos utilizando los vectores de las reivindicaciones 11 a 15, la secuencia heteróloga fusionada a la secuencia del tipo de las reivindicaciones 1 a 10, y el procedimiento de inmovilización de proteínas de fusión de las reivindicaciones 17 a 19.
21. Uso de la secuencia de las reivindicaciones 1 a 10 y del procedimiento de las reivindicaciones 17 a 20 para la inmovilización y purificación de polipéptidos de uso terapéutico, diagnóstico y en cualquier tipo de industria donde se necesiten proteínas recombinantes.
Patentes similares o relacionadas:
 Purificación de proteínas, del 29 de Julio de 2020, de UCB Biopharma SRL: Un proceso para la purificación de una proteína de interés a partir de una mezcla que comprende las etapas de
a) en un ciclo de cromatografía operativo, […]
Purificación de proteínas, del 29 de Julio de 2020, de UCB Biopharma SRL: Un proceso para la purificación de una proteína de interés a partir de una mezcla que comprende las etapas de
a) en un ciclo de cromatografía operativo, […]
Membranas para cromatografía formadas por reacciones de polimerización clic de tiol-eno o tiol-ino, del 10 de Junio de 2020, de Merck Millipore Ltd: Un material compuesto, que comprende: un miembro de soporte, que comprende una pluralidad de poros que se extienden a través del miembro de […]
Un procedimiento de cromatografía de reparto débil, del 6 de Mayo de 2020, de WYETH LLC: Un procedimiento de recuperación de un producto purificado de un fluido de carga que incluye una o más impurezas, que comprende las etapas de: hacer pasar el fluido […]
Procedimiento de purificación de polipéptidos, del 22 de Abril de 2020, de CSL LIMITED: Procedimiento de purificación de un polipéptido de interés por cromatografía de intercambio catiónico en el que un compuesto químico se añade en una concentración de al menos […]
Etiqueta de epítopo y método de detección, captura y/o purificación de polipéptidos etiquetados, del 15 de Abril de 2020, de ChromoTek GmbH: Péptido epítopo aislado que tiene de 12 a 25 aminoácidos, en donde la secuencia de aminoácidos comprende una secuencia según se define en SEQ ID NO: 32 (X1X2RX4X5AX7SX9WX11X12), […]
Matriz de cromografía de afinidad, del 25 de Marzo de 2020, de LABORATOIRE FRANCAIS DU FRACTIONNEMENT ET DES BIOTECHNOLOGIES SOCIETE ANONYME: Matriz de cromatografía de afinidad, en forma de gel, que comprende unas partículas de polímero sobre las cuales se injerta al menos un oligosacárido […]
Sistema de tampón para purificación de proteína, del 26 de Febrero de 2020, de GLAXOSMITHKLINE LLC: Un sistema de cromatografía que comprende: (i) un sistema de tampón de múltiples componentes libre de cloruro de sodio para la purificación de una […]
Proteína mutante, del 19 de Febrero de 2020, de Cytiva BioProcess R&D AB: Una proteína de unión a inmunoglobulina que se une a regiones de una molécula de inmunoglobulina distintas de las regiones determinantes de […]