DESCRIPTORES DE OBJETOS TRIDIMENSIONALES, USOS DE LOS MISMOS Y METODOS PARA GENERARLOS.
Un método informatizado para generar un descriptor de un objeto tridimensional,
seleccionándose dicho objeto tridimensional entre el grupo constituido por
1) una superficie obtenida de un bolsillo de biomolécula,
2) una configuración tridimensional de una especie molecular, y
3) una superficie catalítica,
estando dicho objeto tridimensional representado por una serie de coordenadas, siendo dicho descriptor una sucesión de valores de interacción Vi, comprendiendo dicho método la etapa de atribuir una o más propiedades y un valor Vb para cada una de dicha una o más propiedades en coordenadas seleccionadas dentro de dicha serie de coordenadas de dicho objeto tridimensional, caracterizado porque las siguientes etapas se realizan para cada una de una serie de una o más jaulas, implementado dicha serie de una o más jaulas un entorno artificial, teniendo dicha una o más jaulas cada una, una forma tridimensional en cuya superficie se sitúan una serie de puntos, las mimas una o más propiedades atribuidas a dicho objeto tridimensional y atribuyéndose un valor Vc para cada una de dichas una o más propiedades a cada uno de dichos puntos:
i) encerrar completamente dicho objeto tridimensional en dicha jaula,
ii) para cada propiedad, mientras se mantiene a dicho objeto tridimensional completamente encerrado en dicha jaula modificando una o más dimensiones de dicha jaula, determinar el valor de interacción más bajo posible Vi o el valor de interacción más alto posible Vi o un valor de interacción Vi promediado en todas las orientaciones resultantes de la interacción entre dichas coordenadas seleccionadas de dicho objeto tridimensional y dichos puntos de dicha jaula muestreando en todo el espacio rotacional la orientación relativa entre el objeto tridimensional y dicha jaula, y
iii) asignar cada uno de los valores de interacción más bajos posibles Vi o valores de interacción más altos posibles Vi o valor de interacción Vi promediado sobre todas las orientaciones obtenidos, a una posición distinta en dicho descriptor
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E06011342.
Solicitante: SILICOS NV.
Nacionalidad solicitante: Bélgica.
Dirección: WETENSCHAPSPARK 7,3590 DIEPENBEEK.
Inventor/es: DE WINTER,HANS LOUIS JOS, LANGENAEKER,WILFRIED GERT ROGER.
Fecha de Publicación: .
Fecha Solicitud PCT: 1 de Junio de 2006.
Fecha Concesión Europea: 7 de Octubre de 2009.
Clasificación Internacional de Patentes:
- G06F19/00D
Clasificación PCT:
- G06F19/00
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
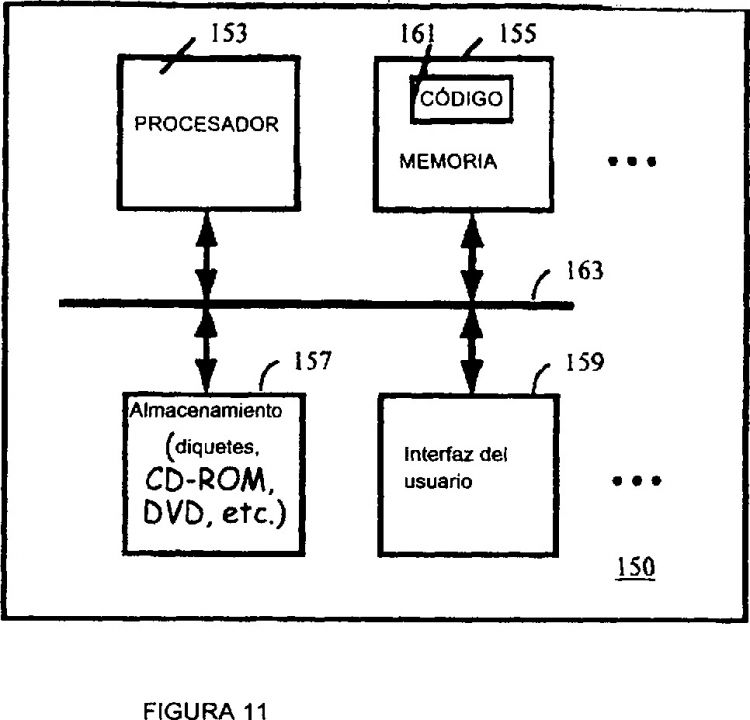
Fragmento de la descripción:
Descriptores de objetos tridimensionales, usos de los mismos y métodos para generarlos.
Campo de la invención
La invención se refiere al campo de la modelización molecular para el descubrimiento de fármacos y a la aplicación de algoritmos de cribado virtual para el descubrimiento químico y en particular al uso de sistemas basados en la informática. En particular, se describe un método para generar descriptores, por ejemplo para transformar cualquier tipo de objetos tridimensionales con propiedades asociadas, tales como especies moleculares con sus propiedades atómicas asociadas, en sucesiones de números reales. Una vez obtenidos, estos descriptores pueden usarse para entender y obtener relaciones de estructura/actividad, cribado virtual de bases de datos moleculares, y síntesis virtual de moléculas con propiedades predefinidas así como conducir a la síntesis de dichos compuestos. Además de su aplicación en moléculas pequeñas, el método también puede aplicarse a los bolsillos de unión de proteínas u otras biomoléculas, por ejemplo convirtiendo este bolsillo en su imagen especular.
Antecedentes de la invención
Tradicionalmente, el diseño de nuevas especies moleculares (por ejemplo fármacos) ha sido esencialmente un proceso de ensayo y error a pesar de los tremendos esfuerzos dedicados por los grupos de investigación farmacéuticos y académicos. En un intento de contrarrestar los costes, que aumentan rápidamente, asociados con el descubrimiento de nuevas medicinas, se realizan nuevos enfoques informatizados. Los enfoques modernos al diseño molecular asistido por ordenador entran dentro de dos categorías generales. La primera incluye métodos basados en la estructura que utilizan la estructura tridimensional de un receptor unido a ligando. El segundo enfoque incluye métodos basados en ligando en los que se caracterizan las propiedades fisicoquímicas o estructurales de especies moleculares de ligando. Un ejemplo clásico de este concepto es un modelo de relación cuantitativa de estructura-actividad (QSAR). Las relaciones cuantitativas de estructura-actividad son relaciones matemáticas que unen estructuras químicas -representadas en forma de descriptores moleculares- y actividad farmacológica de manera cuantitativa para una serie de especies moleculares.
El cribado virtual es el proceso de cálculo mediante el cual se realiza una búsqueda, en bibliotecas de especies moleculares existentes o virtuales, de especies moleculares que cumplan criterios bien definidos. En general, el cribado virtual se aplica a la búsqueda de especies moleculares que pudieran ser activas contra algunas proteínas relacionadas con enfermedades, mientras que la actividad se obtiene de la interacción calculada entre la proteína y la especie molecular. La valoración de las especies moleculares se realiza usando funciones matemáticas bien definidas con objeto de priorizar estas especies moleculares para un análisis adicional. Típicamente, pueden distinguirse dos tendencias principales de cribado virtual.
La primera tendencia consiste en el enfoque basado en la estructura de proteínas en el que se usa el bolsillo de unión potencial de una proteína como función de referencia. La selección de bolsillos de unión potenciales sigue siendo un desafío principal en la industria farmacéutica. Una vez que se conoce la función de referencia, puede comenzarse el cribado de especies moleculares que tienen las propiedades deseadas con respecto a la unión a la proteína diana. Se han descritos varios métodos adecuados:
La segunda tendencia consiste en un enfoque basado en ligando, en el que se usan especies moleculares con afinidad conocida por una proteína diana o modelo de enfermedad, como función de referencia. Se obtiene un modelo a partir de estas especies moleculares de referencia y puede usarse para anotar otras especies moleculares con respecto a sus capacidades de unión potenciales. Se han descrito varios enfoques basados en ligando adecuados. Algunos son bidimensionales y otros son tridimensionales. Los métodos en 2D tienen la ventaja de aplicarse de forma muy eficaz para la búsqueda de bases de datos moleculares. La desventaja es que son bastante inespecíficos, lo que no es el caso para los algo más lentos métodos en 3D. Estas formas de cribado virtual pueden integrarse con resultados disponibles de cribado de alto rendimiento (HTS). A continuación se proporciona una visión general de una aplicación de cribado virtual basado en ligando típica, que se combina con cribado de alto rendimiento:
Dichas modernas plataformas de cribado de alto rendimiento requieren la implementación e integración de propiedades y algoritmos de cribado virtual eficaces y robustos.
Para ser adecuados para su uso en un contexto de cálculo, la información molecular debe traducirse a una forma adecuada, llamada generalmente un descriptor. La complejidad de los descriptores moleculares puede variar enormemente. Un ejemplo sencillo puede ser un descriptor clave estructural, que toma la forma de un indicador binario variable que codifica la presencia de ciertas características de subestructura o funcionales. Otros descriptores, tales como energías HOMO (Orbital Molecular más Alto Ocupado) y LUMO (Orbital Molecular más Bajo Ocupado), requieren cálculos semi-empíricos o de mecánica cuántica y, por lo tanto, requieren más tiempo para calcularlos. Los descriptores moleculares se clasifican a menudo de acuerdo con su dimensionalidad, que se refiere a la representación estructural a partir de la cual se obtienen los valores del descriptor. En general, pueden clasificarse los actuales descriptores moleculares como unidimensionales (en 1D), bidimensionales (en 2D), o tridimensionales (en 3D).
Los descriptores unidimensionales son un reflejo de las propiedades "en masa" de especies moleculares, como el peso molecular, el número de átomos, o la distribución molecular entre fases hidrófilas y lipófilas. Los descriptores unidimensionales se calculan generalmente con rapidez y pueden calcularse a partir de la composición molecular en solitario. Sin embargo, los descriptores unidimensionales carecen de cualquier información sobre la conectividad molecular entre los átomos y son, por lo tanto, de una precisión bastante limitada cuando se aplican a problemas de descubrimiento de fármacos y cribado virtual.
El cálculo de descriptores bidimensionales requiere conocimientos de la topología molecular, y comprende información sobre la presencia o ausencia de restos funcionales bien definidos, distancias topológicas entre átomos bien definidos, e información respecto a cadenas laterales y sistemas de anillos. Los descriptores bidimensionales han encontrado utilidad en análisis de similitud química y relaciones de estructura-actividad, y son útiles para complementar a descriptores tridimensionales. Los descriptores bidimensionales usados más ampliamente son huellas moleculares, índices de "estado E", y descriptores de hologramas QSAR.
Las huellas moleculares son esencialmente mapas de bits que constan de bits "on" y "off", donde cada posición a lo largo del mapa de bits se asigna a un fragmento molecular específico y bien definido. Si ese fragmento particular existe en las especies moleculares en consideración, entonces el bit correspondiente se pone en "on", en caso contrario se deja en "off". Existen dos métodos generales de generación de huellas en 2D. El primero, conocido como el método "resumido" ("hashed"), usa una serie de reglas para generar los fragmentos para la huella. El segundo método, conocido como el método "en clave" ("keyed"), requiere a priori definiciones subestructurales para todos los fragmentos que deben buscarse durante el...
Reivindicaciones:
1. Un método informatizado para generar un descriptor de un objeto tridimensional, seleccionándose dicho objeto tridimensional entre el grupo constituido por
estando dicho objeto tridimensional representado por una serie de coordenadas, siendo dicho descriptor una sucesión de valores de interacción Vi, comprendiendo dicho método la etapa de atribuir una o más propiedades y un valor Vb para cada una de dicha una o más propiedades en coordenadas seleccionadas dentro de dicha serie de coordenadas de dicho objeto tridimensional, caracterizado porque las siguientes etapas se realizan para cada una de una serie de una o más jaulas, implementado dicha serie de una o más jaulas un entorno artificial, teniendo dicha una o más jaulas cada una, una forma tridimensional en cuya superficie se sitúan una serie de puntos, las mimas una o más propiedades atribuidas a dicho objeto tridimensional y atribuyéndose un valor Vc para cada una de dichas una o más propiedades a cada uno de dichos puntos:
2. Un método de acuerdo con la reivindicación 1, caracterizado porque dicho valor Vc es +1, 0 o -1.
3. Un método de acuerdo con la reivindicación 2, caracterizado porque dicho valor Vc es +1 o -1.
4. Un método de acuerdo con cualquiera de las reivindicaciones 1 a 3, caracterizado porque la suma de los valores Vc para cada jaula es cero.
5. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque dicho valor de interacción Vi viene dado por la siguiente fórmula:

en la que nCoordenadas es el número total de dichas coordenadas seleccionadas en dicho objeto tridimensional, nPuntos el número total de dichos puntos, Vb el valor de la propiedad en la coordenada bésima, Vc el valor de la propiedad en el césimo punto de jaula, dbc la distancia entre la coordenada b y el punto de jaula c, y f(), g() y h() son funciones de transformación matemática.
6. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque para la minimización del valor de interacción Vi, las dimensiones de la jaula se seleccionan de modo que al menos dos posiciones en el objeto estén más cerca de 1 nm de la jaula.
7. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque dicha serie de puntos comprende entre cuatro y doce puntos.
8. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque al menos una de dicha jaulas es estereoespecífica.
9. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque dicha una o más jaulas se selecciona entre jaulas cuboideas en cuya superficie dicha serie de puntos son:
10. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque dicha una o más jaulas son cuatro o más jaulas.
11. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque cada valor Vb se normaliza antes de realizar la etapa ii).
12. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque al menos una de dicha una o más jaulas es cuboidea.
13. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque dicho objeto tridimensional es una configuración tridimensional de una especie molecular y en el que dichas coordenadas seleccionadas son posiciones atómicas seleccionadas.
14. Un método de acuerdo con cualquier reivindicación anterior, caracterizado porque dichas posiciones atómicas seleccionadas son todas las posiciones atómicas de dicha configuración tridimensional de dichas especies moleculares.
15. Un método de acuerdo con la reivindicación 5, caracterizado porque dicho objeto tridimensional es una configuración tridimensional de una especie molecular, n coordenadas es el número total de dichas posiciones atómicas seleccionadas en dicha configuración tridimensional de una especie molecular, Vb es el valor de la propiedad en la bésima posición atómica, dbc es la distancia entre el átomo b y el punto de jaula c.
16. Un método de acuerdo con cualquiera de las reivindicaciones 13 a 15, caracterizado porque dicha configuración tridimensional se determina mediante una simulación por ordenador, mediante uno o más medios de análisis de laboratorio o mediante una combinación de simulación por ordenador y uno o más medios de análisis de laboratorio.
17. Un método de acuerdo con cualquiera de las reivindicaciones 13 a 16, que comprende además la etapa de optimizar la conformación de dicha configuración tridimensional de dicha especie molecular después de la etapa i) y antes de la etapa iii) para minimizar el valor de interacción Vi calculado resultante de la interacción entre dicha configuración tridimensional de dicha especie molecular y dicha jaula.
18. Un método de acuerdo con cualquier reivindicación anterior, para su uso en el cribado virtual de bases de datos moleculares, síntesis virtual de moléculas con propiedades predefinidas, y en la comprensión y obtención de estructuras/relaciones de dichas especies moleculares.
19. Un método para evaluar la similitud entre un objeto tridimensional de referencia y un objeto tridimensional de ensayo, calculando la similitud entre los descriptores correspondientes generados mediante el método de cualquier reivindicación anterior.
20. Un método para generar una relación cuantitativa de estructura-actividad tridimensional 3D-QSAR de una serie de especies moleculares que comprende las etapas de:
21. Un producto de programa informático que comprende un código de software para implementar cualquiera de los métodos de las reivindicaciones 1 a 20, cuando se ejecuta en un sistema de cálculo.
22. Un soporte de datos legible por una máquina que almacena el programa informático de la reivindicación 21.
Patentes similares o relacionadas:
SELECCIÓN DE PACIENTES CON CÁNCER DE CABEZA Y CUELLO PARA TRATAMIENTO CON FÁRMACOS DIRIGIDOS A LA VÍA EGFR, del 22 de Noviembre de 2011, de Biodesix Inc: Un procedimiento para determinar si un paciente con carcinoma de células escamosas de cabeza y cuello (CCECC) tiene probabilidades de beneficiarse del tratamiento con […]
 METODOS DE ESCRUTINIO EMPLEANDO UN MODELO ESTRUCTURAL DE FIH, del 20 de Abril de 2010, de ISIS INNOVATION LIMITED: Un método para identificar, explorar, caracterizar o diseñar una identidad química que se une a un factor inhibidor de HIF (FIH), comprendiendo dicho […]
METODOS DE ESCRUTINIO EMPLEANDO UN MODELO ESTRUCTURAL DE FIH, del 20 de Abril de 2010, de ISIS INNOVATION LIMITED: Un método para identificar, explorar, caracterizar o diseñar una identidad química que se une a un factor inhibidor de HIF (FIH), comprendiendo dicho […]
Sistemas y métodos para tratar, diagnosticar y predecir la aparición de una afección médica, del 15 de Julio de 2020, de Fundação D. Anna Sommer Champalimaud E Dr. Carlos Montez Champalimaud: Aparato para evaluar si es probable que un paciente tenga un estadio patológico favorable de cáncer de próstata, el aparato comprende: (a) un dispositivo de obtención […]
Dispositivo de procesamiento de datos para el procesamiento de valores de medición, del 22 de Abril de 2020, de F. HOFFMANN-LA ROCHE AG: Dispositivo de procesamiento de datos para el procesamiento de valores de medición de un equipo de medición médico, con una unidad de entrada , una […]
Sistema de análisis apuntador de acontecimientos y de gestión de medicación, del 30 de Octubre de 2019, de CAREFUSION 303, INC: Un método para reducir el riesgo de errores de medicación, el método comprende las etapas de: recibir en un dispositivo móvil un primer valor de un parámetro […]
Sistema y método de visualización en 2D y 3D para la inspección de un tubo de horno, del 11 de Septiembre de 2019, de Quest Integrity USA, LLC: Un sistema para la visualización de datos de inspección recogidos de un horno con una geometría física especificada, en donde dicho horno comprende […]
 Reconocimiento anatómico y análisis dimensional para asistir en la cirugía de mama, del 25 de Julio de 2019, de ALLERGAN, INC.: Método implementado por ordenador de determinación de mediciones de la mama de manera automática a partir de una representación tridimensional (3-D) de la parte superior […]
Reconocimiento anatómico y análisis dimensional para asistir en la cirugía de mama, del 25 de Julio de 2019, de ALLERGAN, INC.: Método implementado por ordenador de determinación de mediciones de la mama de manera automática a partir de una representación tridimensional (3-D) de la parte superior […]
Sistemas y métodos para tratar, diagnosticar y predecir la aparición de una afección médica, del 24 de Julio de 2019, de Fundação D. Anna Sommer Champalimaud E Dr. Carlos Montez Champalimaud: Un aparato para evaluar un riesgo de fallo clínico en un paciente después de que el paciente se haya sometido a una prostatectomía radical, y el aparato […]