CONJUNTO DE CEBADORES, PROCEDIMIENTO Y KIT PARA EL DIAGNÓSTICO Y LA TIPIFICACIÓN DE LA BACTERIA PATÓGENA CAUSANTE DE LA TUBERCULOSIS DEL OLIVO, PSEUDOMONAS SAVASTANOI PV. SAVASTANOI.
Conjunto de cebadores, procedimiento y kit para el diagnóstico y la tipificación de la bacteria patógena causante de la tuberculosis del olivo,
Pseudomonas savastanoi pv. savastanoi.
La presente invención permite el diagnóstico (amplificación por PCR de una muestra de ADN usando un conjunto de cebadores reivindicado, digestión mediante una enzima de restricción de los productos resultantes, y análisis de los fragmentos resultantes mediante electroforesis) y la tipificación (amplificaciones por PCR de una muestra de ADN diagnosticada usando los conjuntos de cebadores reivindicados, y análisis de los productos finales mediante electroforesis) de Pseudomonas savastanoi pv. savastanoi, agente causante de la tuberculosis del.olivo. Las principales ventajas de la presente invención son: el uso de cebadores no fluorescentes, lo que evita la utilización de equipamiento especializado y de alto coste, así como más estables y eficaces; y la obtención de resultados fácilmente analizables electroforesis en gel y consecuentemente diagnósticos más específicos.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201000535.
Solicitante: UNIVERSIDAD DE MALAGA.
Nacionalidad solicitante: España.
Inventor/es: RAMOS RODRIGUEZ,CAYO JUAN, LÓPEZ GONZÁLEZ,María Milagros, MATAS CASADO,ISABEL, PEREZ MARTINEZ,ISABEL, PENYALVER NAVARRO,RAMON.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
Conjunto de cebadores, procedimiento y kit para el diagnóstico y la tipificación de la bacteria patógena causante de la tuberculosis del olivo, Pseudomonas savastanoi pv. savastanoi.
Sector de la técnica La presente invención se refiere a un conjunto de cebadores, procedimiento y kit para el diagnóstico y tipificación de bacterias patógenas del olivo, y más concretamente para el diagnóstico y tipificación de Pseudomonas savastanoi pv. savastanoi, agente causante de la tuberculosis del olivo.
Estado de la técnica
En paralelo a la generación de un mercado creciente de consumidores de aceite de oliva en todo el mundo, la superficie dedicada a este cultivo también aumenta, no únicamente en los países mediterráneos donde el cultivo del olivo juega desde hace siglos un papel económico, nutricional, cultural, social y medioambiental, sino también en regiones tan alejadas del origen del cultivo como California, Argentina, Australia (Hall et al., 2004) o Nueva Zelanda (Taylor et al., 2001) .
La Unión Europea (UE) es el principal productor mundial de aceite de oliva, ya que supera el 75% de la producción mundial. Dentro de la UE, España, Italia y Grecia generan más del 98% de la producción. La superficie del olivar en estos tres países representa alrededor del 49% de la superficie mundial y suministra más del 77% de la producción total de aceite, demostrando su alto nivel de rendimiento (FAO, 2005) . Datos de la campaña 2007 indican que la superficie olivarera total en España es de 2.456.719 hectáreas dedicadas principalmente a la producción de aceite de oliva, de las cuales en este mismo año, Andalucía cultiva aproximadamente un 61% de la superficie total con un porcentaje del 82% de la producción total de aceite de oliva (Datos del informe nº 4 del Ministerio de Agricultura, Pesca y Alimentación, 2007) .
Con el objetivo fundamental de mantener y potenciar su posición en el mercado mundial, la política actual de la UE se dirige a fomentar la producción de un producto de gran calidad que redunde en beneficio de los agricultores, transformadores, comerciantes y consumidores. Entre otras medidas, cabe destacar la elaboración de una lista de organismos nocivos y enfermedades que afectan a la calidad del aceite manera significativa (EU-Directive 92/34 reflejada en el ordenamiento jurídico español por el Real Decreto 1678/1999, de 29 de octubre: BOE 22238, 18 de Noviembre de 1999) . En consecuencia, la comercialización de plantones de olivo requiere actualmente la certificar la ausencia de los patógenos incluidos en esta lista.
Pseudomonas savastanoi pv. savastanoi (Pss) , único patógeno bacteriano del olivar incluido en la Directiva de la UE antes mencionada, es el agente causal de la tuberculosis del olivo. Los árboles afectados muestran tumores (hiperplasia del tejido vegetal conocida con el nombre de verrugas o agallas) que llegan a alcanzar varios centímetros de diámetro en troncos, ramas, tallos y brotes. En general, los árboles enfermos muestran menor vigor y reducción del crecimiento; cuando el ataque es muy intenso, los árboles terminan siendo improductivos (Trapero y Blanco, 1996) . En España, Cuesta y Delgado (1995) han evaluado las pérdidas causadas por tuberculosis en 37.822 Tm anuales. Datos recientes procedentes del Instituto Valenciano de Investigaciones Agrarias (IVIA) indican que la producción de olivos enfermos de tuberculosis disminuye con respecto a la de los olivos sanos (Quesada et al., 2004) .
Hasta la fecha, y debido a la ausencia de métodos eficaces de control de la tuberculosis se hace necesario establecer una estrategia de lucha preventiva, resultando para ello indispensable disponer de métodos de diagnóstico temprano que permitan determinar la presencia del agente patógeno, incluso en material vegetal asintomático. Por otro lado, la disponibilidad de métodos moleculares de tipificación de agentes patógenos, permitiría llevar a cabo estudios epidemiológicos, los cuales hasta la fecha son escasos para muchos de ellos, como por ejemplo P. savastanoi.
La formación de tumores en plantas de olivo y adelfa depende de la producción y secreción de las fitohormonas ácido indol-3-acético (IAA) y citoquininas (Cks) por parte de la bacteria (Smidt y Kosuge, 1978; Comai y Kosuge, 1980; Comai y Kosuge, 1982; Surico et al., 1985a; MacDonald et al., 1986; Iacobellis et al., 1994) . La producción de IAA en cepas de P. savastanoi ocurre mediante la conversión del triptófano en indolacetamida (IAM) , llevada a cabo por la enzima triptófano monooxigenasa (producto del gen iaaM) y, posteriormente, por la conversión del IAM en IAA, mediante la actividad de la indolacetamida hidrolasa (producto del gen iaaH) (Magie et al., 1963; Kosuge et al., 1966) . Además, P. savastanoi es capaz de transformar el IAA en el conjugado IAA-lisina mediante la actividad indo 1-3-acetil-L-lisina sintetasa, cuya actividad está codificada por el gen iaaL (Hutzinger y Kosuge, 1968; Glass y Kosuge, 1986a) . Estudios de expresión génica en P. savastanoi, demostraron que la expresión de los genes iaaM e iaaH es constitutiva e independiente de la concentración de triptófano presente en el medio (Gaffney et al., 1990) . Por lo tanto, el aumento de la concentración intracelular de triptófano conduce a una sobreproducción de IAA, que provoca una mayor severidad en los síntomas ocasionados por la bacteria (Kuo y Kosuge, 1969; Smidt y Kosuge, 1978) . Por el contrario, mutantes defectivos en la producción de IAA son incapaces de formar tumores completos en la planta hospedadora (Iacobellis et al., 1994) .
La aplicación de técnicas de PCR en el estudio de patógenos vegetales ha permitido la detección y caracterización de bacterias fitopatógenas con gran fiabilidad y relativa sencillez, destacando su sensibilidad y especificidad alcanzadas. La primera aplicación de PCR al diagnóstico de P. savastanoi pv. savastanoi fue desarrollada por Penyalver y colaboradores (2000) . Este método se basa en el enriquecimiento previo de la población de este patógeno en medios semiselectivos de cultivo, seguido de una PCR en la que se utilizan cebadores específicos del gen iaaL que amplifican un fragmento interno de 454 pb. Para ello, se utilizan dos oligonucleótidos, denominados IAAL-F e IAAL-R (Penyalver et al., 2000) . La sensibilidad de este método permite la detección de hasta 10 UFC/ml en material vegetal asintomático proveniente de plantas con tumores. Otras técnicas moleculares desarrolladas posteriormente, como la nested-PCR, también permite detectar a este patógeno en material asintomático de olivo (Bertolini et al., 2003a) . Además, se ha puesto a punto una multiplex nested RT-PCR que permite la detección simultánea, y en una sola reacción, de P. savastanoi pv. savastanoi y de cuatro virus que afectan al olivo (Bertolini et al., 2003b) .
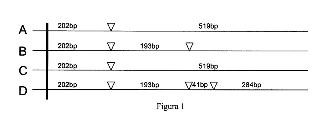
Recientemente se ha descrito que las cepas de P. savastanoi pv. savastanoi contienen dos alelos diferentes del gen iaaL, siendo la secuencia de uno de ellos, denominado alelo iaaL-Psn, 100% idéntica a la del gen ortólogo en la bacteria patógena de adelfa (Nerium oleander) P. savastanoi pv. nerii. La secuencia del otro alelo, denominado alelo iaaL-Psv, es 93% idéntica a la de su ortólogo (iaaL-Pto) en la bacteria patógena de tomate P. syringae pv. syringae DC3000. Además, se ha descrito que la secuencia del alelo iaaL-Psv de P. savastanoi pv. savastanoi contiene un número variable de repeticiones en tándem del trinucleótido TAC (Matas et al., 2009) . Las repeticiones TAC se localizan dentro del fragmento interno de 454 pb utilizado por Penyalver y colaboradores (2000) para la identificación de esta bacteria patógena mediante PCR. Se ha determinado que el número de repeticiones TAC dentro de la secuencia del alelo iaaL-Psv de P. savastanoi pv. savastanoi varía de una cepa a otra entre 3 y 15 veces. De entre las 30 cepas analizadas, el mayor número de repeticiones detectadas (entre 8 y 15) corresponden a cepas aisladas en la Península Ibérica (España y Portugal) (Matas et al., 2009) .
Las variaciones en la secuencia de nucleótidos del fragmento de 454 pb procedente de los alelos iaaL-Pto, iaaL-Psn e iaaL-Psv, permiten su diferenciación mediante PCR-RFLP (Restriction Fragment Length Polymorphism) utilizando la enzima HaeIII. Para ello, la visualización de los productos de restricción debe llevarse a cabo en geles de agarosa al 3% (Matas et al., 2009) , en lugar de en gel de agarosa al 2%, tal y como indicaban Penaylver y colaboradores (2000) .
El número de repeticiones TAC de los alelos iaaL-Pto, iaaL-Psn e iaaL-Psv puede determinarse mediante electroforesis...
Reivindicaciones:
1. Conjunto de cebadores para el diagnóstico de Pseudomonas savastanoi pv. savastanoi caracterizado porque al menos dos de los cebadores de dicho conjunto presentan secuencias idénticas a las secuencias mostradas en las SEQ ID NO1 y SEQ ID NO 2.
2. Conjunto de cebadores para el diagnóstico de Pseudomonas savastanoi pv. savastanoi según la reivindicación anterior caracterizado porque dicho conjunto está formado por un primer cebador cuya secuencia es idéntica a la secuencia de nucleótidos mostrada en la SEQ ID NO 1 y por un segundo cebador cuya secuencia es idéntica a la secuencia de nucleótidos mostrada en la SEQ ID NO 2.
3. Conjunto de cebadores para la tipificación de Pseudomonas savastanoi pv. savastanoi caracterizado porque al menos dos de los cebadores de dicho conjunto presentan secuencias idénticas a las secuencias mostradas en las SEQ ID NO1 y SEQ ID NO 3.
4. Conjunto de cebadores para la tipificación de Pseudomonas savastanoi pv. savastanoi según la reivindicación anterior caracterizado porque dicho conjunto está formado por un primer cebador cuya secuencia es idéntica a la secuencia de nucleótidos mostrada en la SEQ ID NO 1 y por un segundo cebador cuya secuencia es idéntica a la secuencia de nucleótidos mostrada en la SEQ ID NO 3.
5. Procedimiento para el diagnóstico y tipificación de Pseudomonas savastanoi pv. savastanoi caracterizado porque comprende:
a. la amplificación mediante PCR de una muestra de ADN usando un conjunto de cebadores descrito según cualquiera de las reivindicaciones1a2,
b. la digestión mediante la enzima de restricción HaeIII de los productos resultantes de la reacción de PCR, y
c. el diagnóstico de la muestra de ADN amplificada mediante PCR (a) mediante análisis de los fragmentos resultantes de la digestión (b) mediante electroforesis.
6. Procedimiento para el diagnóstico y tipificación de Pseudomonas savastanoi pv. savastanoi según la reivindicación anterior caracterizado porque la electroforesis se realiza en gel de agarosa al 3%.
7. Procedimiento para el diagnóstico y tipificación de Pseudomonas savastanoi pv. savastanoi caracterizado porque comprende:
a. la amplificación mediante PCR de una muestra de ADN usando un conjunto de cebadores descrito según cualquiera de las reivindicaciones1a2,
b. la amplificación mediante PCR usando un conjunto de cebadores descrito según cualquiera de las reivindicaciones 3 a 4 de una muestra de ADN previamente diagnosticada como Pseudomonas savastanoi pv. savastanoi, y
c. la tipificación de la muestra de ADN amplificada mediante PCR (b) mediante análisis de los productos resultantes mediante electroforesis.
8. Procedimiento para el diagnóstico y tipificación de Pseudomonas savastanoi pv. savastanoi según la reivindicación anterior caracterizado porque la electroforesis se realiza en gel de acrilamida al 6%.
9. Procedimiento para el diagnóstico y tipificación de Pseudomonas savastanoi pv. savastanoi según cualquiera de las reivindicaciones 7 a 8 caracterizado porque para la tipificación de la muestra de ADN, amplificada mediante PCR usando un conjunto de cebadores descrito según cualquiera de las reivindicaciones 3 a 4, mediante análisis de los productos resultantes mediante electroforesis se emplea un marcador de repeticiones TAC que comprende una colección de fragmentos de ADN amplificados a partir de alelos iaaL-Psv con un número variable de repeticiones TAC.
10. Kit para el diagnóstico y tipificación de Pseudomonas savastanoi pv. savastanoi que comprende al menos un conjunto de cebadores descrito según cualquiera de las reivindicaciones 1 a 2, y al menos un conjunto de cebadores descrito según cualquiera de las reivindicaciones3a4.
11. Kit para el diagnóstico y tipificación de Pseudomonas savastanoi pv. savastanoi según la reivindicación anterior que comprende, además, plásmidos control generados mediante la clonación en vectores plásmidos de fragmentos de ADN generados mediante PCR usando el conjunto de cebadores para el diagnóstico de Pseudomonas savastanoi pv. savastanoi descrito según cualquiera de las reivindicaciones1a2.
LISTA DE SECUENCIAS
<110> Universidad de Málaga Instituto Valenciano de Investigaciones Agrarias (IVIA)
<120> Conjunto de cebadores, procedimiento y kit para el diagnóstico y la tipificación de la bacteria patógena causante de la tuberculosis del olivo, Pseudomonas savastanoi pv. savastanoi <130> I Matas et al., 2010
<160> 3
<170> PatentIn version 3.3
<210> 1
<211> 18
<212> ADN
<213> Secuencia artificial <223> Secuencia cebador directo IAAL-F1
<400> 1 gcgaaactgc ctgatcgg 18
<210> 2
<211> 18
<212> ADN
<213> Secuencia artificial <223> Secuencia cebador directo IAAL-R1
<400> 2 gtcctcccgc tggatgtc 18
<210> 3
<211> 18
<212> ADN
<213> Secuencia artificial <223> Secuencia cebador directo IAAL-R2
<400> 3 tgctgcggaa gacgttgg 18
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]