CEBADORES ESPECIALES PARA LA REACCIÓN EN CADENA DE LA POLIMERASA.
Cebadores especiales para la reacción en cadena de la polimerasa.
La presente invención pertenece al campo de las técnicas de biología molecular, en particular al campo de la técnica de la reacción en cadena de la polimerasa (PCR). Más específicamente, se refiere a cebadores diseñados especialmente para la PCR y a la detección directa de una secuencia diana. Esto se consigue debido a su diseño especial y, más específicamente, a la presencia en el producto de amplificación obtenido, con el uso de dichos cebadores, de colas de cadena sencilla. Estas colas permiten la captura del producto de amplificación a un sustrato funcionalizado con sondas de captura y/o su detección sencilla con sondas informadoras.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201230120.
Solicitante: UNIVERSITAT ROVIRA I VIRGILI.
Nacionalidad solicitante: España.
Inventor/es: FRANK, RAINER, KATAKIS, IOANIS, O\'SULLIVAN,Ciara, BENI,Valerio, WILLEMS,Andreas, HANSEN-HAGGE,Thomas, JODA,Hamdi Abdelazim Osman.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
CEBADORES ESPECIALES PARA LA REACCiÓN EN CADENA DE LA POLlMERASA
CAM PO DE LA INVENCiÓN
La presente invención pertenece al campo de las técnicas de biología molecular, en particular al campo de la técnica de la reacción en cadena de la polimerasa (PCR) . Más específicamente, se refiere a cebadores diseñados especialmente para la PCR para la detección directa de secuencias diana amplificadas. Esto se consigue debido a su diseño especial y más específicamente a la presencia en los productos de amplificación obtenidos, con el uso de dichos cebadores, de colas de cadena sencilla. Estas colas permiten la captura del producto de amplificación a un sustrato funcional izado con sondas de captura y/o su detección sencilla con sondas informadoras marcadas.
ANTECEDENTES DE LA INVENCiÓN
La reacción en cadena de la polimerasa (PCR) es una técnica de biología molecular para amplificar una o varias copias de un segmento de ADN en varios órdenes de magnitud, generando miles de millones de copias de una secuencia de ADN particular.
Desarrollada en 1983 por Kar y Mullis, la PCR es en la actualidad una técnica habitual y a menudo indispensable, usada en laboratorios de investigación médica y biológica para una variedad de aplicaciones. Éstas incluyen la clonación de ADN para su secuenciación, filogenia basada en ADN, o el análisis funcional de genes; el diagnóstico de enfermedades hereditarias; la identificación de huellas genéticas (usadas en ciencia forense y en pruebas de paternidad) y la detección y diagnóstico de enfermedades infecciosas.
El procedimiento se basa en ciclos térmicos, que consisten en ciclos repetidos de calentamiento y enfriamiento de la reacción para la desnaturalización del ADN y el procesamiento enzimático del ADN. Los cebadores (fragmentos cortos de ADN) , que contienen secuencias complementarias a la región diana, junto con una ADN-polimerasa son componentes claves para permitir una amplificación selectiva y repetida. A medida que la PCR progresa, el propio ADN generado se usa como molde para su replicación, poniendo en marcha una reacción en cadena en la que el molde de ADN se amplifica exponencialmente. La PCR se puede modificar exhaustivamente para llevar a cabo una amplia variedad de manipulaciones genéticas. Casi todas las aplicaciones de la PCR emplean una ADN-polimerasa térmicamente estable, tal como la Taq polimerasa. Esta ADN-polimerasa ensambla enzimáticamente una nueva cadena de ADN a partir de bloques de construcción de ADN, los nucleótidos, usando ADN de cadena sencilla como molde y cebadores de ADN, que son necesarios para la iniciación de la síntesis del ADN. La amplia mayoría de procedimientos de PCR usan ciclos térmicos, es decir, el calentamiento y enfriamiento alternado de la muestra de PCR hasta una serie de pasos de temperatura definidos. Estos pasos de los ciclos térmicos son necesarios, en primer lugar, para separar físicamente las dos cadenas de una doble hélice de ADN a una temperatura elevada en un proceso denominado desnaturalización del ADN. A continuación, a una temperatura más baja, mediante la ADN-polimerasa cada cadena se usa como molde en la síntesis del ADN para amplificar selectivamente el ADN diana. La selectividad de la PCR proviene del uso de cebadores que son complementarios a la región de ADN elegida para su amplificación en condiciones de ciclos térmicos específicos.
Se han desarrollado muchas variantes de la PCR realizando pequeñas modificaciones del protocolo de PCR estándar, o ajustando o variando diferentes elementos de la PCR tales como, por ejemplo, el tipo de polimerasa usada.
Además, diferentes variantes de la PCR resultan de la variación de los cebadores para la amplificación. Por ejemplo, una de dichas variantes conocidas consiste en la inclusión de colas en el extremo 5' del cebador. Normalmente, los cebadores son totalmente complementarios al extremo 3' de la secuencia diana. No obstante, la polimerasa es tolerante a desapareamientos más allá del extremo 3'. Los cebadores con cola incluyen secuencias no complementarias en sus extremos 5'. Un procedimiento habitual es colocar sitios de restricción en los extremos de estas colas, facilitando su posterior inserción en vectores de clonación.
La presencia de colas en el producto de amplificación de la PCR también puede ser interesante puesto que permite la captura directa mediante la hibridación del producto a un sustrato o superficie que tenga sondas complementarias en él. También permite la unión de sondas complementarias marcadas para una fácil detección del producto de amplificación.
El documento US 2004/0038194 describe un procedimiento para la amplificación por PCR en el que los cebadores contienen un agente de captura. El documento menciona que, entre otros, el agente de captura puede ser un grupo sulfhidrilo, biotina, un dominio de unión a celulosa o una secuencia de nucleótidos específica. Sin embargo, el
documento no proporciona muchas indicaciones sobre el modo en el que se puede usar
una secuencia de nucleótidos específica como agente de captura.
El documento US 2003/0215926 proporciona una idea general de un procedimiento para la obtención de ADN de doble cadena con colas de cadena sencilla que comprende el uso de cebadores híbridos formados por ADN en el extremo 3' y por ARN en el extremo 5'. El extremo de ADN en 3' es complementario a la secuencia diana y sirve como cebador para la amplificación. Una vez realizada la PCR con estos cebadores se obtiene un dúplex híbrido de ADN-ARN que posteriormente se somete a digestión con ARNasa. De esta digestión resulta un dúplex de ADN con una cola de cadena sencilla en cada extremo que se puede detectar inmovilizado con sondas adecuadas. En el documento US 2009/0203531 también se describe un enfoque similar.
Hayashi G. Y col. (Nucleic Acids Symposium Series, 2005, No 49: 261-262; ChemBio Chem, 2007, 8: 169-171; y Journal of Biotechnology (2008) 135: 157-160) sigue un enfoque completamente diferente que se basa en el uso de las propiedades especiales del ADN-L. Hayashi G. usa como cebador una quimera de ADN-D y ADN-L. El cebador comprende ADN-D en el extremo 3' y ADN-L en el extremo 5'. Durante la PCR, la polimerasa detiene su amplificación en el límite entre el ADN-D y ADN-L, dejando al ADN-L en forma de cadenas sencillas en el producto de amplificación. A continuación el ADN-L se puede hibridar a sondas de ADN-L complementarias previamente inmovilizadas.
Los autores de la presente invención proporcionan cebadores diseñados especialmente, que contienen elementos diferentes unidos operativamente que permiten la generación de un producto de amplificación de la PCR de doble cadena con colas de cadena sencilla en uno o en ambos extremos del dúplex.
BREVE DESCRIPCiÓN DE LAS FIGURAS
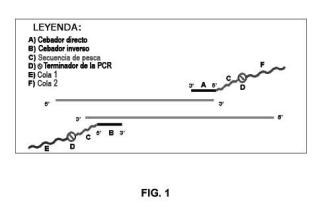
Figura 1: Esquema que representa un posible diseño del par de cebadores según la invención que comprenden tanto una secuencia de pesca como colas de captura e informadoras, respectivamente.
Figura 2: Esquema del ensayo de detección propuesto usando los pares de cebadores diseñados específicamente y detección electroquímica y/o espectrofotométrica.
Figura 3: A) Electroforesis en gel de la amplificación de una muestra positiva con los cebadores diseñados especiales en la que 1, 2, 3 Y 4 son el producto de la amplificación de una muestra positiva usando el par de cebadores L 1 Y L2 Y el Control NT es el producto de la amplificación en ausencia de la muestra (molde) . B) Electroforesis en gel de la muestra extraída y su comparación con el Marcador de pesos moleculares 200. El producto amplificado tiene una longitud de 240 pb que es consistente con lo esperado.
Figura 4: Prueba de concepto de la detección de ADNds, dt (ADN de doble cadena y doble cola) mediante ensayo ELONA. Control: Respuesta asociada a adsorción inespecífica de la sonda informadora enzimática; Producto extraído de la PCR: Respuesta obtenida para el ADNds, dt extraído del producto de la PCR usando el kit de extracción PureLink Quick Gel Extraction Kit.; Producto no extraído de la PCR: Respuesta obtenida para el mismo producto de la PCR antes de la extracción del ADNds, dt. Muestra de partida de la PCR diluida a 1 en 50.
Figura 5: Esquema de uno de los posibles enfoques contemplados para la limpieza de la muestra mediante la eliminación del cebador en exceso sin reaccionar usando perlas magnéticas.
Figura 6: Prueba de concepto de la viabilidad de la eliminación del cebador mediante pesca con perlas magnéticas. Control: Respuesta asociada a la adsorción inespecífica de la sonda informadora marcada enzimáticamente; PCR + imanes: Respuesta obtenida usando producto no purificado de la PCR después de su puesta...
Reivindicaciones:
1. El cebador para la amplificación por PCR que comprende operativamente unidos en direcció.
3. 5':
a. Una secuencia de acceso que consta de un oligonucleótido de 10 a nucleótidos capaz de hibridarse específicamente al extremo 3' de secuencia que se pretende amplificar, 35 la
b. Opcionalmente, una secuencia de pesca para la eliminación del cebador en exceso después de la reacción de PCR que consta de al menos 2 nucleótidos, la secuencia que está diseñada para hibridarse a una sonda, opcionalmente inmovilizada a un sustrato y que no interacciona con la secuencia diana,
c. Un terminador de la amplificación de la PCR,
d. Una cola de captura o informadora para la hibridación del producto de amplificación resultante a una sonda opcionalmente inmovilizada sobre un sustrato o una sonda marcada respectivamente, que consta de un ácido nucleico con una secuencia con una longitud de al menos dos bases diseñado para su hibridación a dicha sonda opcionalmente inmovilizada sobre un sustrato o dicha sonda marcada.
2. El cebador según la reivindicación 1, en el que la secuencia de acceso y la secuencia de pesca son dos secuencias de ADN.
3. El cebador según cualquiera de las reivindicaciones anteriores, en el que la secuencia de pesca tiene no menos de 15 nucleótidos.
4. El cebador según cualquiera de las reivindicaciones anteriores, en el que el terminador de la amplificación por PCR puede ser un terminador inorgánico como hexaetilenglicol, trietilenglicol, propilo, 1', 2'-didesoxirribosa o bases o ácidos nucleicos no naturales que no son reconocidos por la polimerasa tales como PNA, LNA, oligos Morfolino o ARN.
5. El cebador según cualquiera de las reivindicaciones anteriores, en el que la cola de captura o informadora comprende un ácido nucleico seleccionado entre ADN, PNA, oligos Morfolino o LNA y mezclas de estos ácidos nucleicos.
6. El cebador según cualquiera de las reivindicaciones anteriores, en el que el cebador
es un cebador directo o inverso.
7. Par de cebadores para la amplificación por PCR que consta de un cebador directo y un cebador inverso, en el que uno o los dos son según las reivindicaciones 1 a 6.
8. Par de cebadores según la reivindicación 7, en el que el cebador directo comprende una cola de captura y el cebador inverso comprende una cola informadora o en el que el cebador directo comprende una cola informadora y el cebador inverso comprende una cola de captura.
9. Uso de un cebador según cualquiera de las reivindicaciones 1 a 6 o de un par de cebadores según cualquiera de las reivindicaciones 7 a 8 para llevar a cabo una amplificación por PCR.
10. Uso según la reivindicación 9, en el que, después de la reacción por PCR, los cebadores en exceso se eliminan y se separan del producto de amplificación mediante hibridación a una sonda de pesca, opcionalmente inmovilizada a un sustrato, que comprende una secuencia capaz de hibridarse específicamente a la secuencia de pesca.
11. Producto de la PCR que se puede obtener mediante el uso de al menos un cebador según la reivindicación 1 que consiste en un dúplex de ADN con una cola de cadena sencilla correspondiente a la cola de captura o informadora presente en el cebador según la reivindicación 1.
12. Producto de la PCR que se puede obtener mediante el uso de un par de cebadores según la reivindicación 8, que comprende un dúplex de ADN con dos colas de cadena sencilla en cada extremo del dúplex, correspondientes a la cola de captura y/o informadora del par de cebadores según la reivindicación 8.
13. Un grupo de dos o más pares de cebadores según cualquiera de las reivindicaciones 7-8 para una PCR multiplex, en el que las colas de captura de cada uno de los pares de cebadores son diferentes entre sí y están diseñadas para ser individuales para cada secuencia de acceso, y en el que las colas informadoras de cada uno de los pares de cebadores están diseñadas para ser comunes entre sí o individuales para cada secuencia de acceso.
14. Un procedimiento para detectar la presencia de una secuencia diana en una muestra biológica que comprende:
a) preparar sondas de captura capaces de hibridarse a la cola de captura del cebador según cualquiera de las reivindicaciones 1 a 6 o del par de cebadores según cualquiera de las reivindicaciones 8 a 9, y/o sondas informadoras capaces de hibridarse a la cola informadora del cebador según cualquiera de las reivindicaciones 1 a 6 o del par de cebadores según cualquiera de las reivindicaciones 8 a 9,
b) realizar una amplificación por PCR con al menos un cebador según cualquiera de las reivindicaciones 1 a 6 o con un par de cebadores según cualquiera de las reivindicaciones 7 a 8 para obtener un producto de amplificación según las reivindicaciones 11 ó 12 respectivamente,
c) opcionalmente eliminar los cebadores en exceso que permanecen después de la amplificación por PCR de la etapa b) por medio de digestión enzimática o hibridación a una sonda de pesca, opcionalmente inmovilizada a un sustrato, que comprende una secuencia capaz de hibridarse específicamente a la secuencia de pesca,
d) permitir la hibridación entre las sondas de captura y/o las sondas informadoras de la etapa a) con las colas de captura y/o las colas informadoras del producto de amplificación,
e) detectar la presencia de hibridación entre las sondas de captura y/o las sondas informadoras de la etapa a) con las colas de captura y/o las colas informadoras del producto de amplificación, donde la presencia de dicha hibridación implica la presencia de la secuencia específica en la muestra y la no hibridación implica la ausencia de dicha secuencia problema en la muestra.
15. Un procedimiento según la reivindicación 14, donde las sondas de captura están inmovilizadas a un sustrato.
16. Un procedimiento según cualquiera de las reivindicaciones 14 y 15, donde la sonda informadora comprende un marcador que incluye moléculas electroquímicas redox, enzimas capaces de producir una reacción detectable, fluoróforos, moléculas capaces de producir electroquimioluminiscencia, cambios de color, quimioluminiscencia o fluorescencia, liposomas, partículas/cilindros metálicos, partículas/cilindros inorgánicos, partículas/cilindros poliméricos, partículas/cilindros magnéticos, puntos cuánticos, dendrímeros, marcadores radiactivos.
17. Un procedimiento según las reivindicaciones 14 y 16, donde la sonda de captura y la sonda informadora pueden ser un ácido nucleico seleccionado entre ADN, PNA, oligos Morfolino, LNA o sus combinaciones.
18. Un procedimiento según cualquiera de las reivindicaciones 14 a 17, donde la detección de la hibridación se lleva a cabo por medio de procedimientos electroquímicos,
ópticos, gravimétricos, termométricos, magnéticos y micromecánicos.
19. Un procedimiento según cualquiera de las reivindicaciones 14 a 18 en el que la amplificación por PCR es una PCR multiplex y se lleva a cabo con un grupo de cebadores según la reivindicación 13.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]