Amplificación preferente de ARNm respecto a ADN utilizando cebadores modificados químicamente.
Método de amplificación selectiva de un ARN mensajero diana, que comprende las etapas de:
a) hibridar un primer oligonucleótido con dicha diana de ARNm y llevar a cabo una síntesis de ADN dirigida por ARN utilizando por lo menos un enzima capaz de síntesis dirigida por ARN, en el que dicho primer oligonucleótido:
i. comprende por lo menos un nucleótido con una base adenina, guanina o citosina modificada covalentemente en el grupo amino exocíclico con un grupo bencilo o un grupo bencilo sustituido,
ii. es por lo menos parcialmente complementario a dicha diana de ARNm, y
iii. comprende una unión exón-exón en la diana,
b) amplificar el producto de la etapa a) utilizando dicho primer oligonucleótido y un segundo oligonucleótido con por lo menos un enzima capaz de síntesis de ADN dirigida por ADN, en el que dicho segundo oligonucleótido es por lo menos parcialmente complementario a dicha diana de ARNm.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2010/007559.
Solicitante: F. HOFFMANN-LA ROCHE AG.
Inventor/es: WILL,STEPHEN G, STEINER,LORI, TSAN,ALISON, NEWTON,NICOLAS.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2440073_T3.pdf
Fragmento de la descripción:
Amplificación preferente de ARNm respecto a ADN utilizando cebadores modificados químicamente
CAMPO DE LA INVENCIÓN
La presente invención se refiere al campo de la amplificación de los ácidos nucleicos y más concretamente al campo de la amplificación del ARN mediante reacción en cadena de la polimerasa con transcripción inversa (RT-PCR) .
ANTECEDENTES DE LA INVENCIÓN
La reacción en cadena de la polimerasa con transcripción inversa es un método para generar y amplificar exponencialmente copias de ADN a partir de un molde de ADN. El método presenta aplicaciones tanto cualitativas como cuantitativas en el campo de la expresión génica. El método permite tanto detectar como medir los niveles de ARNm expresados por un organismos.
La principal dificultad de la RT-PCR es la contaminación de las preparaciones de ARN con ADN genómico. Tal como admiten el distribuido principal de reactivos y tecnologías de aislamiento de ARN, la mayoría de técnicas de aislamiento de ARN proporcionan ARN con una cantidad significativa de ADN genómico contaminante (Ambion, Austin, Tex., (Life Technologies, Inc.) , Technical Bulletin nº 176, "Avoiding DNA contamination in RT-PCR") . La contaminación de ADN resulta especialmente problemática para la RT-PCR, en la que una cantidad mínima de ADN contaminante será amplificada exponencialmente. Un método para reducir la amplificación del ADN mediante RT-PCR implica el pretratamiento de las muestras con una desoxirribonucleasa, tal como ADNasa I; ver Huang et al., Biotechniques 20:1012-1020, 1996. Desafortunadamente, este enfoque no está exento de problemas. Tras el pretratamiento, la ADNasa debe inactivarse por completo para evitar la digestión de los amplicones de ADN nacientes durante el curso de la RT-PCR. Sin embargo, las elevadas temperaturas necesarias para completar la inactivación de la ADNasa causan la degradación del molde de ARN. A modo de alternativa al calentamiento, puede eliminarse químicamente la ADNasa mediante extracción con fenol o utilizando diversos reactivos complejos y costosos que eliminan la ADNasa de la mezcla de reacción. En resumen, la utilización de ADNasa no resulta práctica en la RT-PCR, ya que requiere múltiples etapas adicionales y con frecuencia amenaza la frágil diana de ARN.
Debido a que el problema de la contaminación con ADN se considera intratable, los esfuerzos se han dirigido a prevenir la amplificación del ADN contaminante mediante la RT-PCR. Una de estas estrategias aprovecha la presencia de intrones en el ADN genómico eucariótico. En el ARNm maduro, no hay intrones. En el caso de que los cebadores se diseñen para flanquear un intrón, éste se encontrará ausente del amplicón generado a partir de ARNm. Sin embargo, el intrón se incluirá en el amplicón de RT-PCR generado a partir del molde de ADN genómico correspondiente. En el caso de que el intrón sea suficientemente grande, la secuencia de ARNm más corta (y posteriormente la secuencia de ADNc) será amplificada preferentemente, mientras que el ADN genómico será amplificado menos eficientemente o no será amplificado en absoluto (Ambion, Tech. Bull. nº 176) . En el peor de los casos, el ADN genómico será coamplificado con la diana de ARNm deseada, pero los dos amplicones podrán distinguirse mediante electroforesis.
Desafortunadamente, el diseño del cebador no siempre puede superar el problema de contaminación con ADN. Muchos ensayos de PCR ahora implican PCR en tiempo real, una técnica que no incluye electroforesis pero que puede detectar ácidos nucleicos simultáneamente con la amplificación; ver las patentes US nº 5.994.056 y nº
5.876.930 y patentes relacionadas. Sin electroforesis, la PCR en tiempo real no puede eliminar en el análisis los amplicones de tamaño diferente generados con el mismo juego de cebadores. Cualquier sonda de PCR en tiempo real que detecte una diana de ARNm inevitablemente también detectará el contaminante de ADN genómico correspondiente. No se distinguirá un ARNm de su ADN genómico correspondiente. Por lo tanto, en el caso de que los intrones en la región de interés sean excesivamente pequeños para impedir la amplificación de ADN genómico, no podrá utilizarse la PCR en tiempo real.
Por lo tanto, resulta deseable crear un nuevo método de diseño de cebadores que garantiza que los contaminantes de ADN genómico no sean coamplificados con el ARNm durante la RT-PCR. Dicho método de diseño de cebadores permitiría la amplificación cuantitativa de dianas de ARNm con independencia del tamaño del intrón presente en el amplicón.
DESCRIPCIÓN RESUMIDA DE LA INVENCIÓN
En un primer aspecto, la invención se refiere a un método para amplificar selectivamente una diana de ARN mensajero en una muestra, que comprende una unión exón-exón, que comprende las etapas de: a) hibridar un primer oligonucleótido con dicha diana de ARNm y llevar a cabo síntesis de ADN dirigida por ARN utilizando por lo menos un enzima capaz de síntesis dirigida por ARN, en el que dicho primer oligonucleótido comprende por lo menos un nucleótido modificado en el grupo amino exocíclico, es por lo menos parcialmente complementario a dicha diana de ARN, y comprende una unión exón-exón en la diana, y b) amplificar el producto de la etapa a) utilizando dicho primer oligonucleótido y un segundo oligonucleótido con por lo menos un enzima capaz de síntesis de ADN dirigida por ADN, en el que dicho segundo oligonucleótido es por lo menos parcialmente complementario a dicha diana de ARNm. También se dan a conocer oligonucleótidos, mezclas de reacción y kits para la puesta en práctica de la presente invención.
BREVE DESCRIPCIÓN DE LAS FIGURAS
La figura 1 es una representación esquemática de una unión exón-exón tal como se define posteriormente en la presente memoria en el contexto de la invención, mostrada como un cebador que comprende la unión exón-exón.
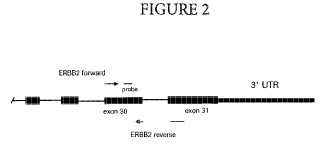
La figura 2 es una representación esquemática de un diseño de cebador según la presente invención.
La figura 3 muestra una curva de amplificación utilizando el cebador SEC ID nº 3 utilizado en el método según la presente invención.
La figura 4 muestra una curva de amplificación utilizando el cebador SEC ID nº 6 utilizado en el método según la presente invención.
La figura 5 muestra una curva de amplificación utilizando el cebador SEC ID nº 7 utilizado en el método según la presente invención.
La figura 6 muestra una curva de amplificación utilizando el cebador SEC ID nº 10 utilizado en el método según la presente invención.
La figura 7 muestra una curva de amplificación utilizando el cebador SEC ID nº 11 utilizado en el método según la presente invención.
La figura 8 muestra una curva de amplificación utilizando el cebador SEC ID nº 12 utilizado en el método según la presente invención.
La figura 9 muestra una curva de amplificación utilizando el cebador SEC ID nº 13 utilizado en el método según la presente invención.
La figura 10 muestra una curva de amplificación utilizando el cebador SEC ID nº 14 utilizado en el método según la presente invención.
La figura 11 muestra una curva de amplificación utilizando el cebador SEC ID nº 15 utilizado en el método según la presente invención.
DESCRIPCIÓN DETALLADA DE LA INVENCIÓN
Definiciones En la descripción y reivindicación de la presente invención se utilizarán las definiciones siguientes. A menos que se indique lo contrario, todos los términos técnicos y científicos utilizados en la presente memoria presentan los mismos significados entendidos comúnmente por el experto ordinario en la materia a la que se refiere la presente invención. La expresión "ARN mensajero" o "ARNm" se refiere al ARN que es transcrito a partir de ADN genómico y que porta la secuencia codificante para la síntesis de proteína. En los organismos eucarióticos, la secuencia de nucleótidos del ARNm se modifica con el fin de formar la secuencia codificante de proteína. Típicamente, la modificación implica el "procesamiento" o eliminación de los intrones de la secuencia de ARNm. En algunos casos, la secuencia de nucleótidos del ARNm se modifica mediante "edición" con el fin de formar la secuencia codificante de proteína.
En el contexto de la síntesis de ARNm, la expresión "ADN genómico correspondiente" se refiere al ADN genómico que contiene el molde para el ARNm en cuestión. El ADN genómico correspondiente puede contener secuencias adicionales que son similares o complementarias al molde del ARNm, tal como el gen en cuestión, así como duplicaciones de ese gen y pseudogenes. Típicamente, el ADN genómico correspondiente procede del mismo organismo que el ARNm;... [Seguir leyendo]
Reivindicaciones:
1. Método de amplificación selectiva de un ARN mensajero diana, que comprende las etapas de:
a) hibridar un primer oligonucleótido con dicha diana de ARNm y llevar a cabo una síntesis de ADN dirigida por ARN utilizando por lo menos un enzima capaz de síntesis dirigida por ARN, en el que dicho primer oligonucleótido:
i. comprende por lo menos un nucleótido con una base adenina, guanina o citosina modificada covalentemente en el grupo amino exocíclico con un grupo bencilo o un grupo bencilo sustituido,
ii. es por lo menos parcialmente complementario a dicha diana de ARNm, y
iii. comprende una unión exón-exón en la diana,
b) amplificar el producto de la etapa a) utilizando dicho primer oligonucleótido y un segundo oligonucleótido con por lo menos un enzima capaz de síntesis de ADN dirigida por ADN, en el que dicho segundo oligonucleótido es por lo menos parcialmente complementario a dicha diana de ARNm.
2. Método según la reivindicación 1, en el que la base modificada covalentemente en el grupo amino exocíclico del
nucleótido se selecciona de entre el grupo que consiste de N6-bencil-adenina, N6-para-terc-butil-bencil-adenina, N2alquil-guanina, N4-bencil-citosina y N4-para-terc-butil-bencil-citosina.
3. Método según la reivindicación 1, que comprende además una etapa c) de detección del producto de dicha síntesis de ADN dirigida por ARN y dirigida por ADN.
4. Método según la reivindicación 1, en el que el enzima capaz de síntesis dirigida por ARN y el enzima capaz de síntesis de ADN dirigida por ADN son el mismo enzima.
5. Método según la reivindicación 1, en el que dicho enzima o enzimas son polimerasas que presentan una actividad 30 de nucleas.
5. 3' que es 50% o menos de la actividad de la ADN polimerasa Taq.
6. Método según la reivindicación 1, en el que dicho enzima o enzimas se seleccionan de entre el grupo que consiste de ADN polimerasa Taq, ADN polimerasa Z05, ADN polimerasa delta-Z05 y ADN polimerasa delta-Z05-Gold o mutantes de las mismas.
7. Método según la reivindicación 1, en el que dicho primer oligonucleótido se selecciona de entre un grupo que consiste de SEC ID nº 3, 10, 11, 13, 14 y 15.
8. Oligonucleótido para la amplificación selectiva de un ARN mensajero diana, que comprende una unión exón-exón,
que comprende una secuencia de nucleótidos por lo menos parcialmente complementaria a dicho ARNm diana y que comprende la unión exón-exón en la diana y que además comprende por lo menos un nucleótido que presenta una base adenina, guanina o citosina modificada covalentemente en el grupo amino exocíclico con un grupo bencilo o un grupo bencilo sustituido.
9. Oligonucleótido según la reivindicación 8, en el que dicha base, modificada covalentemente en el grupo amino exocíclico se selecciona de entre el grupo que consiste de N6-bencil-adenina, N6-para-terc-butil-bencil-adenina, N2alquil-guanina, N4-bencil-citosina y N4-para-terc-butil-bencil-citosina.
10. Oligonucleótido según la reivindicación 8, en el que las estructuras de uno de dichos nucleótidos con una base 50 modificada covalentemente en el grupo amino exocíclico se selecciona de entre el grupo que consiste de:
en el que S representa una fracción sacárida y R representa un grupo bencilo o un grupo bencilo sustituido.
11. Mezcla de reacción para la amplificación selectiva de un ARN mensajero diana, que comprende una unión exónexón, comprendiendo la mezcla por lo menos un primer oligonucleótido, por lo menos parcialmente complementario a dicha diana de ARNm y que comprende la unión exón-exón en la diana, en la que dicho primer oligonucleótido comprende por lo menos un nucleótido con una base adenina, guanina o citosina modificada covalentemente en el grupo amino exocíclico con un grupo bencilo o un grupo bencilo sustituido, y por lo menos un segundo oligonucleótido por lo menos parcialmente complementario a dicha diana de ARNm.
12. Kit de amplificación selectiva de un ARN mensajero diana, que comprende una unión exón-exón, comprendiendo el kit: por lo menos un primer oligonucleótido, por lo menos parcialmente complementario a dicha diana de ARNm y que comprende una unión exón-exón en la diana, en el que dicho primer oligonucleótido comprende por lo menos un nucleótido con una base adenina, guanina o citosina modificada covalentemente en el grupo amino exocíclico con un grupo bencilo o un grupo bencilo sustituido, y por lo menos un segundo oligonucleótido por lo menos parcialmente complementario a dicha diana de ARNm.
13. Kit según la reivindicación 12 ó la mezcla de reacción según la reivindicación 11, que comprende además uno o más enzimas capaces de síntesis de ADN dirigida por ARN o ADN.
14. Kit según la reivindicación 12 ó la mezcla de reacción según la reivindicación 11, que comprende además nucleósidos trifosfato e iones orgánicos e inorgánicos, adecuado para proporcionar soporte a la polimerización de ácidos nucleicos.
15. Kit según la reivindicación 12 ó la mezcla de reacción según la reivindicación 11, que comprende además reactivos necesarios para la detección de ácidos nucleicos.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]