Proteína muNS capaz de formar inclusiones en el retículo endoplasmático, métodos de uso y usos de la misma.
Proteína muNS capaz de formar inclusiones en el retículo endoplasmático,
métodos de uso y usos de la misma.
La invención se relaciona con un polinucleótido que codifica un polipéptido basado en la región mínima de la proteína muNS de los Orthoreovirus capaz de formar inclusiones en el retículo endoplasmático, así como con dicho polipéptido. Asimismo, la invención se relaciona con un método de purificación y un método para la detección de la interacción entre dos polipéptidos, basado en la capacidad de algunas regiones de la proteína muNS de los Orthoreovirus de incorporarse a las inclusiones, junto con un péptido de interés.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201431378.
Solicitante: UNIVERSIDADE DE SANTIAGO DE COMPOSTELA.
Nacionalidad solicitante: España.
Inventor/es: BENAVENTE MARTINEZ,Francisco Javier, MARTÍNEZ COSTAS,José Manuel, BARREIRO PIÑEIRO,Natalia.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07K14/14 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 14/00 Péptidos con más de 20 aminoácidos; Gastrinas; Somatostatinas; Melanotropinas; Sus derivados. › Reoviridae, p. ej. rotavirus, virus de la lengua azul de la oveja, virus de la fiebre de garrapatas del Colorado.
- C07K19/00 C07K […] › Péptidos híbridos (Inmoglobulinas híbridas compuestas solamente de inmoglobulinas C07K 16/46).
- C12N15/46 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Reoviridae, p. ej. rotavirus, virus de la lengua azul de la oveja, virus de la fiebre de garrapatas del Colorado.
- C12N15/62 C12N 15/00 […] › Secuencias de ADN que codifican proteínas de fusión.
- C12N5/10 C12N […] › C12N 5/00 Células no diferenciadas humanas, animales o vegetales, p. ej. líneas celulares; Tejidos; Su cultivo o conservación; Medios de cultivo para este fin (reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00). › Células modificadas por introducción de material genético extraño, p. ej. células transformadas por virus.
PDF original: ES-2548784_A1.pdf
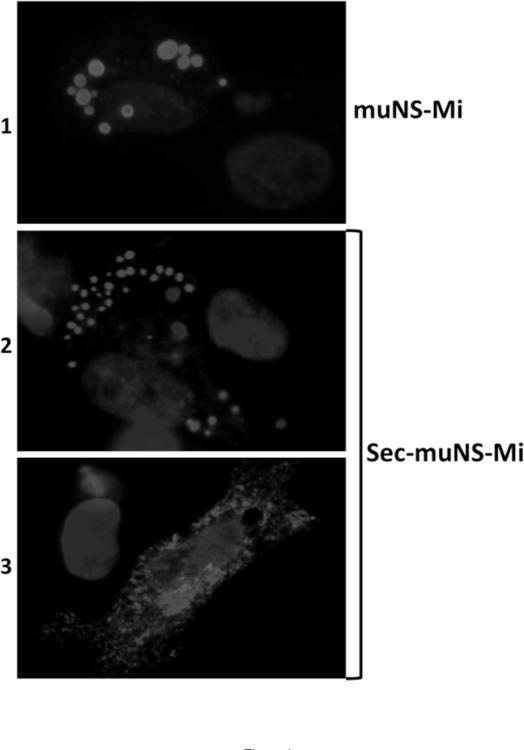
Fragmento de la descripción:
PROTEÍNA muNS CAPAZ DE FORMAR INCLUSIONES EN EL RETÍCULO ENDOPLASMÁTICO, MÉTODOS DE USO Y USOS DE LA MISMA
CAMPO DE LA INVENCIÓN
La invención se relaciona con un polinucleótido que codifica un polipéptido basado en la región mínima de la proteína muNS de los Orthoreovirus capaz de formar inclusiones en el
retículo endoplasmático, así como con dicho polipéptido. Asimismo, la invención se relaciona con un método de purificación y un método para la detección de la interacción entre dos polipéptidos, basado en la capacidad de algunas regiones de la proteína muNS de los Orthoreovirus de incorporarse a las inclusiones, junto con un péptido de interés.
ANTECEDENTES DE LA INVENCIÓN
Los reovirus aviares son miembros del género Orthoreovirus, uno de los 12 géneros de la familia Reoviridae. Estos virus son importantes patógenos de aves y causan importantes pérdidas económicas en la industria avícola. Los reovirus aviares son virus sin envoltura 20 lipídica que se replican en el citoplasma de las células infectadas y poseen un genoma de 10 segmentos de ARN de doble cadena rodeados por una doble cubierta proteica concéntrica de 85 nm de diámetro. Los segmentos genómicos están divididos en tres clases en función de su movilidad electroforética, tres de la clase L (large o grande) , otros tres de la M (medium o mediana) y cuatro de la S (small o pequeña) . Con excepción del segmento S1, que es 25 tricistrónico, todos los demás genes son monocistrónicos. Los segmentos genómicos se transcriben por medio de una polimerasa dependiente de ARN para producir ARN mensajeros (ARNm) con una secuencia nucleotídica idéntica a la de la cadena positiva del segmento de ARN de doble cadena. Los ARNm virales ejercen una doble función en las células infectadas: programan la síntesis de proteínas virales en los ribosomas, y sirven como molde para la síntesis de las cadenas negativas de los segmentos genómicos.
El genoma del reovirus aviar codifica al menos 12 proteínas, de las cuales 8 son estructurales (que se incorporan al virión) , y 4 no estructurales, que se expresan en células infectadas, pero no forman parte de los reoviriones maduros. Las proteínas codificadas por
clase se les ha asignado un sufijo alfabético (λA, λB, etc.) según su movilidad electroforética. El reovirión contiene al menos 10 proteínas estructurales diferentes, 8 de las cuales (λA, λB, λC, µA, µB, σA, σB y σC) son productos primarios de traducción de sus mRNAs, mientras que las otras dos, µBN y µBC, se originan por procesamiento proteolítico del precursor µB. Además de las proteínas estructurales, los reovirus aviares expresan cuatro proteínas no estructurales. Así, los genes M3 y S4 expresan dos proteínas no estructurales mayoritarias, denominadas µNS y σNS, respectivamente mientras que p10 y p17 están codificadas por los dos primeros cistrones del gen S1.
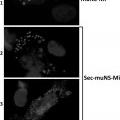
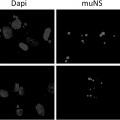
Los reovirus aviares se replican en inclusiones globulares citoplasmáticas denominadas factorías virales o viroplasmas, las cuales contienen proteínas virales estructurales y no estructurales, sin embargo carecen de membranas y organelas celulares. La expresión individual de proteínas virales en células transfectadas reveló que la proteína no estructural muNS es la única proteína del reovirus aviar capaz de formar inclusiones cuando se expresa en ausencia de otros factores virales (Touris-Otero et al., 2004; citado ad supra) . Esto, y el hecho de que las inclusiones globulares citoplasmáticas formadas por muNS en células transfectadas son muy similares en apariencia a las factorías virales de células infectadas, sugieren que muNS es el factor viral mínimo requerido para la formación de factorías virales en células infectadas con el reovirus aviar. El análisis de células transfectadas que coexpresan muNS y otras proteínas virales reveló que muNS juega un papel importante en estadios tempranos de la morfogénesis del virus y que el reclutamiento de proteínas del reovirus aviar a las factorías virales es un proceso selectivo y controlado temporalmente.
Los reovirus de mamífero también se replican en inclusiones globulares citoplasmáticas. De la misma manera que con los reovirus aviares, la proteína muNS no estructural se ha visto implicada en la formación de las inclusiones, así como en el reclutamiento de otros componentes a las inclusiones para posibles implicaciones en la replicación del genoma y en el ensamblaje de las partículas.
A pesar de que las proteínas muNS de reovirus aviar y de mamífero muestran únicamente un 28, 3% de identidad de secuencia, ambas contienen dos regiones en su extremo C-terminal con una alta probabilidad de estructura “coiled-coil”. Por otro lado, la proteína de mamífero es 86 aminoácidos más larga y es capaz de hacer más contactos primarios con otras proteínas virales estructurales y no estructurales que la proteína aviar. Aunque las proteínas muNS de todas las cepas de reovirus mamíferos (MRV) producen inclusiones globulares cuando se expresan en células transfectadas, la mayoría de las cepas producen factorías virales con 3 10
morfología filamentosa durante la infección. El fenotipo filamentoso de las factorías de reovirus de mamífero se ha atribuido a la proteína mu2, debido a su capacidad de asociarse tanto con microtúbulos y con muNS de reovirus mamífero. La expresión de versiones truncadas de muNS de MRV en células transfectadas reveló que el segmento entre los residuos 471-721 es la región más pequeña de muNS necesaria y suficiente para formar inclusiones. Se predice que esta región contiene dos segmentos de secuencias con alta probabilidad de formar estructuras “coiled-coil”, que están unidas por una región, precedida por un tramo de aproximadamente 50 residuos y seguido por una cola C-terminal.
Existen en la actualidad varios sistemas diseñados para la determinación de la interacción entre proteínas, de los cuales el sistema del doble híbrido es el más popular. Este sistema se basa en la expresión de dos proteínas de fusión: una en la que la proteína X está fusionada al dominio de unión a DNA del factor de transcripción GCN4; y otra en la que la proteína Y está fusionada al dominio de activación de transcripción del mismo factor GCN4. Si X e Y interaccionan, se supone que reconstituyen un GCN4 funcional en la célula que activará la transcripción de un gen reportero. Entre los problemas más obvios de este sistema se incluyen: i) aunque X e Y interaccionen, la arquitectura de dicha interacción muchas veces no permite la reconstrucción de un GCN4 funcional; ii) las fusiones pueden alterar las estructuras de los diferentes dominios de GCN4 o de los dominios de interacción de las proteínas problema.
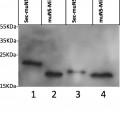
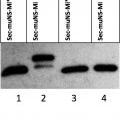
Se ha descrito un nuevo sistema que utiliza la formación de inclusiones por la proteína muNS de los reovirus de mamífero como una plataforma para detectar interacciones entre proteínas in vivo en células de mamífero y que ha sido adaptado también para usarse en levaduras. En este sistema, la proteína problema se fusiona con la zona C-terminal de muNS para que la fusión genere inclusiones citoplasmáticas y atraiga a ellas al ligando de la proteína problema. En el sistema de levaduras, estos autores demuestran que su sistema es superior al del doble híbrido en el número y tipo de interacciones detectadas, al menos con las proteínas ensayadas en dicho trabajo. Sin embargo, este sistema presenta varios problemas, entre los que se destacan: i) ciertas proteínas pueden plegarse incorrectamente al fusionarlas con la muNS-Mi y perder capacidad de interacción con sus ligandos; ii) algunas proteínas pueden interferir con la capacidad de formación de inclusiones de muNS-Mi y bien, no formarlas o generar agregados intracelulares, viéndose enormemente alterada la detección de interacciones; iii) la localización intracelular de la proteína problema o del ligando puede no ser la adecuada para poder ser detectada en inclusiones citoplasmáticas.
El documento WO 2011/098652 describe un sistema que emplea la formación de inclusiones por la proteína muNS de Orthoreovirus como plataforma para la purificación de proteínas y para la detección de interacciones entre proteínas tanto in vivo como in vitro. Esta plataforma está basada en la mínima región de la proteína muNS de Orthoreovirus aviar capaz de formar inclusiones, la cual recluta a las mismas una etiqueta peptídica y a las proteínas unidas a la misma. La etiqueta peptídica comprende la mínima región de la proteína muNS de un Orthoreovirus con capacidad de ser incorporada a las inclusiones formadas por la proteína muNS.
No obstante, ninguna de las aproximaciones... [Seguir leyendo]
Reivindicaciones:
1. Un polinucleótido que codifica un polipéptido que comprende (i) un primer componente que comprende un péptido señal de la vía secretora, y
(ii) un segundo componente que comprende la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula o una variante funcionalmente equivalente de la misma,
en donde el primer componente se encuentra en posición N-terminal con respecto al 10 segundo componente.
2. Polinucleótido según la reivindicación 1, en donde el primer componente comprende la secuencia SEQ ID NO: 1.
3. Polinucleótido según las reivindicaciones 1 o 2 en donde el segundo componente comprende la secuencia SEQ ID NO: 11.
4. Polinucleótido según la reivindicaciones 3, en donde el segundo componente comprende la secuencia SEQ ID NO: 11 o una variante funcionalmente equivalente de 20 la misma en donde el aminoácido en posición 57 no es un resto de asparagina.
5. Polinucleótido según la reivindicación 4, en donde el segundo componente comprende la secuencia SEQ ID NO: 13.
6. Polinucleótido según cualquiera de las reivindicaciones 1 a 5, en donde el polipéptido comprende además un péptido para facilitar su purificación en posición C-terminal con respecto al primer componente.
7. Polinucleótido según cualquiera de las reivindicaciones 1 a 6, en donde el polipéptido comprende además un polipéptido de interés en posición C-terminal con respecto al primer componente, y en donde el polipéptido de interés se selecciona del grupo formado por un antígeno viral, un antígeno bacteriano, un antígeno fúngico, un alérgeno o antígeno medioambiental y un antígeno tumoral.
8. Un polipéptido codificado por el polinucleótido de cualquiera según las reivindicaciones 1 a 7.
9. Un polinucleótido que codifica una proteína de fusión que comprende:
(i) un primer componente que comprende un péptido señal de la vía secretora,
(ii) un segundo componente que comprende un polipéptido de interés, y
(iii) un tercer componente que comprende la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de incorporarse a inclusiones formadas por una proteína que comprende la mínima región de muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula, o una variante funcionalmente equivalente de la misma. en donde el primer componente se encuentra en posición N-terminal con respecto al segundo y al tercer componente, y en donde el polipéptido de interés se selecciona del grupo formado por un antígeno viral, un antígeno bacteriano, un antígeno fúngico, un alérgeno o antígeno medioambiental y un antígeno tumoral.
10. Polinucleótido según la reivindicación 9, en donde el primer componente comprende la secuencia SEQ ID NO: 1.
11. Polinucleótido según cualquiera de las reivindicaciones 9 a 10, en donde dicho tercer 20 componente se selecciona del grupo formado por -un polipéptido que comprende la secuencia SEQ ID NO: 21, o una variante funcionalmente equivalente de la misma; -un polipéptido que comprende la secuencia SEQ ID NO: 22, o una variante funcionalmente equivalente de la mism.
25. un polipéptido que comprende la secuencia SEQ ID NO: 23, o una variante funcionalmente equivalente de la misma y
- un polipéptido que comprende la secuencia SEQ ID NO: 24, o una variante funcionalmente equivalente de la misma.
12. Polinucleótido según cualquiera de las reivindicaciones 10 u 11, en donde la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de incorporarse a inclusiones formadas por una proteína que comprende la mínima región de muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula, o la variante funcionalmente equivalente de la misma, carecen de secuencias consenso de N-glicosilación.
13. Polinucleótido según la reivindicación 12, en donde el tercer componente comprende la secuencia SEQ ID NO: 25.
14. Polinucleótido según la reivindicación 13, en donde el tercer componente comprende 5 la secuencia SEQ ID NO: 26.
15. Polinucleótido según cualquiera de las reivindicaciones 10 a 14, en donde la proteína de fusión comprende además un péptido para facilitar su purificación en posición C-terminal con respecto al primer componente.
16. Una proteína de fusión codificada por el polinucleótido según cualquiera de las reivindicaciones 9 a 15.
17. Una célula que comprende el polinucleótido según cualquiera de las reivindicaciones 1
a 7 o 9 a 15, el polipéptido según la reivindicación 9, o la proteína de fusión según la reivindicación 16.
18. Un kit que comprende:
(i) un componente A seleccionado del grupo de.
20. un polinucleótido según cualquiera de las reivindicaciones 1 a 7; y
- una célula que expresa un polinucleótido según cualquiera de las reivindicaciones 1 a 7; y
(ii) un componente B que comprende un polinucleótido según cualquiera de las reivindicaciones 9 a 15. 25
19. Método de purificación de un polipéptido que comprende la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula, en donde dicho método comprende (i) expresar en una célula un polinucléotido según la reivindicación 7 en condiciones adecuadas para la translocación del polipéptido a la vía secretora y la formación de inclusiones a partir de dicha región mínima, y
(ii) purificar las inclusiones formadas en la etapa (i) .
20. Método según la reivindicación 19, en donde el polipéptido comprende además un 35 péptido para facilitar su purificación en posición C-terminal con respecto al primer
componente. 76
21. Método de purificación de una proteína de fusión que comprende un polipéptido de
interés y la región mínima de la proteína muNS de un Orthoreovirus que tiene la
capacidad de incorporarse a inclusiones formadas por una proteína que comprende la
5 mínima región de muNS de un Orthoreovirus que tiene la capacidad de formar
inclusiones cuando se expresa en una célula, en donde el polipéptido de interés se
selecciona del grupo formado por un antígeno viral, un antígeno bacteriano, un
antígeno fúngico, un alérgeno o antígeno medioambiental y un antígeno tumoral,
dicho método comprende
10 (i) expresar en una célula un primer polinucleótido, en donde dicho primer
polinucleótido es un polinucleótido de acuerdo a cualquiera de las
reivindicaciones 1 a 7, en condiciones adecuadas para la translocación del
polipéptido a la vía secretora y la formación de inclusiones a partir de dicha
región mínima,
15 (ii) expresar en dicha célula un segundo polinucleótido, en donde dicho segundo
polinucleótido es un polinucleótido de acuerdo a cualquiera de las
reivindicaciones 9 a 15, en condiciones adecuadas para la translocación del
polipéptido a la vía secretora y para el reclutamiento de la proteína de fusión a
las inclusiones producidas en la etapa (i) , y
20 (iii) purificar los complejos que comprenden las inclusiones formadas en la etapa
(i) y las proteínas de fusión producidas en la etapa (ii) ,
en donde las etapas (i) y (ii) se pueden llevar a cabo en cualquier orden
22. Método de purificación de una proteína de fusión que comprende
25 (i) un polipéptido de interés, y
(ii) la región mínima de la proteína muNS de un Orthoreovirus que tiene la
capacidad de incorporarse a inclusiones formadas por una proteína que
comprende la mínima región de muNS de un Orthoreovirus que tiene la
capacidad de formar inclusiones cuando se expresa en una célula,
30 a partir de una composición en la que se encuentra dicha proteína de fusión, en donde
dicho método comprende:
(i) poner en contacto dicha composición con inclusiones formadas por un
polipéptido que comprende la región mínima de la proteína muNS de un
Orthoreovirus que tiene la capacidad de formar inclusiones cuando se
35 expresa en una célula y que carece de secuencias consenso de N
glicosilación en condiciones adecuadas para el reclutamiento de la proteína de fusión a dichas inclusiones;
(ii) purificar el complejo en la etapa (i) formado entre las inclusiones y la proteína de fusión. 5
23. Método según cualquiera de las reivindicaciones 21 o 22, que comprende adicionalmente separar la proteína de fusión de las inclusiones.
24. Método según cualquiera de las reivindicaciones 21 a 23, en donde el polipéptido que
comprende la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula y que carece de secuencias consenso de N-glicosilación comprende la secuencia SEQ ID NO: 11 en donde el aminoácido en posición 57 no es un resto de asparagina.
25. Método según cualquiera de las reivindicaciones 21 a 24, en donde el polipéptido que comprende la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula y que carece de secuencias consenso de N-glicosilación comprende la secuencia SEQ ID NO: 13.
26. Método para la purificación de una proteína de fusión que comprende
(a) un polipéptido de interés, y
(b) la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de incorporarse a inclusiones formadas por una proteína que comprende la mínima región de muNS de un Orthoreovirus que tiene la
capacidad de formar inclusiones cuando se expresa en una célula, a partir de una composición en la que se encuentra dicha proteína de fusión, en donde dicho método comprende:
(i) poner en contacto un polipéptido que comprende la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de formar
inclusiones cuando se expresa en una célula y que carece de secuencias consenso de N-glicosilación con dicha composición; en donde dicho polipéptido con capacidad de formar inclusiones se encuentra en forma soluble, y en donde dicha puesta en contacto se realiza en presencia de una concentración de cationes divalentes adecuada para la formación de 78
inclusiones a partir del polipéptido con capacidad de formar inclusiones y del reclutamiento de la proteína de fusión a dichas inclusiones, y
(ii) purificar el complejo formado entre las inclusiones y la proteína de fusión en la etapa (i) .
27. Método según cualquiera de las reivindicaciones 21 a 26, que comprende adicionalmente separar la proteína de fusión de las inclusiones.
28. Método según cualquiera de las reivindicaciones 21 a 27, en donde dicha la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de incorporarse a inclusiones formadas por una proteína que comprende la mínima región de muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula se selecciona del grupo formado por
- un polipéptido que comprende la secuencia SEQ ID NO: 21, o una variante funcionalmente equivalente de la misma; -un polipéptido que comprende la secuencia SEQ ID NO: 22, o una variante funcionalmente equivalente de la misma -un polipéptido que comprende la secuencia SEQ ID NO: 23, o una variante funcionalmente equivalente de la misma y -un polipéptido que comprende la secuencia SEQ ID NO: 24, o una variante funcionalmente equivalente de la misma.
29. Método según la reivindicación 28, en donde la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de incorporarse a inclusiones formadas por una proteína que comprende la mínima región de muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula comprende la secuencia SEQ ID NO: 8.
30. Método según cualquiera de las reivindicaciones 21 a 29, en donde la proteína de fusión comprende además un péptido para facilitar su purificación en posición C-terminal con respecto al primer componente.
31. Método para la detección de la interacción entre un primer polipéptido y un segundo polipéptido, en donde
- el primer polipéptido es el polipéptido de interés según la reivindicación 7,
y
- el segundo polipéptido está comprendido en una proteína de fusión I,
comprendiendo dicha proteína de fusión I un péptido señal de la vía
5 secretora y dicho segundo polipéptido, en donde en donde el péptido
señal se encuentra en posición N-terminal con respecto al segundo
polipéptido,
dicho método comprende:
(i) expresar en una célula un polinucleótido según la reivindicación 7, y mantener
10 dicha célula en condiciones adecuadas para la formación de inclusiones;
(ii) expresar en dicha célula un polinucleótido que codifica dicha proteína de
fusión I, y mantener dicha célula en condiciones adecuadas para que se
exprese dicho segundo polipéptido y
(iii) determinar si el segundo polipéptido se encuentra asociado al complejo
15 formado por las inclusiones generadas en la etapa (i) , en donde si se detecta
el segundo polipéptido es indicativo de la interacción entre el primer y el
segundo polipéptido,
en donde las etapas (i) y (ii) se llevan a cabo en cualquier orden.
20 32. Método para la detección de la interacción entre un primer polipéptido y un segundo
polipéptido, en donde
- el primer polipéptido está comprendido en una proteína de fusión II,
comprendiendo dicha proteína de fusión II:
(a) un primer componente que comprende un péptido señal de la vía
25 secretora,
(b) un segundo componente que comprende dicho primer polipéptido,
y
(c) un tercer componente que comprende la región mínima de la
proteína muNS de un Orthoreovirus que tiene la capacidad de
30 incorporarse a inclusiones formadas por una proteína que
comprende la mínima región de muNS de un Orthoreovirus que
tiene la capacidad de formar inclusiones cuando se expresa en
una célula,
en donde el primer componente se encuentra en posición N-terminal con
35 respecto al segundo y al tercer componente, y
- el segundo polipéptido está comprendido en una proteína de fusión I,
comprendiendo dicha proteína de fusión I un péptido señal de la vía
secretora y dicho segundo polipéptido, en donde en donde el péptido
señal se encuentra en posición N-terminal con respecto al segundo
5 polipéptido,
dicho método comprende:
(i) expresar en una célula un primer polinucleótido según cualquiera de las
reivindicaciones 1 a 7 y mantener dicha célula en condiciones adecuadas
para la formación de inclusiones;
10 (ii) expresar en dicha célula un segundo polinucleótido que codifica dicha
proteína de fusión II en condiciones adecuadas para que la proteína de fusión
se dirija a las inclusiones formadas en la etapa (i) ;
(iii) expresar en dicha célula un tercer polinucleótido que codifica una proteína de
fusión I, y mantener dicha célula en condiciones adecuadas para que se
15 exprese dicho segundo polipéptido y
(iv) determinar si el segundo polipéptido se encuentra asociado al complejo
formado por las inclusiones generadas en la etapa (i) y la proteína de fusión II
expresada en la etapa (ii) , en donde si se detecta el segundo polipéptido es
indicativo de la interacción entre dicho primer y segundo polipéptido,
20 en donde las etapas (i) , (ii) y (iii) se llevan a cabo en cualquier orden.
33. Método para la detección de la interacción entre un primer polipéptido y un segundo
polipéptido, en donde
- el primer polipéptido está comprendido en una proteína de fusión III,
25 comprendiendo dicha proteína de fusión III la región mínima de la proteína
muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones
cuando se expresa en una célula y que carece de secuencias consenso
de N-glicosilación y el primer polipéptido,
dicho método comprende,
30 (i) poner en contacto inclusiones formadas por dicha proteína de fusión III con el
segundo polipéptido, y
(ii) detectar la asociación del segundo polipéptido con las inclusiones,
en donde la detección del segundo polipéptido en las inclusiones es indicativo de la
interacción entre el primer polipéptido y el segundo polipéptido.
35
34. Método para la detección de la interacción entre un primer polipéptido y un segundo polipéptido, en donde -el primer polipéptido está comprendido en una proteína de fusión IV, comprendiendo dicha proteína de fusión la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de incorporarse a inclusiones formadas por una proteína que comprende la mínima región de muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula y el primer polipéptido,
dicho método comprende 10 (i) poner en contacto (a) inclusiones formadas por una proteína de fusión V que comprende la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula y que carece de secuencias consenso de N-glicosilación,
(b) dicha proteína de fusión IV, y
(c) el segundo polipéptido y
(ii) detectar la asociación del segundo polipéptido con las inclusiones, en donde la detección del segundo polipéptido en las inclusiones es indicativo de la interacción entre el primer polipéptido y el segundo polipéptido.
35. Método según cualquiera de las reivindicaciones 31 o 33, en donde el primer polipéptido comprende además un péptido para facilitar su purificación en posición C-terminal con respecto al primer componente.
36. Método según cualquiera de las reivindicaciones 32 o 34, en donde dicha la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de incorporarse a inclusiones formadas por una proteína que comprende la mínima región de muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula se selecciona del grupo formado por
- un polipéptido que comprende la secuencia SEQ ID NO: 21, o una variante funcionalmente equivalente de la misma; -un polipéptido que comprende la secuencia SEQ ID NO: 22, o una variante funcionalmente equivalente de la misma -un polipéptido que comprende la secuencia SEQ ID NO: 23, o una variante 35 funcionalmente equivalente de la misma y
-un polipéptido que comprende la secuencia SEQ ID NO: 24, o una variante funcionalmente equivalente de la misma.
37. Método según cualquiera de las reivindicaciones 32, 34 o 36, en donde la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de incorporarse a inclusiones formadas por una proteína que comprende la mínima región de muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula carece de secuencias consenso de N-glicosilación.
38. Método según la reivindicación 37, en donde la región mínima de la proteína muNS de un Orthoreovirus que tiene la capacidad de incorporarse a inclusiones formadas por una proteína que comprende la mínima región de muNS de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando se expresa en una célula comprende la secuencia SEQ ID NO: 25.
39. Método según cualquiera de las reivindicaciones 32, 34 o 36 a 38, en donde la proteína de fusión comprende además un péptido para facilitar su purificación en posición C-terminal con respecto al primer componente.
40. Uso de un polinucleótido según cualquiera de las reivindicaciones 9 a 15 para incorporar el segundo componente del polipéptido codificado por dicho polinucleótido a las inclusiones resultantes de la expresión en una célula del polinucleótido según cualquiera de las reivindicaciones 1 a 8.
41. Polinucleótido según cualquiera de las reivindicaciones 1 a 7 o 9 a 15 para su uso en medicina.
42. Uso de un polinucleótido según cualquiera de las reivindicaciones 1 a 7 o 9 a 15 para la preparación de un medicamento para el tratamiento de una enfermedad en la que 30 es necesaria la expresión del polipéptido de interés.
82a
Figura 1 83
Figura 2
Figura 3
Figura 4 86
Figura 5 87
Figura 6
Figura 7
LISTA DE SECUENCIAS
<110> UNIVERSIDADE DE SANTIAGO DE COMPOSTELA
<120> PROTEÍNA muNS CAPAZ DE FORMAR INCLUSIONES EN EL RETÍCULO ENDOPLASMÁTICO, MÉTODOS DE USO Y USOS DE LA MISMA
<130> P11023ES00
<160> 37
<170> PatentIn versión 3.5
<210> 1
<211> 20
<212> PRT
<213> Secuencia Artificial
<220>
<223> péptido señal
<400> 1
Met Gly Trp Ser Leu Ile Leu Leu Phe Leu Val Ala Val Ala Thr Gly 1 5 10 15
Val His Ser Gln 20
<210> 2
<211> 29
<212> PRT
<213> Secuencia Artificial
<220>
<223> péptido señal
<400> 2
Met Met Ser Phe Val Ser Leu Leu Leu Val Gly Ile Leu Phe Trp Ala 1 5 10 15
Thr Glu Ala Glu Gln Leu Thr Lys Cys Glu Val Phe Gln 20 25
<210> 3
<211> 25
<212> PRT
<213> Homo sapiens
<400> 3
Met Gly Thr Ala Arg Ile Ala Pro Gly Leu Ala Leu Leu Leu Cys Cys 1 5 10 15
Pro Val Leu Ser Ser Ala Tyr Ala Leu
<210> 4
<211> 25
<212> PRT
<213> Homo sapiens
<400> 4
Met Ser Val Leu Thr Pro Leu Leu Leu Arg Gly Leu Thr Gly Ser Ala 1 5 10 15
Arg Arg Leu Pro Val Pro Arg Ala Lys 20 25
<210> 5
<211> 20
<212> PRT
<213> Homo sapiens
<400> 5
Met Val Leu Leu Leu Ile Leu Ser Val Leu Leu Leu Lys Glu Asp Val 1 5 10 15
Arg Gly Ser Ala 20
<210> 6
<211> 38
<212> PRT
<213> Homo sapiens
<400> 6
Met Ala Ser Pro Arg Ser Ser Gly Gln Pro Gly Pro Pro Pro Pro Pro 1 5 10 15
Pro Pro Pro Pro Ala Arg Leu Leu Leu Leu Leu Leu Leu Pro Leu Leu 20 25 30
Leu Pro Leu Ala Pro Gly 35
<210> 7
<211> 17
<212> PRT
<213> Homo sapiens
<400> 7
Met Leu Leu Ser Val Pro Leu Leu Leu Gly Leu Leu Gly Leu Ala Val 1 5 10 15
Ala
<210> 8
<211> 31
<212> PRT
<213> Homo sapiens
<400> 8
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly 1 5 10 15
Lys Gly Leu Thr Val Ala Gly Leu Arg Ser Gly His Ile Tyr Gly 20 25 30
<210> 9
<211> 635
<212> PRT
<213> Orthoreovirus aviar
<400> 9
Met Ala Ser Thr Lys Trp Gly Asp Lys Pro Met Ser Leu Ser Met Ser 1 5 10 15
His Asp Gly Ser Ser Ile Arg Ser Ala Ala Ser Gln Phe Leu Ser Val 20 25 30
Pro Leu Ser His Ser Thr Pro Ile Pro Pro Gln Arg Lys Thr Val Leu 35 40 45
Leu Lys Phe Met Ile Gly Asp Glu Leu Ile Thr Val Gln Gly Ala Leu 50 55 60
Ala Pro Phe Asp Glu Tyr Trp Tyr Asp Asn Gln Pro Leu Leu Ala Gln 65 70 75 80
Ala Val Glu Met Leu Ala Ser Ala Asp Arg Leu Arg Gln Phe Glu His 85 90 95
Tyr Glu Lys Phe Leu Leu Lys Lys Gly His Gln Ile Thr Glu Ile Met 100 105 110
Asn Arg Leu Arg Leu Phe Phe Thr Asp Val Leu Lys Val Lys Met Glu 115 120 125
Ala Asp Ala Leu Pro Ala Leu Ala Gln Tyr Leu Met Val Gly Thr Leu
130 135 140
Glu Ala Val Ser Thr Ala Asp Ser Pro Asp Ala Cys Ala Pro Val Thr 145 150 155 160
Ser Lys Ile Leu Ala Lys Gln Gln Thr Ile Ala Lys Ser Pro Gly Arg 165 170 175
Leu Asp Glu Glu Glu Tyr Asn Val Ile Arg Ser Arg Phe Leu Thr His 180 185 190
Glu Val Phe Asp Leu Thr Ser Asp Leu Pro Gly Val Gln Pro Phe Met 195 200 205
Asp Met Tyr Tyr Ala Thr Val Pro Arg Ala Asp Ser Thr Gly Trp Cys 210 215 220
Val Tyr Arg Arg Lys Gly Leu Leu Ile Tyr Ala Pro Asp Glu Gln Phe 225 230 235 240
Ser Asp Leu Thr Ile Phe Ser Thr Arg Leu Thr Ala Ser Arg Glu Leu 245 250 255
Gln Leu Val Ala Gly Asp Val Val Val Ala Cys Phe Asp Leu Met Asp 260 265 270
Val Ser Asp Ile Ala Pro Ser His His Ala Ser Val Gln Glu Glu Arg 275 280 285
Thr Leu Gly Thr Ser Lys Tyr Ser Asn Val Thr Ala Asn Asp His Pro 290 295 300
Leu Val Phe Phe Ser Pro Ser Ala Leu Arg Trp Ala Ile Asp His Ala 305 310 315 320
Cys Thr Asp Ser Leu Val Ser Thr Arg Asn Ile Arg Val Cys Val Gly 325 330 335
Ile Asp Pro Leu Val Thr Arg Trp Thr Arg Asp Gly Val Gln Glu Ala 340 345 350
Ala Ile Leu Met Asp Asp Lys Leu Pro Ser Ala Gly Arg Ala Arg Met 355 360 365
Ala Leu Arg Thr Leu Leu Leu Ala Arg Arg Ser Pro Met Pro Ser Phe 370 375 380
Leu Leu Gly Ala Leu Lys Gln Ser Gly Gly Gln Leu Leu Glu His Tyr 385 390 395 400
Arg Cys Asp Ala Ala Asn Arg Tyr Gly Ser Pro Thr Val Pro Ile Ser 405 410 415
His Pro Pro Pro Cys Ser Lys Cys Pro Glu Leu Lys Glu Gln Ile Ala 420 425 430
Lys Leu Ser Ser Ser Pro Ile Pro Lys Val Asp Ser Ser Val Gly Pro 435 440 445
Ala Ala Leu Leu Ser Lys Ile Ala Asp Leu Gln Arg Ala Asn Arg Glu 450 455 460
Leu Ser Leu Lys Leu Val Asp Val Gln Pro Ala Arg Glu Asp His Leu 465 470 475 480
Leu Ala Tyr Leu Asn Glu His Val Cys Val Asn Ala Lys Asp His Glu 485 490 495
Lys Gly Leu Leu Ala Arg Cys Asn Val Ser Gly Asp Ser Ile Ser Ser 500 505 510
Ile Leu Gly Gln Arg Met Lys Asn Arg Glu Arg Phe Glu Thr Arg Leu 515 520 525
Arg His Glu Ala Ser Ala Glu Trp Glu Pro Arg Val Glu Ala Leu Asn 530 535 540
Gln Glu Leu Ala Lys Ala Arg Val Glu Gln Gln Asp Met Met Thr Gln 545 550 555 560
Ser Leu Gln Tyr Leu Asn Glu Arg Asp Glu Leu Leu Gln Glu Val Asp 565 570 575
Glu Leu Lys Arg Glu Leu Thr Thr Leu Arg Ser Ala Asn Val Arg Leu 580 585 590
Asn Ala Asp Asn His Arg Met Ser Arg Ala Thr Arg Val Gly Asp Ala 595 600 605
Phe Val Ser Asp Val Glu Pro Leu Pro Ser Gly Ile Pro Gly Glu Ser 610 615 620
Lys Pro Ser Met Glu Glu Leu Val Asp Asp Leu 625 630 635
<210> 10
<211> 721
<212> PRT
<213> Orthoreovirus mamífero
<400> 10
Met Ala Ser Phe Lys Gly Phe Ser Ala Asn Thr Val Pro Val Ser Lys 1 5 10 15
Ala Lys Arg Asp Ile Ser Ser Leu Ala Ala Thr Pro Gly Leu Arg Ser 20 25 30
Gln Ser Phe Thr Pro Ser Val Asp Met Ser Gln Ser Arg Glu Phe Leu 35 40 45
Thr Lys Ala Ile Glu Gln Gly Ser Met Ser Ile Pro Tyr Gln His Val 50 55 60
Asn Val Pro Lys Val Asp Arg Lys Val Val Ser Leu Val Val Arg Pro 65 70 75 80
Phe Ser Ser Gly Ala Phe Ser Ile Ser Gly Val Ile Ser Pro Ala His 85 90 95
Ala Tyr Leu Leu Glu Cys Leu Pro Gln Leu Glu Gln Ala Met Ala Phe 100 105 110
Val Ala Ser Pro Glu Ser Phe Gln Ala Ser Asp Val Ala Lys Arg Phe 115 120 125
Ala Ile Lys Pro Gly Met Ser Leu Gln Asp Ala Ile Thr Ala Phe Ile 130 135 140
Asn Phe Val Ser Ala Met Leu Lys Met Thr Val Thr Arg Gln Asn Phe 145 150 155 160
Asp Val Ile Val Ala Glu Ile Glu Arg Leu Ala Ser Thr Ser Val Ser 165 170 175
Val Arg Thr Lys Glu Ala Lys Val Ala Asp Glu Glu Leu Met Leu Phe 180 185 190
Gly Leu Asp His Arg Gly Pro Gln Gln Leu Asp Val Ser Asp Ala Lys
195 200 205
Gly Ile Met Lys Ala Ala Asp Ile Gln Thr Thr His Asp Val His Leu 210 215 220
Ala Pro Gly Val Gly Asn Ile Asp Pro Glu Ile Tyr Asn Glu Gly Arg 225 230 235 240
Phe Met Phe Met Gln His Lys Pro Leu Ala Ala Asp Gln Ser Tyr Phe 245 250 255
Thr Leu Glu Thr Ala Asp Tyr Phe Lys Ile Tyr Pro Thr Tyr Asp Glu 260 265 270
His Asp Gly Arg Met Ala Asp Gln Lys Gln Ser Gly Leu Ile Leu Cys 275 280 285
Thr Lys Asp Glu Val Leu Ala Glu Gln Thr Ile Phe Lys Leu Asp Ala 290 295 300
Pro Asp Asp Lys Thr Val His Leu Leu Asp Arg Asp Asp Asp His Val 305 310 315 320
Val Ala Arg Phe Thr Lys Val Phe Ile Glu Asp Val Ala Pro Gly His 325 330 335
His Ala Ala Gln Arg Ser Gly Gln Arg Ser Val Leu Asp Asp Leu Tyr 340 345 350
Ala Asn Thr Gln Val Ile Ser Ile Thr Ser Ala Ala Leu Lys Trp Val 355 360 365
Val Lys His Gly Val Ser Asp Gly Ile Val Asn Arg Lys Asn Val Lys 370 375 380
Val Cys Val Gly Phe Asp Pro Leu Tyr Thr Leu Ser Thr His Asn Gly 385 390 395 400
Val Ser Leu Cys Ala Leu Leu Met Asp Glu Lys Leu Ser Val Leu Asn 405 410 415
Ser Ala Cys Arg Met Thr Leu Arg Ser Leu Met Lys Thr Gly Arg Asp 420 425 430
Val Asp Ala His Arg Ala Phe Gln Arg Val Leu Ser Gln Gly Tyr Thr 435 440 445
Ser Leu Met Cys Tyr Tyr His Pro Ser Arg Lys Leu Ala Tyr Gly Glu 450 455 460
Val Leu Phe Leu Glu Arg Ser Asn Asp Val Thr Asp Gly Ile Lys Leu 465 470 475 480
Gln Leu Asp Ala Ser Arg Gln Cys His Glu Cys Pro Val Leu Gln Gln 485 490 495
Lys Val Val Glu Leu Glu Lys Gln Ile Ile Met Gln Lys Ser Ile Gln 500 505 510
Ser Asp Pro Thr Pro Val Ala Leu Gln Pro Leu Leu Ser Gln Leu Arg 515 520 525
Glu Leu Ser Ser Glu Val Thr Arg Leu Gln Met Glu Leu Ser Arg Ala 530 535 540
Gln Ser Leu Asn Ala Gln Leu Glu Ala Asp Val Lys Ser Ala Gln Ser 545 550 555 560
Cys Ser Leu Asp Met Tyr Leu Arg His His Thr Cys Ile Asn Gly His 565 570 575
Ala Lys Glu Asp Glu Leu Leu Asp Ala Val Arg Val Ala Pro Asp Val 580 585 590
Arg Arg Glu Ile Met Glu Lys Arg Ser Glu Val Arg Gln Gly Trp Cys 595 600 605
Glu Arg Ile Ser Lys Glu Ala Ala Ala Lys Cys Gln Thr Val Ile Asp 610 615 620
Asp Leu Thr Leu Met Asn Gly Lys Gln Ala Gln Glu Ile Thr Glu Leu 625 630 635 640
Arg Asp Ser Ala Glu Lys Tyr Glu Lys Gln Ile Ala Glu Leu Val Ser 645 650 655
Thr Ile Thr Gln Asn Gln Ile Thr Tyr Gln Gln Glu Leu Gln Ala Leu 660 665 670
Val Ala Lys Asn Val Glu Leu Asp Ala Leu Asn Gln Arg Gln Ala Lys 675 680 685
Ser Leu Arg Ile Thr Pro Ser Leu Leu Ser Ala Thr Pro Ile Asp Ser 690 695 700
Ala Asp Gly Val Ala Asp Leu Ile Asp Phe Ser Val Pro Thr Asp Glu 705 710 715 720
Leu
<210> 11
<211> 188
<212> PRT
<213> Orthoreovirus aviar
<400> 11
Pro Ala Val Leu Leu Ser Lys Ile Ala Asp Leu Gln Arg Ala Asn Arg 1 5 10 15
Glu Leu Ser Leu Lys Leu Val Asp Val Gln Pro Ala Arg Glu Asp His 20 25 30
Leu Leu Ala Tyr Leu Asn Glu His Val Cys Val Asn Ala Lys Asp His 35 40 45
Glu Lys Gly Leu Leu Ala Arg Cys Asn Val Ser Gly Asp Ser Ile Ser 50 55 60
Ser Ile Leu Gly Gln Arg Met Lys Asn Arg Glu Arg Phe Glu Thr Arg 65 70 75 80
Leu Arg His Glu Ala Ser Ala Glu Trp Glu Pro Arg Val Glu Ala Leu 85 90 95
Asn Gln Glu Leu Ala Lys Ala Arg Val Glu Gln Gln Asp Met Met Thr 100 105 110
Gln Ser Leu Gln Tyr Leu Asn Glu Arg Asp Glu Leu Leu Gln Glu Val 115 120 125
Asp Glu Leu Lys Arg Glu Leu Thr Thr Leu Arg Ser Ala Asn Val Arg 130 135 140
Leu Asn Ala Asp Asn His Arg Met Ser Arg Ala Thr Arg Val Gly Asp 145 150 155 160
Ala Phe Val Ser Asp Val Glu Pro Leu Pro Ser Gly Ile Pro Gly Glu
165 170 175
Ser Lys Pro Ser Met Glu Glu Leu Val Asp Asp Leu 180 185
<210> 12
<211> 251
<212> PRT
<213> Orthoreovirus mamífero
<400> 12
Ser Asn Asp Val Thr Asp Gly Ile Lys Leu Gln Leu Asp Ala Ser Arg 1 5 10 15
Gln Cys His Glu Cys Pro Val Leu Gln Gln Lys Val Val Glu Leu Glu 20 25 30
Lys Gln Ile Ile Met Gln Lys Ser Ile Gln Ser Asp Pro Thr Pro Val 35 40 45
Ala Leu Gln Pro Leu Leu Ser Gln Leu Arg Glu Leu Ser Ser Glu Val 50 55 60
Thr Arg Leu Gln Met Glu Leu Ser Arg Ala Gln Ser Leu Asn Ala Gln 65 70 75 80
Leu Glu Ala Asp Val Lys Ser Ala Gln Ser Cys Ser Leu Asp Met Tyr 85 90 95
Leu Arg His His Thr Cys Ile Asn Gly His Ala Lys Glu Asp Glu Leu 100 105 110
Leu Asp Ala Val Arg Val Ala Pro Asp Val Arg Arg Glu Ile Met Glu 115 120 125
Lys Arg Ser Glu Val Arg Gln Gly Trp Cys Glu Arg Ile Ser Lys Glu 130 135 140
Ala Ala Ala Lys Cys Gln Thr Val Ile Asp Asp Leu Thr Leu Met Asn 145 150 155 160
Gly Lys Gln Ala Gln Glu Ile Thr Glu Leu Arg Asp Ser Ala Glu Lys 165 170 175
Tyr Glu Lys Gln Ile Ala Glu Leu Val Ser Thr Ile Thr Gln Asn Gln 180 185 190
Ile Thr Tyr Gln Gln Glu Leu Gln Ala Leu Val Ala Lys Asn Val Glu 195 200 205
Leu Asp Ala Leu Asn Gln Arg Gln Ala Lys Ser Leu Arg Ile Thr Pro 210 215 220
Ser Leu Leu Ser Ala Thr Pro Ile Asp Ser Val Asp Asp Val Ala Asp 225 230 235 240
Leu Ile Asp Phe Ser Val Pro Thr Asp Glu Leu 245 250
<210> 13
<211> 188
<212> PRT
<213> Secuencia Artificial
<220>
<223> variante de la región mínima de la proteína muNS de Orthoreovirus aviar que tiene la capacidad de formar inclusiones cuando se expresa en una célula
<400> 13
Pro Ala Val Leu Leu Ser Lys Ile Ala Asp Leu Gln Arg Ala Asn Arg 1 5 10 15
Glu Leu Ser Leu Lys Leu Val Asp Val Gln Pro Ala Arg Glu Asp His 20 25 30
Leu Leu Ala Tyr Leu Asn Glu His Val Cys Val Asn Ala Lys Asp His 35 40 45
Glu Lys Gly Leu Leu Ala Arg Cys Ser Val Ser Gly Asp Ser Ile Ser 50 55 60
Ser Ile Leu Gly Gln Arg Met Lys Asn Arg Glu Arg Phe Glu Thr Arg 65 70 75 80
Leu Arg His Glu Ala Ser Ala Glu Trp Glu Pro Arg Val Glu Ala Leu 85 90 95
Asn Gln Glu Leu Ala Lys Ala Arg Val Glu Gln Gln Asp Met Met Thr 100 105 110
Gln Ser Leu Gln Tyr Leu Asn Glu Arg Asp Glu Leu Leu Gln Glu Val 115 120 125
Asp Glu Leu Lys Arg Glu Leu Thr Thr Leu Arg Ser Ala Asn Val Arg 130 135 140
Leu Asn Ala Asp Asn His Arg Met Ser Arg Ala Thr Arg Val Gly Asp 145 150 155 160
Ala Phe Val Ser Asp Val Glu Pro Leu Pro Ser Gly Ile Pro Gly Glu 165 170 175
Ser Lys Pro Ser Met Glu Glu Leu Val Asp Asp Leu 180 185
<210> 14
<211> 251
<212> PRT
<213> Secuencia Artificial
<220>
<223> variante de la región mínima de la proteína muNS de Orthoreovirus mamífero que tiene la capacidad de formar inclusiones cuando se expresa en una célula
<400> 14
Ser Asn Asp Val Thr Asp Gly Ile Lys Leu Gln Leu Asp Ala Ser Arg 1 5 10 15
Gln Cys His Glu Cys Pro Val Leu Gln Gln Lys Val Val Glu Leu Glu 20 25 30
Lys Gln Ile Ile Met Gln Lys Ser Ile Gln Ser Asp Pro Thr Pro Val 35 40 45
Ala Leu Gln Pro Leu Leu Ser Gln Leu Arg Glu Leu Ser Ser Glu Val 50 55 60
Thr Arg Leu Gln Met Glu Leu Ser Arg Ala Gln Ser Leu Asn Ala Gln 65 70 75 80
Leu Glu Ala Asp Val Lys Ser Ala Gln Ser Cys Ser Leu Asp Met Tyr 85 90 95
Leu Arg His His Thr Cys Ile Ser Gly His Ala Lys Glu Asp Glu Leu 100 105 110
Leu Asp Ala Val Arg Val Ala Pro Asp Val Arg Arg Glu Ile Met Glu 115 120 125
Lys Arg Ser Glu Val Arg Gln Gly Trp Cys Glu Arg Ile Ser Lys Glu
130 135 140
Ala Ala Ala Lys Cys Gln Thr Val Ile Asp Asp Leu Thr Leu Met Asn 145 150 155 160
Gly Lys Gln Ala Gln Glu Ile Thr Glu Leu Arg Asp Ser Ala Glu Lys 165 170 175
Tyr Glu Lys Gln Ile Ala Glu Leu Val Ser Thr Ile Thr Gln Asn Gln 180 185 190
Ile Thr Tyr Gln Gln Glu Leu Gln Ala Leu Val Ala Lys Asn Val Glu 195 200 205
Leu Asp Ala Leu Asn Gln Arg Gln Ala Lys Ser Leu Arg Ile Thr Pro 210 215 220
Ser Leu Leu Ser Ala Thr Pro Ile Asp Ser Val Asp Asp Val Ala Asp 225 230 235 240
Leu Ile Asp Phe Ser Val Pro Thr Asp Glu Leu 245 250
<210> 15
<211> 251
<212> PRT
<213> Secuencia Artificial
<220>
<223> variante de la región mínima de la proteína muNS de Orthoreovirus mamífero que tiene la capacidad de formar inclusiones cuando se expresa en una célula
<400> 15
Ser Asn Asp Val Thr Asp Gly Ile Lys Leu Gln Leu Asp Ala Ser Arg 1 5 10 15
Gln Cys His Glu Cys Pro Val Leu Gln Gln Lys Val Val Glu Leu Glu 20 25 30
Lys Gln Ile Ile Met Gln Lys Ser Ile Gln Ser Asp Pro Thr Pro Val 35 40 45
Ala Leu Gln Pro Leu Leu Ser Gln Leu Arg Glu Leu Ser Ser Glu Val 50 55 60
Thr Arg Leu Gln Met Glu Leu Ser Arg Ala Gln Ser Leu Asn Ala Gln 65 70 75 80
Leu Glu Ala Asp Val Lys Ser Ala Gln Ser Cys Ser Leu Asp Met Tyr 85 90 95
Leu Arg His His Thr Cys Ile Asn Gly His Ala Lys Glu Asp Glu Leu 100 105 110
Leu Asp Ala Val Arg Val Ala Pro Asp Val Arg Arg Glu Ile Met Glu 115 120 125
Lys Arg Ser Glu Val Arg Gln Gly Trp Cys Glu Arg Ile Ser Lys Glu 130 135 140
Ala Ala Ala Lys Cys Gln Thr Val Ile Asp Asp Leu Thr Leu Met Ser 145 150 155 160
Gly Lys Gln Ala Gln Glu Ile Thr Glu Leu Arg Asp Ser Ala Glu Lys 165 170 175
Tyr Glu Lys Gln Ile Ala Glu Leu Val Ser Thr Ile Thr Gln Asn Gln 180 185 190
Ile Thr Tyr Gln Gln Glu Leu Gln Ala Leu Val Ala Lys Asn Val Glu 195 200 205
Leu Asp Ala Leu Asn Gln Arg Gln Ala Lys Ser Leu Arg Ile Thr Pro 210 215 220
Ser Leu Leu Ser Ala Thr Pro Ile Asp Ser Val Asp Asp Val Ala Asp 225 230 235 240
Leu Ile Asp Phe Ser Val Pro Thr Asp Glu Leu 245 250
<210> 16
<211> 251
<212> PRT
<213> Secuencia Artificial
<220>
<223> variante de la región mínima de la proteína muNS de Orthoreovirus mamífero que tiene la capacidad de formar inclusiones cuando se expresa en una célula
<400> 16
Ser Asn Asp Val Thr Asp Gly Ile Lys Leu Gln Leu Asp Ala Ser Arg 1 5 10 15
Gln Cys His Glu Cys Pro Val Leu Gln Gln Lys Val Val Glu Leu Glu 20 25 30
Lys Gln Ile Ile Met Gln Lys Ser Ile Gln Ser Asp Pro Thr Pro Val 35 40 45
Ala Leu Gln Pro Leu Leu Ser Gln Leu Arg Glu Leu Ser Ser Glu Val 50 55 60
Thr Arg Leu Gln Met Glu Leu Ser Arg Ala Gln Ser Leu Asn Ala Gln 65 70 75 80
Leu Glu Ala Asp Val Lys Ser Ala Gln Ser Cys Ser Leu Asp Met Tyr 85 90 95
Leu Arg His His Thr Cys Ile Ser Gly His Ala Lys Glu Asp Glu Leu 100 105 110
Leu Asp Ala Val Arg Val Ala Pro Asp Val Arg Arg Glu Ile Met Glu 115 120 125
Lys Arg Ser Glu Val Arg Gln Gly Trp Cys Glu Arg Ile Ser Lys Glu 130 135 140
Ala Ala Ala Lys Cys Gln Thr Val Ile Asp Asp Leu Thr Leu Met Ser 145 150 155 160
Gly Lys Gln Ala Gln Glu Ile Thr Glu Leu Arg Asp Ser Ala Glu Lys 165 170 175
Tyr Glu Lys Gln Ile Ala Glu Leu Val Ser Thr Ile Thr Gln Asn Gln 180 185 190
Ile Thr Tyr Gln Gln Glu Leu Gln Ala Leu Val Ala Lys Asn Val Glu 195 200 205
Leu Asp Ala Leu Asn Gln Arg Gln Ala Lys Ser Leu Arg Ile Thr Pro 210 215 220
Ser Leu Leu Ser Ala Thr Pro Ile Asp Ser Val Asp Asp Val Ala Asp 225 230 235 240
Leu Ile Asp Phe Ser Val Pro Thr Asp Glu Leu 245 250
<210> 17
<211> 5
<212> PRT
<213> Secuencia Artificial
<220>
<223> sitio de corte de enteroquinasa
<400> 17
Asp Asp Asp Asp Lys 1 5
<210> 18
<211> 5
<212> PRT
<213> Secuencia Artificial
<220>
<223> sitio de corte de factor Xa
<400> 18
Ile Glu Asp Gly Arg 1 5
<210> 19
<211> 6
<212> PRT
<213> Secuencia Artificial
<220>
<223> sitio de corte de trombina
<400> 19
Leu Val Pro Arg Gly Ser 1 5
<210> 20
<211> 7
<212> PRT
<213> Secuencia Artificial
<220>
<223> sitio de corte de proteasa TEV
<400> 20
Glu Asn Leu Tyr Phe Gln Gly 1 5
<210> 21
<211> 68
<212> PRT
<213> Orthoreovirus aviar
<400> 21
Met Pro Ser Phe Leu Leu Gly Ala Leu Lys Gln Ser Gly Gly Gln Leu 1 5 10 15
Leu Glu His Tyr Arg Cys Asp Ala Ala Asn Arg Tyr Gly Ser Pro Thr 20 25 30
Val Pro Ile Ser His Pro Pro Pro Cys Ser Lys Cys Pro Glu Leu Lys 35 40 45
Glu Gln Ile Ala Lys Leu Ser Ser Ser Pro Ile Pro Lys Val Asp Ser 50 55 60
Ser Val Gly Pro 65
<210> 22
<211> 30
<212> PRT
<213> Orthoreovirus aviar
<400> 22
Pro Ala Ala Leu Leu Ser Lys Ile Ala Asp Leu Gln Arg Ala Asn Arg 1 5 10 15
Glu Leu Ser Leu Lys Leu Val Asp Val Gln Pro Ala Arg Glu 20 25 30
<210> 23
<211> 66
<212> PRT
<213> Orthoreovirus aviar
<400> 23
Glu Asp His Leu Leu Ala Tyr Leu Asn Glu His Val Cys Val Asn Ala 1 5 10 15
Lys Asp His Glu Lys Gly Leu Leu Ala Arg Cys Asn Val Ser Gly Asp 20 25 30
Ser Ile Ser Ser Ile Leu Gly Gln Arg Met Lys Asn Arg Glu Arg Phe 35 40 45
Glu Thr Arg Leu Arg His Glu Ala Ser Ala Glu Trp Glu Pro Arg Val 50 55 60
Glu Ala
<210> 24
<211> 67
<212> PRT
<213> Orthoreovirus aviar
<400> 24
Arg Val Glu Ala Leu Asn Gln Glu Leu Ala Lys Ala Arg Val Glu Gln 1 5 10 15
Gln Asp Met Met Thr Gln Ser Leu Gln Tyr Leu Asn Glu Arg Asp Glu 20 25 30
Leu Leu Gln Glu Val Asp Glu Leu Lys Arg Glu Leu Thr Thr Leu Arg 35 40 45
Ser Ala Asn Val Arg Leu Asn Ala Asp Asn His Arg Met Ser Arg Ala 50 55 60
Thr Arg Val 65
<210> 25
<211> 66
<212> PRT
<213> Secuencia Artificial
<220>
<223> variante de la región mínima de la proteína muNS de un Orthoreovirus con capacidad de incorporarse a inclusiones formadas por una proteína que comprende muNS-Mi de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando
<220>
<221> SITE
<222> (28) .. (28)
<223> cualquier aminoácido excepto Asn
<400> 25
Glu Asp His Leu Leu Ala Tyr Leu Asn Glu His Val Cys Val Asn Ala 1 5 10 15
Lys Asp His Glu Lys Gly Leu Leu Ala Arg Cys Xaa Val Ser Gly Asp 20 25 30
Ser Ile Ser Ser Ile Leu Gly Gln Arg Met Lys Asn Arg Glu Arg Phe 35 40 45
Glu Thr Arg Leu Arg His Glu Ala Ser Ala Glu Trp Glu Pro Arg Val 50 55 60
Glu Ala 65
<210> 26
<211> 66
<212> PRT
<213> Secuencia Artificial
<220>
<223> variante de la región mínima de la proteína muNS de un Orthoreovirus con capacidad de incorporarse a inclusiones formadas por una proteína que comprende muNS-Mi de un Orthoreovirus que tiene la capacidad de formar inclusiones cuando
<400> 26
Glu Asp His Leu Leu Ala Tyr Leu Asn Glu His Val Cys Val Asn Ala 1 5 10 15
Lys Asp His Glu Lys Gly Leu Leu Ala Arg Cys Ser Val Ser Gly Asp 20 25 30
Ser Ile Ser Ser Ile Leu Gly Gln Arg Met Lys Asn Arg Glu Arg Phe 35 40 45
Glu Thr Arg Leu Arg His Glu Ala Ser Ala Glu Trp Glu Pro Arg Val 50 55 60
Glu Ala 65
<210> 27
<211> 31
<212> DNA
<213> Secuencia Artificial
<220>
<223> cebador Forward
<400> 27 ttggcgcgca aatgccagcc gtactgctgt c 31
<210> 28
<211> 28
<212> DNA
<213> Secuencia Artificial
<220>
<223> cebador Reverse
<400> 28 ttgcggccgc aatcacagat catccacc 28
<210> <211> <212> <213> 29 43 DNA Secuencia Artificial
<220> <223> oligonucleótido para efectuar mutagénesis
<400> 29 gggcctgctc gctcgttgta gcgtatctgg tgattcaatc tcc 43
<210> <211> <212> <213> 30 43 DNA Secuencia Artificial
<220> <223> oligonucleótido para efectuar mutagénesis
<400> 30 ggagattgaa tcaccagata cgctacaacg agcgagcagg ccc 43
<210> <211> <212> <213> 31 31 DNA Secuencia Artificial
<220> <223> cebador Forward_VSV
<400> 31 cggctagcat gaagtgcctt ttgtacttag c 31
<210> <211> <212> <213> 32 34 DNA Secuencia Artificial
<220> <223> cebador Reverse_VSV
<400> 32 cccaagcttg ggagagctct tccaactact gaac 34
<210> <211> <212> <213> 33 64 DNA Secuencia Artificial
<220> <223> cebador para añadir adaptador
<400> 33 gccggaattc cgaaggcaaa ccaatcccaa acccactgct gggcctggat ttgcggccgc aaat 60 64
<210> <211> <212> <213> 34 64 DNA Secuencia Artificial
<220> <223> cebador para añadir adaptador <400> 34 atttgcggcc gcaaatccag gcccagcagt gggtttggga ttggtttgcc ttcggaattc 60
cggc 64
<210> <211> <212> <213> 35 8 PRT Secuencia Artificial
<220> <223> <400> sitio de corte de proteasa PreScission 35
Leu Glu Val Leu Phe Gln Gly Pro1 5
<210> <211> <212> <213> 36 34 DNA Secuencia Artificial
<220> <223> cebador Forward IC
<400> 36 ttgcggccgc aagaagatca cttgttggct tatc 34
<210> <211> <212> <213> 37 30 DNA Secuencia Artificial
<220> <223> cebador Reverse IC
<400> 37 gcgtctagat tacgcttcca cacggggttc 30
Patentes similares o relacionadas:
Cadena ligera de enteroquinasa modificada, del 22 de Julio de 2020, de NOVO NORDISK A/S: Un análogo de la cadena ligera de la enteroquinasa bovina que comprende una secuencia de aminoácidos establecida en la SEQ ID NO: 1, en donde dicho análogo comprende […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos y composiciones para ingeniería genómica, del 3 de Junio de 2020, de Sangamo Therapeutics, Inc: Una pareja de nucleasas de dedo de zinc (ZFN) que comprende una ZFN izquierda y una ZFN derecha, comprendiendo cada ZFN un dominio de escisión […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Antígenos de coagulasa estafilocócica y métodos para su uso, del 13 de Mayo de 2020, de UNIVERSITY OF CHICAGO: Una composición inmunógena que comprende al menos dos dominios 1-2 de coagulasa estafilocócica diferentes, en donde cada uno de los al menos dos dominios […]
Reconocimiento de unión a diana celular mediante un agente bioactivo usando transferencia de energía de resonancia de bioluminiscencia intracelular, del 6 de Mayo de 2020, de PROMEGA CORPORATION: Un sistema de ensayo que comprende: (a) una biblioteca de agentes bioactivos, cada uno de los cuales está fijado a un fluoróforo; (b) una diana celular fusionada a […]
Etiqueta de epítopo y método de detección, captura y/o purificación de polipéptidos etiquetados, del 15 de Abril de 2020, de ChromoTek GmbH: Péptido epítopo aislado que tiene de 12 a 25 aminoácidos, en donde la secuencia de aminoácidos comprende una secuencia según se define en SEQ ID NO: 32 (X1X2RX4X5AX7SX9WX11X12), […]
Utilización diagnóstica de un polipéptido de fusión que comprende una proteína vírica y un enzima MGMT, del 15 de Abril de 2020, de INSTITUT PASTEUR: Utilización in vitro de un polipéptido de fusión que comprende una proteína vírica y i) el enzima 6-metilguanina-ADN-metiltransferasa (MGMT, EC 2.1.1.63) o un homólogo […]