DETECCION DE LA UNION DE RECOMBINACION DE ADN.
Un método para determinar la presencia o ausencia de un polinucleótido de inserción integrado en un sitio de unión en una secuencia polinucleotídica diana,
comprendiendo el método, poner en contacto el polinucleótido diana con un primer cebador, un segundo cebador, un polinucleótido bloqueante, una polimerasa, y uno o más nucleótidos trifosfato, en el que:
(i) el polinucleótido diana comprende una cadena polinucleotídica que comprende un sitio de unión que, cuando un polinucleótido de inserción integrado está ausente, abarca en un lado una primera secuencia diana y en el otro lado una segunda secuencia diana que es contigua a la primera secuencia diana,
(ii) el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas cuando el polinucleótido de inserción integrado está ausente,
(iii) el primer cebador hibrida con una primera región de la primera secuencia diana que está próxima al sitio de unión, de manera que
(iv) el segundo cebador hibrida con una segunda región de la primera secuencia diana que está lejos del sitio de unión, en el que el segundo cebador es capaz de cebar la síntesis de una copia de la primera secuencia diana que comprende la primera y segunda regiones de la primera secuencia diana,
de manera que cuando una secuencia de inserción integrada está presente en el sitio de unión, el primer y segundo cebadores causan la amplificación exponencial de la primera y segunda regiones de la primera secuencia diana, y cuando una secuencia de inserción integrada está ausente del sitio de unión, el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas de manera que se inhibe la amplificación de la primera y segunda regiones de la primera secuencia diana, según lo cual se determina la presencia o ausencia del polinucleótido de inserción integrado en el sitio de unión en el polinucleótido diana
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2007/011523.
Solicitante: CEPHEID.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 904 CARIBBEAN DRIVE,SUNNYVALE, CA 94089.
Inventor/es: ZHANG,JIAN PING, FRIEDLANDER,ERNEST JAY, RUHFEL,ROBERT, SWENSON,DAVID DOUGLAS, WORTMAN,ALAN THOMAS.
Fecha de Publicación: .
Fecha Concesión Europea: 12 de Mayo de 2010.
Clasificación Internacional de Patentes:
- C12Q1/68D2G
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
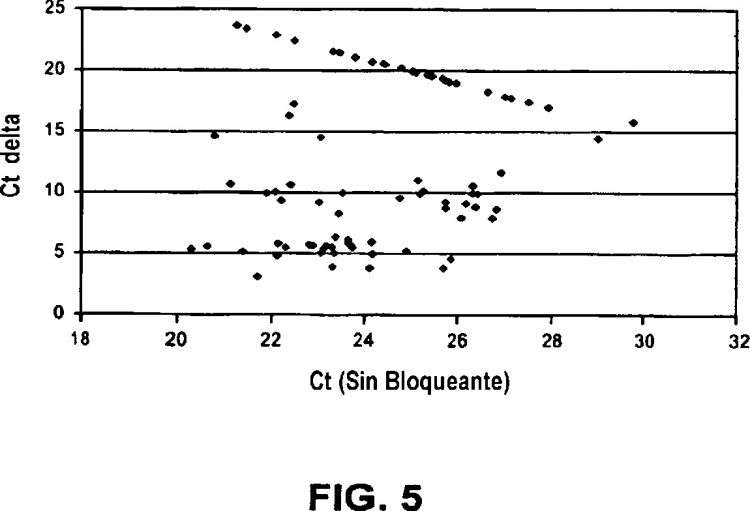
Fragmento de la descripción:
Detección de la unión de recombinación de ADN.
Antecedentes de la invención
La detección de secuencias de inserción de nucleótidos integradas es importante en muchos contextos. Por ejemplo, las secuencias de inserción de nucleótidos pueden transportar oncogenes en mamíferos, genes de resistencia a antibióticos en bacterias y genes que expresan rasgos identificables en plantas. La resistencia a antibióticos bacteriana es un problema clínico importante a nivel mundial y una preocupación de salud pública (véanse, por ejemplo, Sheldon, Clin Lab Sci (2005) 18:170 y French, Adv Drug Deliv Rev (2005) 57:1514). Claramente, un método eficaz, sensible y fiable para detectar la presencia o ausencia de un nucleótido de inserción integrado es valioso en el diagnóstico clínico y en otros contextos.
Otros han desarrollado métodos para detectar secuencias de inserción de nucleótidos integradas. En una estrategia, el nucleótido de inserción integrado se detecta usando PCR en la que un cebador hibrida con la secuencia de ácido nucleico diana y el otro cebador hibrida con el nucleótido de inserción. La detección positiva del amplicón indica la presencia de un polinucleótido de inserción. Este método puede resultar en falsos negativos, o nucleótidos de inserción no detectados, ya que las secuencias de los nucleótidos de inserción son a menudo variables. Los cebadores disponibles pueden o no hibridar con el polinucleótido de inserción. Véase, por ejemplo, el Ensayo IDI-MRSATM de GeneOhm Sciences, San Diego, CA.
En otra estrategia, el nucleótido de inserción integrado se detecta usando PCR en la que los dos cebadores hibridan con el polinucleótido de inserción. De nuevo, la detección positiva del amplicón indica la presencia de un polinucleótido de inserción integrado. Este método también tiene la desventaja de que puede resultar en falsos negativos, debido a la naturaleza polimórfica de los nucleótidos de inserción integrados. Además, no está claro si el polinucleótido de inserción está integrado en la secuencia de ácido nucleico diana cuando hibridan con el polinucleótido de inserción cebadores directos e inversos. Véanse, por ejemplo, Kreiswirth, et al., J Clin Microbiol (2005) 43:4585 y el Kit de Detección LightCycler® MRSA de Roche Diagnostics, Alameda, CA.
En una estrategia más, la presencia o ausencia del polinucleótido de inserción integrado se identifica usando PCR en la que un cebador hibrida con la secuencia de ácido nucleico diana y el otro cebador hibrida con una secuencia que se extiende a ambos lados del sitio de integración entre la secuencia de ácido nucleico diana y el polinucleótido de inserción. Aquí, la detección negativa (ausencia de amplificación) del amplicón indica la presencia de un polinucleótido de inserción integrado. Este método tiene la desventaja de que una señal negativa indica la integración positiva de un polinucleótido de inserción (véase, Patente de EEUU No. 6.156.507).
Todavía existe la necesidad de métodos eficaces, sensibles para detectar polinucleótidos de inserción integrados que proporcionen una señal positiva indicativa de la integración del polinucleótido de inserción.
Resumen breve de la invención
La presente invención proporciona composiciones (p. ej., disoluciones, mezclas de reacción), métodos y kits para detectar la presencia o ausencia de un polinucleótido de inserción en un ácido nucleico. Las composiciones, métodos y kits encuentran un uso en la detección, por ejemplo, de polinucleótidos de inserción integrados que confieren resistencia en bacterias a antibióticos.
Respecto a los métodos, la invención proporciona métodos para determinar la presencia o ausencia de un polinucleótido de inserción integrado en un sitio de unión en una secuencia de polinucleótido diana. En algunas realizaciones, los métodos comprenden poner en contacto el polinucleótido diana con un primer cebador, un segundo cebador, un polinucleótido bloqueante, una polimerasa, y uno o más nucleótidos trifosfato, en el que:
(i) el polinucleótido diana comprende una cadena polinucleotídica que comprende un sitio de unión que, cuando un polinucleótido de inserción integrado está ausente, abarca en un lado una primera secuencia diana y en el otro lado una segunda secuencia diana que es contigua a la primera secuencia diana,
(ii) el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas cuando el polinucleótido de inserción integrado está ausente,
(iii) el primer cebador hibrida con una primera región de la primera secuencia diana que está próxima al sitio de unión, de manera que
(iv) el segundo cebador hibrida con una segunda región de la primera secuencia diana que está lejos del sitio de unión, en el que el segundo cebador es capaz de cebar la síntesis de una copia de la primera secuencia diana que comprende la primera y segunda regiones de la primera secuencia diana,
de manera que cuando una secuencia de inserción integrada está presente en el sitio de unión, el primer y segundo cebadores causan la amplificación exponencial de la primera y segunda regiones de la primera secuencia diana, y cuando una secuencia de inserción integrada está ausente del sitio de unión, el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas de manera que se inhibe la amplificación de la primera y segunda regiones de la primera secuencia diana, según lo cual se determina la presencia o ausencia del polinucleótido de inserción integrado en el sitio de unión en el polinucleótido diana.
Respecto a las composiciones, la invención proporciona composiciones, incluyendo mezclas de reacción y disoluciones para determinar la presencia o ausencia de un polinucleótido de inserción integrado en un sitio de unión de una primera secuencia diana y una segunda secuencia diana en un polinucleótido diana. En algunas realizaciones, las composiciones comprenden el polinucleótido diana, un primer cebador, un segundo cebador y un polinucleótido bloqueante, en el que:
(i) el polinucleótido diana comprende una cadena polinucleotídica que comprende un sitio de unión que, cuando un polinucleótido de inserción integrado está ausente, abarca en un lado una primera secuencia diana y en el otro lado una segunda secuencia diana que es contigua a la primera secuencia diana,
(ii) el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas cuando el polinucleótido de inserción integrado está ausente,
(iii) el primer cebador hibrida con una primera región de la primera secuencia diana que está próxima al sitio de unión, de manera que
(iv) el segundo cebador hibrida con una segunda región de la primera secuencia diana que está lejos del sitio de unión, en el que el segundo cebador es capaz de cebar la síntesis de una copia de la primera secuencia diana que comprende la primera y segunda regiones de la primera secuencia diana,
de manera que cuando una secuencia de inserción integrada está presente en el sitio de unión, el primer y segundo cebadores causan la amplificación exponencial de la primera y segunda regiones de la primera secuencia diana, y cuando una secuencia de inserción integrada está ausente del sitio de unión, el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas de manera que se inhibe la amplificación de la primera y segunda regiones de la primera secuencia diana.
Respecto a los kits, la invención proporciona kits para determinar la presencia o ausencia de un polinucleótido de inserción integrado en un sitio de unión de una primera secuencia diana y una segunda secuencia diana en un polinucleótido diana. En algunas realizaciones, los kits comprenden un primer cebador, un segundo cebador y un polinucleótido bloqueante, en el que:
(i) el polinucleótido diana comprende una cadena polinucleotídica que comprende un sitio de unión que, cuando un polinucleótido de inserción integrado está ausente, abarca en un lado una primera secuencia diana y en el otro lado una segunda secuencia diana que es contigua a la primera secuencia diana,
(ii) el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas cuando el polinucleótido de inserción integrado está ausente,
(iii) el primer cebador hibrida con una primera región de la primera secuencia diana que...
Reivindicaciones:
1. Un método para determinar la presencia o ausencia de un polinucleótido de inserción integrado en un sitio de unión en una secuencia polinucleotídica diana, comprendiendo el método, poner en contacto el polinucleótido diana con un primer cebador, un segundo cebador, un polinucleótido bloqueante, una polimerasa, y uno o más nucleótidos trifosfato, en el que:
(i) el polinucleótido diana comprende una cadena polinucleotídica que comprende un sitio de unión que, cuando un polinucleótido de inserción integrado está ausente, abarca en un lado una primera secuencia diana y en el otro lado una segunda secuencia diana que es contigua a la primera secuencia diana,
(ii) el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas cuando el polinucleótido de inserción integrado está ausente,
(iii) el primer cebador hibrida con una primera región de la primera secuencia diana que está próxima al sitio de unión, de manera que
(iv) el segundo cebador hibrida con una segunda región de la primera secuencia diana que está lejos del sitio de unión, en el que el segundo cebador es capaz de cebar la síntesis de una copia de la primera secuencia diana que comprende la primera y segunda regiones de la primera secuencia diana,
de manera que cuando una secuencia de inserción integrada está presente en el sitio de unión, el primer y segundo cebadores causan la amplificación exponencial de la primera y segunda regiones de la primera secuencia diana, y cuando una secuencia de inserción integrada está ausente del sitio de unión, el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas de manera que se inhibe la amplificación de la primera y segunda regiones de la primera secuencia diana, según lo cual se determina la presencia o ausencia del polinucleótido de inserción integrado en el sitio de unión en el polinucleótido diana.
2. El método de la reivindicación 1, en el que la longitud completa del primer cebador hibrida con la primera secuencia diana de manera competitiva con el bloqueante, o en el que una parte del primer cebador hibrida con la primera secuencia diana de manera competitiva con el bloqueante.
3. El método de la reivindicación 1, en el que los nucleótidos trifosfato son nucleótidos dNTP, y/o en el que la polimerasa es una ADN polimerasa, y/o en el que la ADN polimerasa es una Taq polimerasa, y/o en el que el polinucleótido de inserción tiene una longitud de al menos 10 bases nucleotídicas.
4. El método de la reivindicación 1, (a) en el que el método se realiza en formato múltiple; y/o (b) que comprende además la etapa de exponer el polinucleótido diana a condiciones de amplificación.
5. El método de la reivindicación 4(b), en el que la amplificación se evalúa por PCR; o en el que la amplificación se evalúa por PCR en tiempo real.
6. Una mezcla de reacción para determinar la presencia o ausencia de un polinucleótido de inserción integrado en un sitio de unión de una primera secuencia diana y una segunda secuencia diana en un polinucleótido diana, comprendiendo la mezcla de reacción, el polinucleótido diana, un primer cebador, un segundo cebador y un polinucleótido bloqueante, en el que:
(i) el polinucleótido diana comprende una cadena polinucleotídica que comprende un sitio de unión que, cuando un polinucleótido de inserción integrado está ausente, abarca en un lado una primera secuencia diana y en el otro lado una segunda secuencia diana que es contigua a la primera secuencia diana,
(ii) el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas cuando el polinucleótido de inserción integrado está ausente,
(iii) el primer cebador hibrida con una primera región de la primera secuencia diana que está próxima al sitio de unión, de manera que
(iv) el segundo cebador hibrida con una segunda región de la primera secuencia diana que está lejos del sitio de unión, en el que el segundo cebador es capaz de cebar la síntesis de una copia de la primera secuencia diana que comprende la primera y segunda regiones de la primera secuencia diana,
de manera que cuando una secuencia de inserción integrada está presente en el sitio de unión, el primer y segundo cebadores causan la amplificación exponencial de la primera y segunda regiones de la primera secuencia diana, y cuando una secuencia de inserción integrada está ausente del sitio de unión, el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas de manera que se inhibe la amplificación de la primera y segunda regiones de la primera secuencia diana.
7. La mezcla de reacción de la reivindicación 6, en la que el polinucleótido de inserción se integra en el sitio de unión, o en la que el polinucleótido de inserción no se integra en el sitio de unión.
8. La mezcla de reacción de la reivindicación 7, que comprende además nucleótidos dNTP; y/o que comprende además una ADN polimerasa, opcionalmente en la que la ADN polimerasa es una Taq polimerasa; y/o que comprende además una sonda para detectar el amplicón amplificado por PCR en tiempo real, y/o que comprende una pluralidad de bloqueantes para detectar un polinucleótido de inserción integrado en una pluralidad de diferentes polinucleótidos diana.
9. Un kit para determinar la presencia o ausencia de un polinucleótido de inserción integrado en un sitio de unión de una primera secuencia diana y una segunda secuencia diana en un polinucleótido diana, comprendiendo el kit, un primer cebador, un segundo cebador y un polinucleótido bloqueante, en el que:
(i) el polinucleótido diana comprende una cadena polinucleotídica que comprende un sitio de unión que, cuando un polinucleótido de inserción integrado está ausente, abarca en un lado una primera secuencia diana y en el otro lado una segunda secuencia diana que es contigua a la primera secuencia diana,
(ii) el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas cuando el polinucleótido de inserción integrado está ausente,
(iii) el primer cebador hibrida con una primera región de la primera secuencia diana que está próxima al sitio de unión, de manera que
(iv) el segundo cebador hibrida con una segunda región de la primera secuencia diana que está lejos del sitio de unión, en el que el segundo cebador es capaz de cebar la síntesis de una copia de la primera secuencia diana que comprende la primera y segunda regiones de la primera secuencia diana,
de manera que cuando una secuencia de inserción integrada está presente en el sitio de unión, el primer y segundo cebadores causan la amplificación exponencial de la primera y segunda regiones de la primera secuencia diana, y cuando una secuencia de inserción integrada está ausente del sitio de unión, el polinucleótido bloqueante hibrida con la primera y segunda secuencias diana contiguas de manera que se inhibe la amplificación de la primera y segunda regiones de la primera secuencia diana.
10. El método de la reivindicación 1, mezcla de reacción de la reivindicación 7 o kit de la reivindicación 9, en el que el primer cebador y el polinucleótido bloqueante son los dos sustancialmente complementarios a la primera región en el polinucleótido diana y el primer cebador tiene menos nucleótidos con emparejamiento erróneo que el polinucleótido bloqueante respecto a la primera región en el polinucleótido diana; o en el que el primer cebador es completamente complementario a la primera región en el polinucleótido diana y el polinucleótido bloqueante porta al menos un emparejamiento erróneo interno comparado con la primera región del polinucleótido diana.
11. El método de la reivindicación 1, mezcla de reacción de la reivindicación 7 o kit de la reivindicación 9, en el que la primera secuencia diana y la segunda secuencia diana son partes de orfX de Staphylococcus aureus, el sitio de unión es un sitio de integración attB, el polinucleótido de inserción es al menos una parte de un complejo SCCmec, y el polinucleótido diana es ADN de Staphylococcus aureus.
12. El método de la reivindicación 1, mezcla de reacción de la reivindicación 7 o kit de la reivindicación 9, en el que el polinucleótido bloqueante comprende un resto en su extremo 3' seleccionado del grupo que consiste en fosfato y hexilamina, o en el que el polinucleótido bloqueante comprende al menos una base análoga de ácido nucleico.
13. El kit de la reivindicación 9, que comprende además un polinucleótido diana control que comprende la primera secuencia diana y segunda secuencia diana.
14. El kit de la reivindicación 13, en el que el polinucleótido de inserción se integra en el sitio de unión, o en el que el polinucleótido de inserción no se integra en el sitio de unión.
15. El kit de la reivindicación 13, que comprende además una sonda para detectar el amplicón amplificado por PCR en tiempo real, y/o que comprende además un sustrato multipocillo.
Patentes similares o relacionadas:
DETERMINACIÓN DEL GRADO DE METILACIÓN DE ADN, del 23 de Noviembre de 2011, de HEINRICH-HEINE-UNIVERSITÄT DÜSSELDORF: Procedimiento para determinar el grado de metilación de ADN normalizado que comprende las etapas: a) determinación cuantitativa de la presencia de un transposón […]
PROCEDIMIENTO DE OBTENCION DE UN DISPOSITIVO PARA DIAGNOSTICO DE MUTACIONES FRECUENTES DE LA PROTEINA LRRK2 BASADO EN PCR EN TIEMPO REAL, del 1 de Julio de 2011, de UNIVERSIDAD DE EXTREMADURA FUNDESALUD: La presente invención se refiere a un procedimiento de obtención de un dispositivo para diagnóstico de mutaciones frecuentes de la proteína Irrk2 […]
CONJUNTO DE CEBADORES, SONDAS, PROCEDIMIENTO Y KIT PARA EL GENOTIPADODEL POLIMORFISMO GENETICO -250G/A DEL GEN LIPC, del 24 de Junio de 2011, de UNIVERSIDAD DE MALAGA: Conjunto de cebadores, sondas, procedimiento y kit para el genotipado del polimorfismo genético -250G/A del gen LIPC.La presente invención se refiere a un conjunto de cebadores, […]
 AMPLIFICACIÓN DE FRAGMENTOS DE ADN, del 29 de Abril de 2011, de COMMONWEALTH SCIENTIFIC AND INDUSTRIAL RESEARCH ORGANISATION: Un procedimiento para la amplificación selectiva de una muestra de una molécula de ácido nucleico que tiene una secuencia diana adyacente […]
AMPLIFICACIÓN DE FRAGMENTOS DE ADN, del 29 de Abril de 2011, de COMMONWEALTH SCIENTIFIC AND INDUSTRIAL RESEARCH ORGANISATION: Un procedimiento para la amplificación selectiva de una muestra de una molécula de ácido nucleico que tiene una secuencia diana adyacente […]
 MÉTODOS PARA EL RÁPIDO ANÁLISIS FORENSE DE ADN MITOCONDRIAL Y CARATERIZACIÓN HETEROPLASMIA DE ADN MITOCONDRIAL, del 16 de Febrero de 2011, de IBIS BIOSCIENCES, INC: Un método forense que comprende los pasos de: (i) determinar una primera masa molecular de un primer producto de amplificación de un Cebador amplicón identificador […]
MÉTODOS PARA EL RÁPIDO ANÁLISIS FORENSE DE ADN MITOCONDRIAL Y CARATERIZACIÓN HETEROPLASMIA DE ADN MITOCONDRIAL, del 16 de Febrero de 2011, de IBIS BIOSCIENCES, INC: Un método forense que comprende los pasos de: (i) determinar una primera masa molecular de un primer producto de amplificación de un Cebador amplicón identificador […]
CONJUNTO DE CEBADORES, SONDAS, PROCEDIMIENTO Y KIT PARA EL GENOTIPADODEL POLIMORFISMO GENETICO S447X DEL GEN LPL, del 27 de Enero de 2011, de UNIVERSIDAD DE MALAGA: Conjunto de cebadores, sondas, procedimiento y kit para el genotipado del polimorfismo genético S447X del gen LPL.La presente invención se refiere a un conjunto de […]
GENERACION DE MUESTRAS PARA GENOTIPAR MEDIANTE ESPECTROMETRIA DE MASAS, del 29 de Octubre de 2010, de CENTRE NATIONAL DE GENOTYPE: Método para el genotipado de polimorfismos de un nucleótido único (SNP) mediante espectrometría de masas, que comprende las etapas siguientes: a. […]
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]