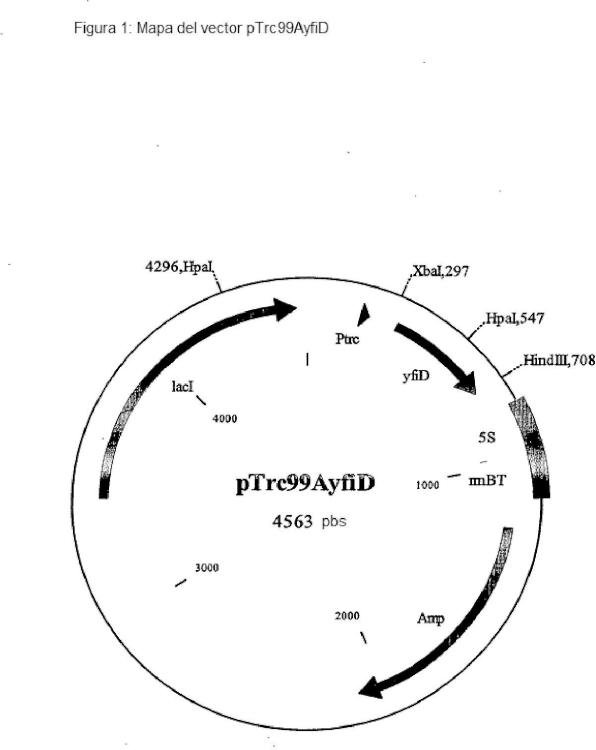
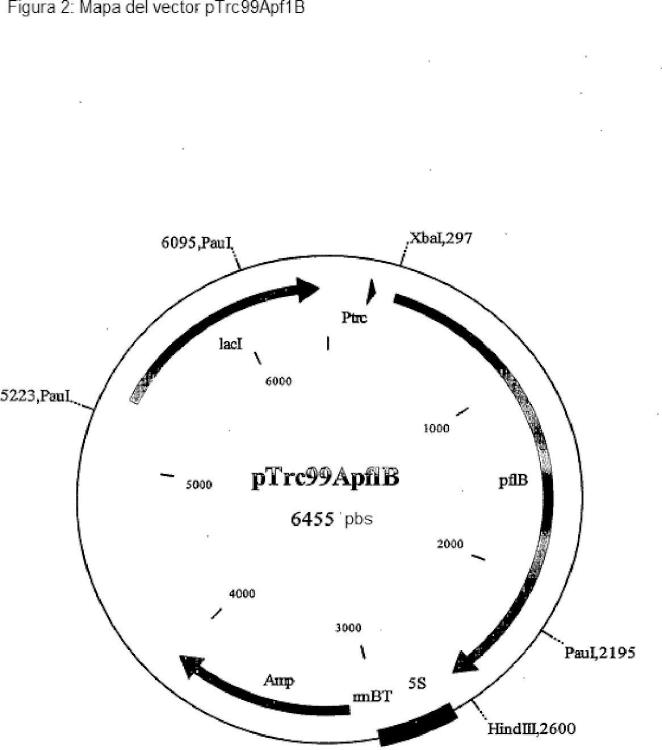
Procedimiento para la producción de L-treonina, utilizando cepas de la familia Enterobacteriaceae que contienen un marco de lectura abierto yfiD y/o un gen pflB intensificado.
Un procedimiento para la producción de L-treonina mediante fermentación de microorganismos recombinantes de la familia Enterobacteriaceae,
en el que
a) en los microorganismos que ya producen L-treonina se sobre-expresan el ORF yfiD y/o el gen pflB o las secuencias de nucleótidos que codifican los productos génicos, y dichos microorganismos se cultivan en un medio en condiciones en las que la L-treonina está enriquecida en el medio o en las células, y
b) la L-treonina se aísla, con lo que opcionalmente constituyentes del caldo de fermentación y/o la biomasa en su totalidad o en porciones permanecen en el producto aislado o se separan por completo.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2004/003207.
Solicitante: EVONIK DEGUSSA GMBH.
Nacionalidad solicitante: Alemania.
Dirección: RELLINGHAUSER STRASSE 1-11 45128 ESSEN ALEMANIA.
Inventor/es: FARWICK, MIKE, RIEPING, MECHTHILD.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N1/21 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 1/00 Microorganismos, p.ej. protozoos; Composiciones que los contienen (preparaciones de uso médico que contienen material de protozoos, bacterias o virus A61K 35/66, de algas A61K 36/02, de hongos A61K 36/06; preparación de composiciones de uso médico que contienen antígenos o anticuerpos bacterianos, p. ej. vacunas bacterianas, A61K 39/00 ); Procesos de cultivo o conservación de microorganismos, o de composiciones que los contienen; Procesos de preparación o aislamiento de una composición que contiene un microorganismo; Sus medios de cultivo. › modificados por la introducción de material genético extraño.
- C12N15/54 C12N […] › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Transferasas (2).
- C12P13/08 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 13/00 Preparación de compuestos orgánicos que contienen nitrógeno. › Lisina; Acido diaminopimélico; Treonina; Valina.
PDF original: ES-2553605_T3.pdf
Patentes similares o relacionadas:
Microorganismo con productividad de l-lisina aumentada y procedimiento para producir l-lisina utilizando el mismo, del 27 de Mayo de 2020, de CJ CHEILJEDANG CORPORATION: Una subunidad beta prima (subunidad-β') mutante de la ARN polimerasa, en la que la subunidad beta prima (subunidad-β') mutante de la ARN polimerasa tiene […]
Construcción de nuevas variantes de dextransacarasa DSR-S por ingeniería genética, del 6 de Mayo de 2020, de CENTRE NATIONAL DE LA RECHERCHE SCIENTIFIQUE: 1. Una dextransacarasa que consiste en una secuencia que tiene el 90 %, el 95 % o el 98 % de similitud de secuencia con una secuencia de aminoácidos seleccionada del fragmento […]
Construcción de nuevas variantes de dextransacarasa DSR-S por ingeniería genética, del 22 de Abril de 2020, de CENTRE NATIONAL DE LA RECHERCHE SCIENTIFIQUE: Una secuencia de nucleótidos que consiste en una secuencia de nucleótidos como se define en SEQ ID NO: 1 o la secuencia complementaria a la secuencia como […]
Procedimiento para producir 7-deshidrocolesterol y vitamina D3, del 27 de Noviembre de 2019, de KYOWA HAKKO BIO CO., LTD: Procedimiento para producir 7-deshidrocolesterol (en adelante, "7DHC"), que comprende: cultivar, en un medio, un microorganismo de Labyrinthulea que produce 7DHC en el que […]
ADN polimerasas quiméricas, del 12 de Junio de 2019, de Kapa Biosystems, Inc: Una ADN polimerasa quimérica que comprende: una primera secuencia al menos un 90% idéntica a los residuos aminoacídicos 156 a 301 de SEQ IN NO: 11; una segunda secuencia […]
Transaminasa y utilización de la misma, del 15 de Mayo de 2019, de Asymchem Laboratories (Tianjin) Co., Ltd: Transaminasa o compuesto modificado, o fragmento funcional de la misma, en la/el que una secuencia de aminoácidos de la transaminasa comprende una […]
ADN POLIMERASAS INDEPENDIENTES DE CEBADOR Y SU USO PARA LA SINTESIS DE ADN, del 24 de Abril de 2019, de CONSEJO SUPERIOR DE INVESTIGACIONES CIENTIFICAS: ADN polimerasas independientes de cebador y su uso para la síntesis de ADN. La presente invención proporciona un péptido aislado de SEQ ID NO: 1, […]
Microorganismo que produce O-succinil homoserina y método para producir O-succinil homoserina usando el mismo, del 27 de Febrero de 2019, de CJ CHEILJEDANG CORPORATION: Una O-succinilhomoserina que produce un microorganismo de Escherichia sp. que expresa un polipéptido, que tiene una resistencia a la inhibición por retroalimentación […]