Sistemas de despliegue de levaduras.
Una biblioteca de células huésped, en donde cada célula huésped comprende:
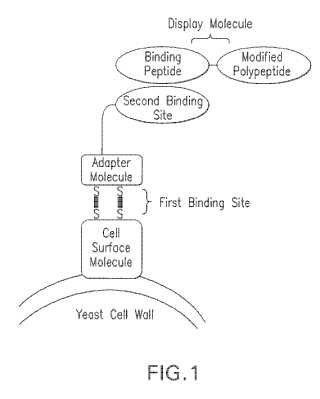
(a) una molécula de la superficie de la célula unida a la superficie de la célula,
(b) una molécula adaptadora que comprende un primer sitio de enlazamiento y un segundo sitio de enlazamiento, y (c) una molécula de despliegue que comprende un polipéptido modificado;
en donde el primer sitio de enlazamiento se enlaza específicamente con la molécula de la superficie de la célula y no puede enlazarse con la molécula de despliegue y el segundo sitio de enlazamiento se enlaza específicamente con la molécula de despliegue y no puede enlazarse con la molécula de la superficie de la célula en donde la molécula adaptadora no es un componente del polipéptido modificado, y en donde cada célula huésped comprende un polipéptido diferente modificado.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2009/067066.
Solicitante: NOVARTIS AG.
Nacionalidad solicitante: Suiza.
Dirección: LICHTSTRASSE 35 4056 BASEL SUIZA.
Inventor/es: LOEW,ANDREAS.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/81 C12N 15/00 […] › para levaduras.
- C40B40/08 C […] › C40 TECNOLOGIA COMBINATORIA. › C40B QUIMICA COMBINATORIA; BIBLIOTECAS, p. ej. QUIMIOTECAS (bibliotecas combinatorias in silico de ácidos nucleicos, proteínas o péptidos G16B 35/00; química combinatoria in silico G16C 20/60). › C40B 40/00 Bibliotecas per se , p. ej. arrays, mezclas. › Bibliotecas que contienen ARN o ADN que codifican proteínas, p. ej. genotecas.
PDF original: ES-2410579_T3.pdf
Fragmento de la descripción:
Sistemas de despliegue de levaduras
Campo La presente invención se refiere al campo de bibliotecas de despliegue de proteína y tamizaje de bibliotecas. De manera más específica, la presente invención se refiere a la producción de proteínas para despliegue sobre superficies de células.
Antecedentes de la invención Los dominios de enlazamiento de proteína se pueden predecir a partir de los datos de la secuencia, sin embargo, el rediseño de proteínas con afinidades de enlazamiento mejoradas o alteradas con frecuencia requiere del análisis de una cantidad de variantes de la proteína rediseñada. Actualmente el mejor método para obtener proteínas con las afinidades de enlazamiento deseadas es generar y tamizar una biblioteca de proteínas que incluya a tales variantes que puedan incluir proteínas rediseñadas racionalmente, proteínas alteradas en forma aleatoria, o una combinación de las mismas. Se han construido y tamizado en forma satisfactoria bibliotecas de muchos tipos de proteína, tales como inmunoglobulinas y proteínas de andamiaje y receptores o ligandos de receptores con respecto a la afinidad de enlazamiento.
Existen muchos métodos para tamizar bibliotecas, pero uno de los métodos más comunes es el método de despliegue en fagos, el cual comprende la fusión de una biblioteca de proteína a las proteínas de recubrimiento de un fago filamentoso (por ejemplo, Huse et al, '89; Clackson et al., '91; Marks et al., '92) . Las fusiones se hacen más comúnmente con una proteína menor de recubrimiento, llamada la proteína del gen III (pIII) , la cual está presente en tres a cinco copias de la punta del fago. La biblioteca fusionada se despliega luego sobre la superficie de la partícula viral. La biblioteca de despliegue en fagos puede ser luego tamizada contra una proteína objetivo inmovilizada. Sin embargo, un inconveniente importante de este método es que las proteínas objetivo que se enlazan a la biblioteca con muy alta afinidad no siempre son identificadas debido a que las condiciones requeridas para eIuir la fase enlazada usualmente desnaturalizan la partícula de fago de modo tal que se hace imposible identificar la proteína de interés. Otro inconveniente de las bibliotecas de despliegue en fago es el requerimiento de que la proteína objetivo se inmovilice sobre una superficie sólida, lo cual puede conducir a dificultades para determinar la afinidad real de una proteína objetivo para la proteína de despliegue en fago. Además, algunas proteínas de interés requieren de modificaciones posteriores a la traducción, tales como glucosilación, metilación, o formación de puentes de disulfuro, que no se pueden lograr cuando se expresan en una partícula de fago.
Un método alternativo para seleccionar bibliotecas de proteína es desplegar la biblioteca sobre la superficie de células bacterianas. Este método resuelve muchos de los inconvenientes asociados con el despliegue en fago, pero tiene sus propios problemas. Un problema con el despliegue en bacterias es que la cápsula bacteriana puede causar impedimento estérico a las proteínas desplegadas sobre la superficie de la bacteria. Además, las bacterias no contienen la maquinaria para plegar de manera adecuada las proteínas eucariotas, de modo que la proteína de interés no siempre puede ser expresada dentro de la bacteria. En forma similar al problema en el fago, las bacterias no pueden proporcionar modificaciones posteriores a la traducción, tales como la formación de puentes de disulfuro, con una proteína eucariota.
Wittrup et al. (WO9936569) han desarrollado un método para una biblioteca que se despliega en célula de levadura. Este es un sistema de dos componentes, en donde el primer componente implica la expresión de una subunidad de la proteína de adhesión de apareamiento de levadura, aglutinina, la cual se ancla a la pared de la célula de levadura. El segundo componente implica la expresión de una biblioteca de proteína fusionada a una segunda subunidad de la proteína de aglutinina que forma puentes de disulfuro de alta afinidad con la primera subunidad de aglutinina. La biblioteca de proteína fusionada a la aglutinina es desplegada por lo tanto sobre la superficie de la célula. La biblioteca puede entonces ser seleccionada. Este método permite el plegamiento apropiado y la modificación posterior a la traducción de las proteínas eucariotas. La solicitud de patente WO2007046825 divulga métodos para detectar antígenos solubles usando células de detección que expresan un Receptor I gamma de Fc y una molécula llamada aequorina, que emite un fotón en respuesta a mayores concentraciones de calcio en la célula. El entrelazamiento de los receptores enlazados al anticuerpo por medio de un antígeno específico sobre la célula de detección resulta en un incremento en la concentración intracelular de calcio y por lo tanto las moléculas de aequorina emiten protones en respuesta al incremento en la concentración de calcio. Regis Cebe et al., divulgan un procedimiento de selección in vitro para fagos no infecciosos que despliegan un ligando capaz de interactuar con una molécula objetivo biotinilada. El dominio de estreptavidina de un adaptador modificado por ingeniería genética sirve como enlace entre una molécula objetivo biotinilada y el fago no infeccioso que carece de una proteína de recubrimiento del gen III funcional pero despliega en vez de eso un ligando de interacción (Regis et al., Size of the ligand complex between the N-terminal domain of the gene III coat protein and the noninfectious phage strongly influences the usefulness of in vitro selective infective phage technology, Biochemical Journal, volumen: 352, páginas: 841 - 849) .
Rakestraw et al. (PCT/US2008/003978) han desarrollado un sistema de tres componentes para desplegar una biblioteca de proteína sobre la superficie de células de levadura. El primer componente implica la expresión de una biblioteca de proteína fusionada a un péptido de enlazamiento de biotina, el segundo componente implica la modificación de la pared de la célula de levadura para expresar biotina, y el tercer componente implica el enlazamiento de avidina a la biotina expresada sobre la superficie de la célula. La biblioteca de proteína fusionada es luego biotinilada y secretada desde la célula de levadura y se enlaza a la avidina sobre la superficie de la célula de levadura, desplegando por lo tanto la biblioteca de proteína sobre la superficie de la célula de levadura. Un inconveniente potencial de este sistema es que la avidina no enlaza específicamente toda la biotina. Otro inconveniente potencial es que la avidina contiene cuatro sitios de enlazamiento, los cuales pueden ocasionar impedimento estérico evitando de esta manera que la biblioteca de proteína biotinilada se enlace con la avidina enlazada a la superficie de la célula. De manera similar, la molécula de avidina se puede enlazar a la biblioteca de proteína biotinilada antes de enlazarse a la pared de célula de levadura biotinilada, impidiendo así el enlazamiento de la avidina con la pared de la célula de levadura. Adicionalmente, este método tiene la complicación adicional de tener que biotinilar la biblioteca de proteína dentro de la célula de levadura. Esta etapa adicional necesaria requiere la modificación adicional de la célula de levadura. Está bien establecido que mientras más modificaciones se hagan a un sistema biológico, menos probable será que dicho sistema se comporte como se diseñó. Además, debido a que la avidina/estreptavidina es multivalente, se debe tener cuidado de no entrelazar las células biotiniladas. Por último, la biotina/estreptavidina y biotina/avidina se utilizan en una cantidad de kits para marcación comercialmente disponibles. Dichos kits podrían ser difíciles de usar en tal sistema de despliegue de biotina/avidina.
El estado del arte carece de un sistema sencillo y eficiente, capaz de enlazarse específicamente a una biblioteca de proteína secretada utilizando una molécula adaptadora que se una a la biblioteca de proteína y a la superficie de una célula eucariota a través de porciones diferentes de enlazamiento.
Resumen de la invención La presente invención satisface esta necesidad proporcionando métodos y composiciones como los divulgados a través de toda la memoria descriptiva. Un aspecto incluye una biblioteca de células huésped con una molécula de la superficie de la célula unida a la superficie de la célula, una molécula adaptadora que comprende un primer sitio de enlazamiento y un segundo sitio de enlazamiento, y una molécula de despliegue que comprende un polipéptido modificado en el que el primer sitio de enlazamiento se enlaza específicamente con la molécula de la superficie de la célula y no se puede enlazar con la molécula de despliegue,... [Seguir leyendo]
Reivindicaciones:
1. Una biblioteca de células huésped, en donde cada célula huésped comprende:
(a) una molécula de la superficie de la célula unida a la superficie de la célula,
(b) una molécula adaptadora que comprende un primer sitio de enlazamiento y un segundo sitio de enlazamiento, y
(c) una molécula de despliegue que comprende un polipéptido modificado;
en donde el primer sitio de enlazamiento se enlaza específicamente con la molécula de la superficie de la célula y no puede enlazarse con la molécula de despliegue y el segundo sitio de enlazamiento se enlaza específicamente con la molécula de despliegue y no puede enlazarse con la molécula de la superficie de la célula en donde la molécula adaptadora no es un componente del polipéptido modificado, y en donde cada célula huésped comprende un polipéptido diferente modificado.
2. La biblioteca de células huésped de la reivindicación 1 en donde las células huésped de dicha biblioteca comprende una pluralidad de moléculas de despliegue.
3. La biblioteca de células huésped de la reivindicación 1 o de la reivindicación 2, en donde la molécula de la superficie de la célula huésped está covalentemente enlazada al primer sitio de enlazamiento.
4. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 3, en donde la molécula de la superficie de la célula huésped comprende una primera aglutinina que es Aga1p, y en donde el primer sitio de enlazamiento comprende una segunda aglutinina que es Aga2p.
5. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 4, en donde la molécula de la superficie de la célula huésped está unida a la membrana de la célula a través de un ancla GIP.
6. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 5, en donde el segundo sitio de enlazamiento está covalentemente enlazado a la molécula de despliegue.
7. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 6, en donde el segundo sitio de enlazamiento comprende un dominio PDZ de InaD.
8. La biblioteca de células huésped de la reivindicación 7, en donde la molécula de despliegue comprende un ligando de NorpA del terminal C.
9. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 6, en donde la molécula de despliegue comprende un dominio PDZ de InaD.
10. La biblioteca de células huésped de la reivindicación 9, en donde el segundo sitio de enlazamiento comprende un ligando de NorpA del terminal C.
11. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 10, en donde el polipéptido modificado se selecciona de entre el grupo que consiste de: una proteína de andamiaje, una proteína de transducción de señal, un anticuerpo, una inmunoglobulina, una inmunoadhesina, un receptor, un ligando, una oncoproteína, un factor de transcripción, una enzima, y un polipéptido de fibronectina.
12. La biblioteca de células huésped de la reivindicación 11, en donde el polipéptido de fibronectina comprende un polipéptido F10.
13. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 12, en donde la molécula de despliegue comprende: un péptido señal de secreción.
14. La biblioteca de células huésped de la reivindicación 13, en donde el péptido señal de secreción comprende un péptido líder híbrido MFalfa/HSA, o un péptido líder MFalfa.
15. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 14, en donde la expresión de la molécula de despliegue está bajo el control de un primer promotor inducible seleccionado del grupo que consiste de un promotor AOX1, un promotor Cup 1, y un promotor Gal 1.
16. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 15, en donde la expresión de la molécula adaptadora está bajo el control de un segundo promotor inducible seleccionado de entre el grupo que consiste de un promotor AOX1, un promotor Cup 1, y un promotor Gal 1.
17. La biblioteca de células huésped de cualquiera de las reivindicaciones 1 - 16, en donde la célula huésped es una célula de levadura.
18. Un método para desplegar un polipéptido modificado que comprende
(a) proveer una célula huésped que comprende una molécula de la superficie de la célula unida a la superficie de la célula y un primer ácido nucleico que codifica un polipéptido de despliegue que comprende un polipéptido modificado,
(b) poner en contacto la célula huésped con una molécula adaptadora que comprende un primer sitio de enlazamiento y un segundo sitio de enlazamiento bajo condiciones en donde el primer sitio de enlazamiento se enlaza con la molécula de la superficie de la célula, y
(c) incubar la célula huésped bajo condiciones en donde la célula huésped exporta al polipéptido de despliegue fuera de la célula huésped bajo condiciones en donde el segundo sitio de enlazamiento se enlaza con el polipéptido de despliegue,
en donde el primer sitio de enlazamiento se enlaza específicamente con la molécula de la superficie de la célula y no puede enlazarse con la molécula de despliegue y el segundo sitio de enlazamiento se enlaza específicamente con el polipéptido de despliegue y no puede enlazarse con la molécula de la superficie de la célula y en donde la molécula adaptadora no es un componente del polipéptido modificado.
19. Un método para generar una biblioteca de despliegue de células huésped que comprende:
la introducción dentro de una pluralidad de células huésped de una biblioteca de despliegue de los primeros ácidos nucleicos cada uno codificando un polipéptido de despliegue que comprende un polipéptido modificado, en donde al menos dos de los primeros ácidos nucleicos introducidos codifican diferentes polipéptidos modificados,
en donde cada célula huésped comprende un segundo ácido nucleico que codifica un polipéptido de la superficie de la célula y un tercer ácido nucleico que codifica una molécula adaptadora que comprende un primer sitio de enlazamiento y un segundo sitio de enlazamiento, en donde el primer sitio de enlazamiento se enlaza con la molécula de la superficie de la célula pero no con el polipéptido de despliegue y el segundo sitio de enlazamiento se enlaza con el polipéptido de despliegue pero no con la molécula de la superficie de la célula, y en donde la molécula adaptadora no es un componente del polipéptido modificado.
Patentes similares o relacionadas:
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Método para romper un ácido nucleico y añadir un adaptador por medio de transposasa y reactivo, del 1 de Julio de 2020, de MGI Tech Co., Ltd: Un metodo para romper un acido nucleico y anadir un adaptador por medio de una transposasa, que comprende las siguientes etapas: interrumpir […]
Vacuna subunitaria contra Mycoplasma spp., del 1 de Julio de 2020, de Agricultural Technology Research Institute: Una composición para prevenir una infección por Mycoplasma spp., que comprende: un principio activo, que comprende una proteína de PdhA; y un adyuvante […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Proteínas de captura de la superficie celular recombinantes, del 13 de Mayo de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para detectar y aislar células que producen altos niveles de una proteína heterodimérica que tiene una primera subunidad y una segunda subunidad, […]
Etiquetado y evaluación de una secuencia diana, del 13 de Mayo de 2020, de RhoDx, Inc: Un método para modificar un ácido nucleico, que comprende: (a) poner en contacto un ácido nucleico de cadena sencilla con una actividad de transferasa […]
Preparación de bibliotecas de ácido nucleico marcado usando protocolo de adición en un solo tubo, del 13 de Mayo de 2020, de ILLUMINA, INC: Un método para preparar una biblioteca de fragmentos de ácido nucleico marcados que comprende: (a) poner en contacto una célula individual […]