Remolacha tolerante al glifosato.
Una planta de remolacha tolerante al glifosato, caracterizada porque
a) un fragmento de ADN del ADN genómico de la planta de remolacha,
partes o semillas de la misma, puedeser amplificado mediante reacción en cadena de polimerasa con un primer cebador que presenta la secuencianucleotídica de SEQ ID NO: 3 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 4,presentando el fragmento de ADN al menos 95% de identidad con la secuencia nucleotídica de SEQ ID NO: 6, y/o
b) un fragmento de ADN del ADN genómico de la planta de remolacha, partes o semillas de la misma, puedeser amplificado mediante reacción en cadena de polimerasa con un primer cebador que presenta la secuencianucleotídica de SEQ ID NO: 9 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 10,presentando el fragmento de ADN al menos 95% de identidad con la secuencia nucleotídica de SEQ ID NO: 12, y/o
c) un fragmento de ADN del ADN genómico de la planta de remolacha, partes o semillas de la misma, puedeser amplificado mediante reacción en cadena de polimerasa con un primer cebador que presenta la secuencianucleotídica de SEQ ID NO: 14 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 16,presentando el fragmento de ADN al menos 95% de identidad con la secuencia nucleotídica de SEQ ID NO: 17.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2004/001469.
Solicitante: KWS SAAT AG.
Nacionalidad solicitante: Alemania.
Dirección: Grimsehlstrasse 31 37555 Einbeck ALEMANIA.
Inventor/es: NEHLS,REINHARD, KRAUS,JOSEF, SAUERBREY,ELKE, LOOCK,ANDREAS, JANSEN,RUDOLF.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- A01H5/00 NECESIDADES CORRIENTES DE LA VIDA. › A01 AGRICULTURA; SILVICULTURA; CRIA; CAZA; CAPTURA; PESCA. › A01H NOVEDADES VEGETALES O PROCEDIMIENTOS PARA SU OBTENCION; REPRODUCCION DE PLANTAS POR TECNICAS DE CULTIVO DE TEJIDOS. › Angiospermas,es decir, plantas con flores, caracterizadas por sus partes vegetales; Angiospermas caracterizadas de forma distinta que por su taxonomía botánica.
- C12N15/82 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › para células vegetales.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2391090_T3.pdf
Fragmento de la descripción:
Remolacha tolerante al glifosato
La invención se refiere a plantas de remolacha, material vegetal y semillas tolerantes al glifosato, así como a un procedimiento y un kit de ensayo para la identificación de una planta de remolacha tolerante al glifosato.
La remolacha (Beta vulgaris) se produce como un cultivo comercial en muchos países, con una cosecha global de más de 240 millones de toneladas métricas.
La N-fosfonometilglicina, comúnmente denominada glifosato, es un herbicida de amplio espectro que se usa extensamente debido a su alto rendimiento, biodegradabilidad y baja toxicidad para animales y seres humanos. El glifosato inhibe la ruta del ácido shikímico que lleva a la síntesis de compuestos aromáticos, incluyendo aminoácidos y vitaminas. En particular, el glifosato inhibe la conversión del ácido fosfoenolpirúvico y el shikimato-3-fosfato en 5enolpiruvil-shikimato-3-fosfato, por inhibición de la enzima 5-enolpiruvil-shikimato-3-fosfato sintasa (EPSP sintasa o EPSPS) . Cuando plantas convencionales son tratadas con glifosato, las plantas no pueden producir los aminoácidos aromáticos (por ejemplo fenilalanina y tirosina) necesarios para crecer y sobrevivir. EPSPS está presente en todas las plantas, bacterias y hongos. No está presente en animales, quienes no sintetizan sus propios aminoácidos aromáticos. Debido a que la ruta biosintética de los aminoácidos aromáticos no está presente en mamíferos, aves ni formas de vida acuáticas, el glifosato tiene poca o ninguna toxicidad para estos organismos. La enzima EPSPS está presente naturalmente en alimentos procedentes de fuentes vegetales y microbianas.
El glifosato es el ingrediente activo en herbicidas como por ejemplo Roundup®, producido por Monsanto Company, EE.UU. Típicamente, se formula como una sal soluble en agua como por ejemplo una sal de amonio, alquilamina, metal alcalino o trimetil-sulfonio. Una de las formulaciones más comunes es la sal de isopropilamina del glifosato, que es la forma empleada en el herbicida Roundup®.
Se ha demostrado que se pueden obtener plantas tolerantes al glifosato insertando en el genoma de la planta la capacidad para producir una EPSP sintasa que es tolerante al glifosato, por ejemplo la CP4-EPSPS de la cepa CP4AcO de Agrobacterium sp.
Se puede obtener una planta de remolacha tolerante al glifosato por transformación mediada por Agrobacterium, introduciendo en el genoma de la planta un gen que codifica para una EPSPS tolerante al glifosato tal como CP4-EPSPS. Esta planta de remolacha que expresa CP4-EPSPS se ha descrito en el documento WO 99/23232. Sin embargo, las plantas de remolacha cultivadas a partir de células transformadas de esta manera con el gen para CP4-EPSPS difieren mucho en sus características, debido al hecho de que el gen es insertado en una posición aleatoria en el genoma de la planta. La inserción de un transgen particular en una ubicación específica en un cromosoma se denomina frecuentemente "evento". El término "evento" también se usa para diferenciar variedades de cultivos modificados por ingeniería genética. Los eventos deseados son muy raros. La gran mayoría de los eventos son desechados porque el transgen se inserta en un gen de la planta importante para el crecimiento, lo que hace que el gen se altere y no se exprese, o el transgen se inserta en una porción del cromosoma que no permite la expresión del transgen o permite una expresión demasiado baja. Por esta razón es necesario examinar un gran número de eventos para identificar un evento caracterizado por una expresión suficiente del gen introducido. Este procedimiento consume mucho tiempo y genera muchos gastos y no garantiza de ninguna manera que pueda encontrarse una planta con propiedades satisfactorias.
Por lo tanto, el objetivo de la presente invención es proporcionar una planta de remolacha que muestre un alto nivel de tolerancia contra el glifosato, pero que no tenga desventajas con respecto a otras propiedades agronómicas importantes, tales como crecimiento, rendimiento, calidad, resistencia a patógenos, etc.
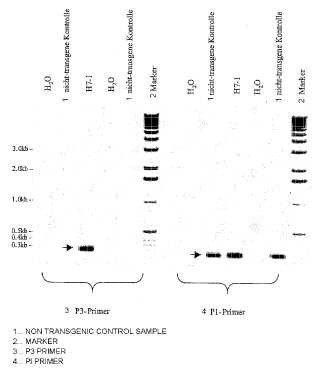
La planta de remolacha tolerante al glifosato de acuerdo con la invención se caracteriza porque: a) un fragmento de ADN del ADN genómico de la planta de remolacha, partes o semillas de la misma, puede ser amplificado mediante reacción en cadena de polimerasa con un primer cebador que presenta la secuencia nucleotídica de SEQ ID NO: 3 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 4, presentando el fragmento de ADN al menos 95% de identidad con la secuencia nucleotídica de SEQ ID NO: 6, y/o b) un fragmento de ADN del ADN genómico de la planta de remolacha, partes o semillas de la misma, puede ser amplificado mediante reacción en cadena de polimerasa con un primer cebador que presenta la secuencia nucleotídica de SEQ ID NO: 9 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 10, presentando el fragmento de ADN al menos 95% de identidad con la secuencia nucleotídica de SEQ ID NO: 12, y/o c) un fragmento de ADN del ADN genómico de la planta de remolacha, partes o semillas de la misma, puede ser amplificado mediante reacción en cadena de polimerasa con un primer cebador que presenta la secuencia nucleotídica de SEQ ID NO: 14 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 16, presentando el fragmento de ADN al menos 95% de identidad con la secuencia nucleotídica de SEQ ID NO: 17.
La reacción en cadena de polimerasa (PCR) es un método estándar bien conocido para amplificar moléculas de ácido nucleico (véase por ejemplo la patente norteamericana US 4.683.202) .
La planta de remolacha de acuerdo con la invención (en adelante denominada "evento H7-1") muestra una alta tolerancia al herbicida glifosato. Además, las características de crecimiento y otras propiedades agronómicas importantes del evento H7-1 no son afectadas por el proceso de transformación. El evento H7-1 expresa a un alto nivel el gen CP4-EPSPS de Agrobacterium, que está incorporado de forma estable en el genoma de la planta y que confiere a la planta tolerancia al glifosato. La planta fue obtenida por tecnología de transformación mediada por Agrobacterium, usando el vector binario PV-BVGT08. Este vector contiene, entre la región de borde (“Border"-Region) izquierda y la derecha, las siguientes secuencias: una región de codificación compuesta de una secuencia de codificación de un péptido de tránsito de los cloroplastos de la EPSPS de Arabidopsis thaliana (denominada ctp2) unida a la secuencia de codificación de CP4-EPSPS, y bajo la regulación del promotor del virus de mosaico de escrofularia (pFMV) y la secuencia de terminación de la transcripción E9-3' de Pisum sativum.
En una forma de realización preferida de la invención, los fragmentos de ADN de 3706 bp, 751 bp y 1042 bp muestran al menos 95%, de preferencia al menos 99%, de mayor preferencia al menos 99, 9% de identidad con las secuencias nucleotídicas de SEQ ID NO: 6, SEQ ID NO: 12 o SEQ ID NO: 17, respectivamente. Por ejemplo, 95% de identidad significa que el 95% de los nucleótidos de una secuencia dada son idénticos a la secuencia comparada. Para este propósito, las secuencias pueden ser alineadas y comparadas usando el programa BLAST (disponible por ejemplo en http://www.ncbi.nlm.nih.gov/blast/bl2seq/bl2.html) . De máxima preferencia, el fragmento de ADN de 3706 bp tiene la secuencia nucleotídica de SEQ ID NO: 6, el fragmento de ADN de 751 bp tiene la secuencia nucleotídica de SEQ ID NO: 12 y/o el fragmento de ADN de 1042 bp tiene la secuencia nucleotídica de SEQ ID NO: 17.
La presente invención también se refiere a la semilla depositada en NCIMB, Aberdeen (Escocia, Reino Unido) , y que tiene el número de acceso NCIMB 41158 o NCIMB 41159. Dicha semilla se puede usar para obtener una planta de remolacha tolerante al glifosato. La semilla se puede sembrar y la planta obtenida será tolerante al glifosato.
La invención también se refiere a una célula, tejido o parte de una planta de remolacha tolerante al glifosato.
Otro aspecto de la presente invención es un método para la identificación de una planta de remolacha tolerante al glifosato, caracterizado porque el método comprende la (s) etapa (s) de: a) amplificar un fragmento de ADN de 3500-3900 bp, de preferencia 3706 bp, del ADN genómico de plantas de remolacha, partes o semillas de la misma, usando una reacción en cadena de polimerasa con un primer cebador que presenta la secuencia nucleotídica de SEQ ID NO: 3 y un segundo cebador que presenta la secuencia nucleotídica... [Seguir leyendo]
Reivindicaciones:
1. Una planta de remolacha tolerante al glifosato, caracterizada porque a) un fragmento de ADN del ADN genómico de la planta de remolacha, partes o semillas de la misma, puede ser amplificado mediante reacción en cadena de polimerasa con un primer cebador que presenta la secuencia nucleotídica de SEQ ID NO: 3 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 4, presentando el fragmento de ADN al menos 95% de identidad con la secuencia nucleotídica de SEQ ID NO: 6, y/o b) un fragmento de ADN del ADN genómico de la planta de remolacha, partes o semillas de la misma, puede ser amplificado mediante reacción en cadena de polimerasa con un primer cebador que presenta la secuencia nucleotídica de SEQ ID NO: 9 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 10, presentando el fragmento de ADN al menos 95% de identidad con la secuencia nucleotídica de SEQ ID NO: 12, y/o c) un fragmento de ADN del ADN genómico de la planta de remolacha, partes o semillas de la misma, puede ser amplificado mediante reacción en cadena de polimerasa con un primer cebador que presenta la secuencia nucleotídica de SEQ ID NO: 14 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 16, presentando el fragmento de ADN al menos 95% de identidad con la secuencia nucleotídica de SEQ ID NO: 17.
2. Semillas de una planta de acuerdo con la reivindicación 1.
3. Célula, tejido o partes de la planta de acuerdo con la reivindicación 1.
4.
71. 790 bp, de preferencia 751 bp, del ADN genómico de dicha planta de remolacha, partes o semillas de la misma, usando una reacción en cadena de polimerasa con un primer cebador que presenta la secuencia nucleotídica de SEQ ID NO: 9 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 10, y/o c) amplificar un fragmento de ADN d.
99. 1100 bp, de preferencia 1042 bp, del ADN genómico de dicha planta de remolacha, partes o semillas de la misma, usando una reacción en cadena de polimerasa con un primer cebador que presenta la secuencia nucleotídica de SEQ ID NO: 14 y un segundo cebador que presenta la secuencia nucleotídica de SEQ ID NO: 16.
5. Kit de ensayo para identificar una planta de remolacha transgénica tolerante al glifosato, sus células, tejidos
o partes, comprendiendo al menos un par de cebadores con un primer y un segundo cebador para una reacción en cadena de polimerasa, donde el primer cebador reconoce una secuencia dentro del ADN foráneo incorporado en el genoma de dicha planta, y el segundo cebador reconoce una secuencia dentro de las regiones flanqueadoras 3' o 5' de dicho ADN, siendo la planta una planta de acuerdo con la reivindicación 1.
6. Kit de ensayo según la reivindicación 5, caracterizado porque a) el primer cebador presenta la secuencia nucleotídica de SEQ ID NO: 9 y el segundo cebador presenta la secuencia nucleotídica de SEQ ID NO: 10, y/o b) el primer cebador presenta la secuencia nucleotídica de SEQ ID NO: 14 y el segundo cebador presenta la secuencia nucleotídica de SEQ ID NO: 16.
Patentes similares o relacionadas:
Producción de partículas similares al virus de la gripe en plantas, del 6 de Mayo de 2020, de MEDICAGO INC.: Un ácido nucleico que comprende una región reguladora activa en una planta y un potenciador de la expresión activo en una planta, la región […]
Combinación de dos elementos genéticos para el control del desarrollo del tipo floral de una planta dicotiledónea, y utilización en procedimientos de detección y selección, del 1 de Abril de 2020, de Institut national de recherche pour l'agriculture, l'alimentation et l'environnement: Utilización de una combinación de dos elementos genéticos para el control del desarrollo del tipo floral de una planta dicotiledónea, comprendiendo dicha combinación, respectivamente: […]
Proteínas insecticidas y métodos para su uso, del 12 de Febrero de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Una construcción de ADN que comprende una molécula de ácido nucleico heteróloga que codifica un polipéptido de PIP-72 que tiene actividad insecticida contra el gusano de […]
Polinucleótidos y polipéptidos aislados, y métodos para usar los mismos para incrementar la eficiencia en el uso de nitrógeno, rendimiento, tasa de crecimiento, vigor, biomasa, contenido de aceite, y/o tolerancia al estrés abiótico, del 13 de Noviembre de 2019, de Evogene Ltd: Un método para incrementar la eficiencia en el uso de nitrógeno, biomasa en condiciones limitantes de nitrógeno, y/o tasa de crecimiento de una planta que comprende: […]
Moléculas pequeñas de ARN que median en la interferencia de ARN, del 22 de Octubre de 2019, de MAX-PLANCK-GESELLSCHAFT ZUR FORDERUNG DER WISSENSCHAFTEN E.V.: Molécula de ARN de doble hebra aislada, en la que cada hebra de ARN tiene una longitud de 19-25 nucleótidos y al menos una hebra tiene un saliente […]
Planta Brassica que comprende un alelo indehiscente mutante, del 11 de Septiembre de 2019, de BASF Agricultural Solutions Seed US LLC: Un alelo mutante defectivo parcial de un gen IND, en el que el gen IND comprende una molécula de ácido nucleico seleccionada del grupo que […]
Receptor de reconocimiento de patrones de plantas y quimeras del mismo para su uso contra infecciones bacterianas, del 11 de Septiembre de 2019, de EBERHARD-KARLS-UNIVERSITAT TUBINGEN: Un receptor de reconocimiento de patrones quiméricos (PRR) para reconocer patrones moleculares asociados a patógenos de plantas, que comprende al menos un ectodominio […]
Plantas de arroz que tienen tolerancia incrementada frente a herbicidas de imidazolinona, del 31 de Julio de 2019, de INSTITUTO NACIONAL DE TECNOLOGIA AGROPECUARIA: Un método para la caracterización molecular de una planta de arroz que comprende amplificar, mediante PCR, un gen AHAS de una planta de arroz, en donde la […]