PRODUCCIÓN ENZIMÁTICA DE NOVO DE MOLÉCULAS DE ÁCIDO NUCLEICO.
Procedimiento para la fabricación de una molécula de ácido nucleico,
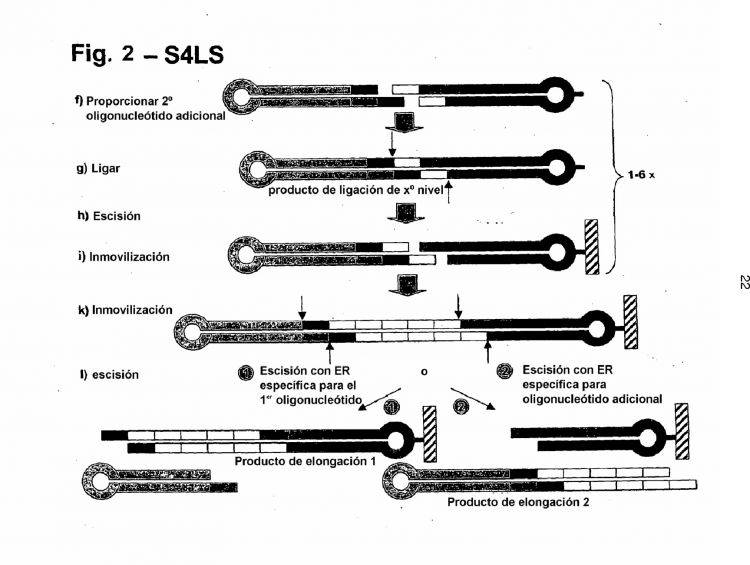
que comprende las etapas de a) proporcionar un primer oligonucleótido al menos parcialmente bicatenario que comprende un sitio de reconocimiento para una primera enzima de restricción de tipo IIS que corta fuera de su sitio de reconocimiento y comprendiendo dicho oligonucleótido un saliente monocatenario; b) proporcionar un segundo oligonucleótido al menos parcialmente bicatenario que comprende una modificación que permite al oligonucleótido acoplarse a una superficie, por lo cual el oligonucleótido comprende adicionalmente un sitio de reconocimiento para una segunda enzima de restricción de tipo IIS que corta fuera de su sitio de reconocimiento y comprendiendo dicho segundo oligonucleótido un saliente monocatenario; c) ligar el primer y el segundo oligonucleótidos mediante sus salientes generando un primer producto de ligación; d) inmovilizar el primer producto de ligación en una superficie mediante la modificación proporcionada por el segundo oligonucleótido; e) cortar el primer producto de ligación inmovilizado con la segunda enzima de restricción de tipo IIS liberando de este modo un primer oligonucleótido elongado que tiene un saliente y un segundo oligonucleótido acortado, que permanece unido a la superficie; f) proporcionar un oligonucleótido adicional al menos parcialmente bicatenario que tiene una modificación que permite al oligonucleótido adicional acoplarse de forma específica a una superficie, por lo cual el oligonucleótido contiene un sitio de reconocimiento para una segunda o una adicional enzima de restricción de tipo IIS y un saliente monocatenario que es complementario al saliente del primer oligonucleótido elongado; g) ligar el oligonucleótido adicional al menos parcialmente bicatenario con el primer oligonucleótido elongado mediante sus salientes generando un producto de ligación de segundo nivel; h) cortar el producto de ligación de segundo nivel con la segunda o adicional enzima de restricción de tipo IIS generando de este modo un oligonucleótido elongado de segundo nivel que tiene un saliente y un oligonucleótido adicional acortado; i) inmovilizar el oligonucleótido adicional acortado; j) repetir las etapas f) a i) al menos una vez, generando en la etapa g) un producto de ligación de nivel superior, por lo cual en la última repetición el oligonucleótido adicional entrante comprende un sitio de reconocimiento para una enzima de restricción de tipo IIS que produce tras la escisión un saliente monocatenario idéntico en longitud al saliente generado por la primer enzima de restricción de tipo IIS específica para el primer oligonucleótido y las etapas h) e i) se reemplazan con las etapas k) y l); k) inmovilizar el producto de ligación de nivel superior mediante la modificación proporcionada por el oligonucleótido adicional; y l) cortar el producto de ligación de nivel superior con la enzima de restricción de tipo IIS adicional, dejando la parte del ácido nucleico a fabricar unida al primer oligonucleótido, que se libera preferentemente al sobrenadante y más preferentemente permitiendo su transferencia a un nuevo recipiente de reacción
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E04001462.
Solicitante: SLONING BIOTECHNOLOGY GMBH.
Nacionalidad solicitante: Alemania.
Dirección: ZEPPELINSTR. 4 82178 PUCHHEIM ALEMANIA.
Inventor/es: SCHATZ, OCTAVIAN, SCHWER,HEINZ, HORN,GUDRUN,DR.
Fecha de Publicación: .
Fecha Solicitud PCT: 23 de Enero de 2004.
Fecha Concesión Europea: 29 de Septiembre de 2010.
Clasificación Internacional de Patentes:
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/66 C12N 15/00 […] › Métodos generales para insertar un gen en un vector para formar un vector recombinante, utilizando la escisión y la unión; Utilización de "linkers" no funcionales o de adaptadores, p. ej. "linkers" que contienen la secuencia para una endonucleasa de restricción.
- C12P19/34 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
Clasificación PCT:
- C12N15/10 C12N 15/00 […] › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/66 C12N 15/00 […] › Métodos generales para insertar un gen en un vector para formar un vector recombinante, utilizando la escisión y la unión; Utilización de "linkers" no funcionales o de adaptadores, p. ej. "linkers" que contienen la secuencia para una endonucleasa de restricción.
- C12P19/34 C12P 19/00 […] › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
Clasificación antigua:
- C12N15/10 C12N 15/00 […] › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/66 C12N 15/00 […] › Métodos generales para insertar un gen en un vector para formar un vector recombinante, utilizando la escisión y la unión; Utilización de "linkers" no funcionales o de adaptadores, p. ej. "linkers" que contienen la secuencia para una endonucleasa de restricción.
- C12P19/34 C12P 19/00 […] › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Eslovenia, Finlandia, Rumania, Chipre, Lituania, Letonia, Ex República Yugoslava de Macedonia, Albania.
Fragmento de la descripción:
La presente invención se refiere a procedimientos para la fabricación de una molécula de ácido nucleico.
La generación de novo de moléculas de ácido nucleico se usa de forma creciente en la investigación biofarmacéutica para reemplazar los procedimientos de clonación con frecuencia bastante complejos necesarios para producir construcciones de ADN deseadas con propiedades optimizadas, por ejemplo alto nivel de expresión de proteínas en sistemas in vivo
o in vitro adecuados. Existe una diversidad de procedimientos conocidos para sintetizar tales moléculas de ADN. Prácticamente todos estos procedimientos se basan en la síntesis, hibridación y ligación posterior de oligonucleótidos monocatenarios sintéticos para ensamblar moléculas de ADN bicatenario mayores que consisten típicamente en más de cien hasta varios miles de pares de bases. Sin embargo, la eficacia de estos procedimientos está limitada por varios factores: (i) la calidad de los oligonucleótidos usados, (ii) el tamaño de la construcción deseada y (iii) la proporción de secuencias “difíciles”, por ejemplo las que tienen regiones autocomplementarias, alto contenido de GC, tétradas de G, acodamientos de ADN o bloques de secuencias repetitivas. Los componentes básicos de los oligonucleótidos están contaminados ellos mismos con diversos productos de terminación y deleciones internas. Son especialmente problemáticos los productos n-1 (oligonucleótidos que contienen deleciones internas de un nucleótido que se producen como resultado de reacciones de protección incompletas), que difícilmente puede separarse del oligonucleótido deseado de cadena completa. Como tienen que ensamblarse muchos oligonucleótidos para generar un gen completo, la probabilidad de obtener un clon sin errores, es decir que no incorpore ni siquiera un oligonucleótido defectuoso con un cambio de base o una deleción interna se acerca al 0%. Por ejemplo, si un gen se ensambla a partir de 50 oligonucleótidos teniendo cada uno una pureza del 90%, la probabilidad de crear un producto sin errores sería de aproximadamente 0,950 = 0,005. Generalmente, deben emplearse procedimientos tediosos de corrección de errores para obtener una construcción 100% sin errores. En muchos casos, los productos de síntesis defectuosa no pueden tolerarse debido a que errores en la secuencia codificante pueden provocar la generación de productos de transcripción o traducción acortados debido a por ejemplo un cambio de fase de la fase abierta de lectura. Aunque los dos primeros problemas pueden mitigarse por el uso de oligonucleótidos de pureza muy alta, la formación de estructuras secundarias no deseadas que puedan provocar deleciones en el producto de síntesis puede en muchos casos suprimirse solamente si se permiten alteraciones en la secuencia de ADN.
En la técnica anterior se conocen una diversidad de procedimientos para producir ADN
sintético. Hace más de 20 años, el trabajo pionero de Khorana y colaboradores (Sekiya y col., 1979) demostró la síntesis completa de novo de un gen de ARNt supresor mediante la ligación de pares de oligonucleótidos hibridados. En este y otros procedimientos relacionados, los oligonucleótidos monocatenarios complementarios que comprenden la secuencia de ADN deseada completa se hibridan en pares para producir fragmentos bicatenarios, que se alinean en el orden correcto en virtud de salientes monocatenarios complementarios (Stabinsky, patente de Estados Unidos 4.652.639). Los fragmentos resultantes se ligan posteriormente secuencialmente o en una reacción en un tubo (Jayaraman, patente de Estados Unidos 5.132.215) enzimática o químicamente. Después de la purificación y/o clonación estos fragmentos génicos pueden unirse entre sí para formar construcciones de ADN más grandes. En la denominada “síntesis de casete”, cada par de oligonucleótidos hibridados se clona de forma separada en un vector plasmídico antes de unir los fragmentos usando endonucleasas de restricción (Richards y col., patente de Estados Unidos 5.093.251).
Como alternativa, las construcciones de ADN pueden ensamblarse a partir de oligonucleótidos parcialmente hibridados, que contienen después de la hibridación huecos monocatenarios que deben llenarse mediante polimerasas de ADN; este procedimiento se denomina comúnmente procedimiento de “llenado de huecos”. De acuerdo con este procedimiento se sintetizan una diversidad de oligonucleótidos parcialmente solapantes, se purifican y posteriormente se hibridan habitualmente en pares o en subgrupos. Después de la síntesis de las cadenas opuestas respectivas usando una ADN polimerasa los fragmentos individuales se ligan entre sí. Los productos de ligación bicatenarios generados de este modo pueden clonarse como fragmentos parciales o amplificarse en una reacción en cadena de la polimerasa (PCR) con cebadores oligonucleotídicos terminales. Sin embargo, este procedimiento está asediado por frecuentes acontecimientos de cebado erróneo y deleciones internas debido a la formación de estructuras secundarias.
Ambos procedimientos son de uso limitado puesto que con una longitud creciente de la molécula de ácido nucleico a sintetizar la probabilidad de que se incorpore uno o varios oligonucleótidos con una secuencia incorrecta en el producto final aumenta. Dichos errores se copian después en la reacción de la ADN polimerasa. Además, también pueden producirse errores de secuencia durante la reacción de PCR.
Se describe una combinación de los procedimientos anteriores en la patente de Estados Unidos 6.472.184 en la que una serie de oligonucleótidos enlazables que representan regiones contiguas en una cadena de la secuencia diana se hibridan con oligonucleótidos no enlazables que son complementarios a los extremos 3' ó 5' de los oligonucleótidos enlazables que van a conectarse. Este procedimiento es relativamente simple y directo pero también se ve asediado por los problemas comunes compartidos por todos los procedimientos que usan oligonucleótidos monocatenarios como componentes básicos: la formación de estructuras secundarias no deseadas y la incorporación de oligonucleótidos n-x, ambos de los cuales conducen a deleciones internas.
Además de estos procedimientos convencionales, existen procedimientos adicionales conocidos en la técnica para la producción de moléculas de ADN sintéticas. La solicitud de patente internacional WO 98/15567 y la patente de Estados Unidos 6.110.668 enseñan un procedimiento dirigido por molde para emparejar oligonucleótidos para producir construcciones de ADN sintético ligando una pluralidad de oligonucleótidos que son al menos parcialmente complementarios al ADN molde monocatenario y se ligan los extremos de dichos oligonucleótidos en el orden correcto en etapas sucesivas de hibridación y desnaturalización. Sin embargo, una condición previa para la aplicación de este procedimiento es la existencia anterior de un ADN molde adecuado excluyendo su uso en síntesis de novo.
La solicitud de patente internacional WO 99/47536 desvela un procedimiento de síntesis génica de fase sólida en la que se ligan secuencialmente oligonucleótidos monocatenarios con una molécula iniciadora inmovilizada en una orientación definida. Una desventaja de este procedimiento es que se requieren muchas etapas para sintetizar genes grandes lo que da como resultado un rendimiento reducido y el enriquecimiento de secuencias defectuosas. Además, este procedimiento es difícil de automatizar lo que es un prerrequisito para una síntesis rápida, normalizada.
La solicitud de patente internacional WO 00/75368 desvela una síntesis en fase sólida combinatoria de ácidos nucleicos usando una biblioteca de oligonucleótidos bicatenarios como componentes básicos normalizados. El uso de componentes básicos normalizados hace innecesario sintetizar un nuevo conjunto de oligonucleótidos para cada nueva síntesis. Estos oligonucleótidos bicatenarios de la biblioteca generalmente comparten una estructura global idéntica y por lo tanto evitan los problemas de síntesis habituales causados por la formación de estructuras secundarias alternativas de los componentes básicos de los oligonucleótidos tales como la introducción de deleciones. En una versión preferida, contienen un bucle terminal, un tallo bicatenario y un saliente corto monocatenario. Existen dos clases diferentes de oligonucleótidos de la biblioteca, que se caracterizan por la presencia de diferentes sitios de reconocimiento para enzimas de restricción de tipo IIS dentro de su secuencia y la presencia o ausencia o el tipo de una modificación interna. Los nucleótidos...
Reivindicaciones:
1. Procedimiento para la fabricación de una molécula de ácido nucleico, que comprende
las etapas de a) proporcionar un primer oligonucleótido al menos parcialmente bicatenario que comprende un sitio de reconocimiento para una primera enzima de restricción de tipo IIS que corta fuera de su sitio de reconocimiento y comprendiendo dicho oligonucleótido un saliente monocatenario; b) proporcionar un segundo oligonucleótido al menos parcialmente bicatenario que comprende una modificación que permite al oligonucleótido acoplarse a una superficie, por lo cual el oligonucleótido comprende adicionalmente un sitio de reconocimiento para una segunda enzima de restricción de tipo IIS que corta fuera de su sitio de reconocimiento y comprendiendo dicho segundo oligonucleótido un saliente monocatenario; c) ligar el primer y el segundo oligonucleótidos mediante sus salientes generando un primer producto de ligación; d) inmovilizar el primer producto de ligación en una superficie mediante la modificación proporcionada por el segundo oligonucleótido; e) cortar el primer producto de ligación inmovilizado con la segunda enzima de restricción de tipo IIS liberando de este modo un primer oligonucleótido elongado que tiene un saliente y un segundo oligonucleótido acortado, que permanece unido a la superficie; f) proporcionar un oligonucleótido adicional al menos parcialmente bicatenario que tiene una modificación que permite al oligonucleótido adicional acoplarse de forma específica a una superficie, por lo cual el oligonucleótido contiene un sitio de reconocimiento para una segunda o una adicional enzima de restricción de tipo IIS y un saliente monocatenario que es complementario al saliente del primer oligonucleótido elongado; g) ligar el oligonucleótido adicional al menos parcialmente bicatenario con el primer oligonucleótido elongado mediante sus salientes generando un producto de ligación de segundo nivel; h) cortar el producto de ligación de segundo nivel con la segunda o adicional enzima de restricción de tipo IIS generando de este modo un oligonucleótido elongado de segundo nivel que tiene un saliente y un oligonucleótido adicional acortado; i) inmovilizar el oligonucleótido adicional acortado; j) repetir las etapas f) a i) al menos una vez, generando en la etapa g) un producto de ligación de nivel superior, por lo cual en la última repetición el oligonucleótido adicional entrante comprende un sitio de reconocimiento para una enzima de restricción de tipo IIS que produce tras la escisión un saliente monocatenario idéntico en longitud al saliente generado por la primer enzima de restricción de tipo IIS específica para el primer oligonucleótido y las etapas h) e i) se reemplazan con las etapas k) y l); k) inmovilizar el producto de ligación de nivel superior mediante la modificación proporcionada por el oligonucleótido adicional; y l) cortar el producto de ligación de nivel superior con la enzima de restricción de tipo IIS adicional, dejando la parte del ácido nucleico a fabricar unida al primer oligonucleótido, que se libera preferentemente al sobrenadante y más preferentemente permitiendo su transferencia a un nuevo recipiente de reacción.
2. Procedimiento para la fabricación de una molécula de ácido nucleico, que comprende
las etapas de a) proporcionar un primer oligonucleótido al menos parcialmente bicatenario que comprende un sitio de reconocimiento para una primera enzima de restricción de tipo IIS que corta fuera de su sitio de reconocimiento y comprendiendo dicho oligonucleótido un saliente monocatenario; b) proporcionar un segundo oligonucleótido al menos parcialmente bicatenario que comprende una modificación que permite al oligonucleótido acoplarse a una superficie, por lo cual el oligonucleótido comprende adicionalmente un sitio de reconocimiento para una segunda enzima de restricción de tipo IIS que corta fuera de su sitio de reconocimiento y comprendiendo dicho segundo oligonucleótido un saliente monocatenario; c) ligar el primer y el segundo oligonucleótidos mediante sus salientes generando un primer producto de ligación; d) inmovilizar el primer producto de ligación en una superficie mediante la modificación proporcionada por el segundo oligonucleótido; e) cortar el primer producto de ligación inmovilizado con la segunda enzima de restricción de tipo IIS liberando de este modo un primer oligonucleótido elongado que tiene un saliente y un segundo oligonucleótido acortado, que permanece unido a la superficie; f) proporcionar un oligonucleótido adicional al menos parcialmente bicatenario que tiene una modificación que permite al oligonucleótido adicional acoplarse específicamente a una superficie, por lo cual el oligonucleótido contiene un sitio de reconocimiento para una segunda o adicional enzima de restricción de tipo IIS y un saliente monocatenario que es complementario al saliente del primer oligonucleótido elongado; g) ligar el oligonucleótido adicional al menos parcialmente bicatenario con el primer oligonucleótido elongado mediante sus salientes generando un producto de ligación de segundo nivel; h) cortar el producto de ligación de segundo nivel con la segunda o adicional enzima de restricción de tipo IIS generando de este modo un oligonucleótido elongado de segundo nivel que tiene un saliente y un oligonucleótido adicional acortado; i) inmovilizar el oligonucleótido adicional acortado; j) repetir las etapas f) a i) al menos una vez, generando en la etapa g) un producto de ligación de mayor nivel, por lo cual en la última repetición el oligonucleótido adicional entrante comprende un sitio de reconocimiento para una enzima de restricción de tipo IIS que produce tras la escisión un saliente monocatenario idéntico en longitud al saliente generado por la primer enzima de restricción de tipo IIS específica para el primer oligonucleótido y las etapas h) e i) se reemplazan con las etapas k) y l); k) inmovilizar el producto de ligación de nivel superior mediante la modificación proporcionada por el oligonucleótido adicional; y l) cortar el producto de ligación de nivel superior inmovilizado con la enzima de restricción de tipo IIS específica para el primer oligonucleótido, dejando la parte del ácido nucleico a fabricar unida al oligonucleótido adicional, que se inmoviliza en una superficie.
3. El procedimiento de acuerdo con la reivindicación 1 y/o 2, en el que como etapa m) el producto de ligación de nivel superior inmovilizado cortado de la etapa l) de la reivindicación 2 se liga con el producto de ligación de nivel superior cortado de la etapa l) de la reivindicación 1.
4. El procedimiento de acuerdo con la reivindicación 3, en el que el producto de ligación de nivel superior cortado de la etapa l) de la reivindicación 3 se escinde con la segunda enzima de restricción de tipo IIS antes de la etapa de ligación m).
5. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 4, en el que el número de repeticiones en la etapa j) es dos, tres, cuatro, cinco o seis.
6. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 5, en el que el saliente es un saliente 5' o 3'.
7. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 6, en el que el saliente se selecciona del grupo que comprende un saliente de un nucleótido, un saliente de dos nucleótidos, un saliente de tres nucleótidos, un saliente de cuatro nucleótidos y un saliente de cinco nucleótidos.
8. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 7, en el que el oligonucleótido al menos parcialmente bicatenario comprende una región constante y una región variable, por lo cual la región constante contiene el sitio de reconocimiento para una enzima de restricción de tipo IIS y la región variable contiene una secuencia de ácido nucleico que corresponde a una parte de la secuencia de ácido nucleico de la molécula de ácido nucleico a fabricar.
9. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 8, en el que la enzima adicional de restricción de tipo IIS es la segunda enzima de restricción de tipo IIS.
10. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 9, en el que el oligonucleótido elongado se transfiere a un recipiente de reacción diferente.
11. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 10, en el que el oligonucleótido elongado de segundo nivel se transfiere a un recipiente de reacción diferente.
12. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 11, en el que el oligonucleótido elongado de segundo nivel se usa como el oligonucleótido elongado en la etapa g).
13. El procedimiento de acuerdo con cualquiera de las reivindicaciones 1 a 12, en el que la modificación del oligonucleótido elongado y/o del producto de ligación de nivel superior está proporcionado por el oligonucleótido adicional al menos parcialmente bicatenario.
Patentes similares o relacionadas:
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Método para romper un ácido nucleico y añadir un adaptador por medio de transposasa y reactivo, del 1 de Julio de 2020, de MGI Tech Co., Ltd: Un metodo para romper un acido nucleico y anadir un adaptador por medio de una transposasa, que comprende las siguientes etapas: interrumpir […]
Vacuna subunitaria contra Mycoplasma spp., del 1 de Julio de 2020, de Agricultural Technology Research Institute: Una composición para prevenir una infección por Mycoplasma spp., que comprende: un principio activo, que comprende una proteína de PdhA; y un adyuvante […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Etiquetado y evaluación de una secuencia diana, del 13 de Mayo de 2020, de RhoDx, Inc: Un método para modificar un ácido nucleico, que comprende: (a) poner en contacto un ácido nucleico de cadena sencilla con una actividad de transferasa […]
Preparación de bibliotecas de ácido nucleico marcado usando protocolo de adición en un solo tubo, del 13 de Mayo de 2020, de ILLUMINA, INC: Un método para preparar una biblioteca de fragmentos de ácido nucleico marcados que comprende: (a) poner en contacto una célula individual […]
Proteínas de captura de la superficie celular recombinantes, del 13 de Mayo de 2020, de REGENERON PHARMACEUTICALS, INC.: Un método para detectar y aislar células que producen altos niveles de una proteína heterodimérica que tiene una primera subunidad y una segunda subunidad, […]