PRODUCCIÓN DE BIODIESEL A PARTIR DE GLICERINA.
Producción de biodiesel a partir de glicerina.
La presente invención describe cepas bacterianas de la especie B.
subtilis CECT 7968 y 7969, capaces de expresar los genes mutados sintéticos heterólogos: pdc y adhB originarios de Z.mobilis, `tesA originario de E. coli y atfl originario de Acinetobacter sp. ADP1. Adicionalmente, dichas cepas pueden sobre-expresan al menos uno de los genes de los complejos enzimáticos ACC (acetil-CoA carboxilasa) y acil CoA sintetasa. Mediante dichas cepas se produce un incremento en la producción de biocombustible, preferentemente biodiesel a partir de glicerina como fuente de carbono. Además, la presente invención describe el uso de dichas cepas bacterianas para la producción de dicho biocombustible, biodiesel, a partir de glicerina, así como un procedimiento de síntesis de biocombustible, preferentemente biodiesel, mediante la utilización de las cepas descritas en la presente invención y el biocombustible propiamente obtenido.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201131400.
Solicitante: IDEN BIOTECHNOLOGY, S.L.
Nacionalidad solicitante: España.
Inventor/es: SCHUJMAN,Gustavo Enrique, DE MENDOZA,Diego.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N1/21 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 1/00 Microorganismos, p.ej. protozoos; Composiciones que los contienen (preparaciones de uso médico que contienen material de protozoos, bacterias o virus A61K 35/66, de algas A61K 36/02, de hongos A61K 36/06; preparación de composiciones de uso médico que contienen antígenos o anticuerpos bacterianos, p. ej. vacunas bacterianas, A61K 39/00 ); Procesos de cultivo o conservación de microorganismos, o de composiciones que los contienen; Procesos de preparación o aislamiento de una composición que contiene un microorganismo; Sus medios de cultivo. › modificados por la introducción de material genético extraño.
- C12N9/02 C12N […] › C12N 9/00 Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas. › Oxidorreductasas (1.), p. ej. luciferasa.
- C12N9/10 C12N 9/00 […] › Transferasas (2.) (ribonucleasas C12N 9/22).
- C12N9/16 C12N 9/00 […] › actúan sobre los enlaces éster (3.1).
- C12P7/64 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 7/00 Preparación de compuestos orgánicos que contienen oxígeno. › Grasas; Aceites; Ceras de tipo éster; Acidos grasos superiores, es decir, con una cadena lineal de al menos siete átomos de carbono unida a un grupo carboxilo; Aceites o grasas oxidadas.
Fragmento de la descripción:
Producción de biodiesel a partir de glicerina.
CAMPO DE LA INVENCIÓN
La presente invención divulga un procedimiento para la síntesis de ésteres etílicos de ácidos grasos, también conocidos como biodiesel (FAEE en sus siglas en inglés) a partir de glicerina o glicerol. Por tanto la presente invención puede englobarse dentro del campo de las biorefinerías industriales.
ESTADO DE LA TÉCNICA
La glicerina cruda o glicerol es un subproducto del proceso de fabricación de biodiesel en las biorefinerías industriales. La principal función del biodiesel es su utilización como combustible en sustitución de los combustibles derivados del petróleo, debido a los problemas inherentes que genera el uso de combustibles derivados del petróleo, en cuanto a contaminación, efecto invernadero y calentamiento global, existiendo así la necesidad de una fuente renovable del petróleo que, además, pueda producirse económicamente.
En este sentido, el biodiesel puede ser utilizado en la mayoría de los motores de combustión interna diesel, ya sea en forma pura, lo que se conoce como "biodiesel limpio", o como una mezcla a cualquier concentración con combustible diesel derivado de petróleo. El biodiesel ofrece ventajas frente a los derivados diesel del petróleo, incluyendo, la reducción de emisiones de gases durante su combustión, además de ser capaz de mantener un ciclo de dióxido de carbono equilibrado, ya que se basa en el uso de materiales biológicos renovables, es biodegradable y da mayor seguridad debido a su alto punto de inflamación y baja inflamabilidad. Otra ventaja es el menor desgaste de los motores, gracias a las buenas propiedades de lubricación que presenta.
Uno de los usos a los que se puede dedicar la glicerina que se obtiene como subproducto en la producción de biodiesel es la propia obtención de biodiesel mediante un proceso alternativo que comprende utilizar dicha glicerina (glicerol) como sustrato en procesos fermentativos bacterianos.
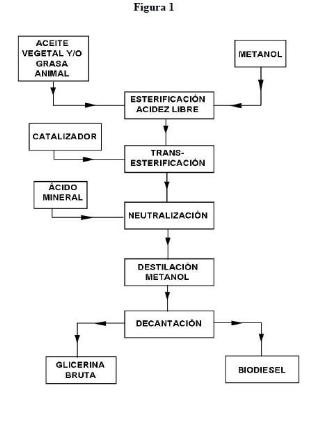
El biodiesel se obtiene principalmente, a partir de aceites vegetales o grasas animales, mediante procesos industriales de esterificación y transesterificación (Figura 1) (Karmakar A., et al 2010) . La glicerina formada en el proceso de obtención de biodiesel industrial, en un desecho industrial del que hay que deshacerse, debido al bajo coste del mismo en los últimos años.
Actualmente, una de las preocupaciones más importantes en el campo de las biorefinerías industriales, es como dar salida a este subproducto que está causando un gran impacto negativo a nivel económico y medioambiental en dichas empresas. Una planta de biodiesel de origen vegetal que genere alrededor de 250.000 toneladas de biodiesel/año, produce un 10% de glicerina, es decir, unas 25.000 toneladas/año de glicerina. Deshacerse de dicho subproducto implica unos enormes gastos, como por ejemplo, el transporte del mismo para, eventualmente, proceder a su posterior comercialización. Ante esta situación, el desarrollo de procesos biotecnológicos encaminados a la conversión de la glicerina cruda en productos de alto valor añadido, es una “necesidad”. Estos procesos biotecnológicos podrían incorporarse en plantas de biodiesel ya existentes, donde se procesaría in situ la glicerina generada como subproducto de la fabricación de dicho biodiesel, o dar lugar a biorefinerías que integren múltiples procesos, en un concepto análogo al utilizado para las refinerías industriales, las cuáles producen múltiples combustibles y productos a partir del petróleo.
Desde hace años se han venido utilizado diferentes cepas bacterianas, principalmente de la especie Escherichia coli, para la producción de FAEE, lográndose avances significativos en el entendimiento de la vía de la síntesis de lípidos y su regulación en bacterias. Dicha regulación se ha presentado, en el pasado, como el principal factor limitante para la utilización de bacterias o plantas como "fábricas" de aceites o combustibles. Vaneechoutte M. et al, describen que para producir FAEE a partir de E. coli, es necesaria la esterificación del etanol producido en el metabolismo de la síntesis de dichos FAEE con los restos acilo unidos a la Coenzima A, mediante la sobre-expresión de la enzima sintetasa de triacilgliceroles y ceras (Atf1) de Acinetobacter baylyi (Atsumi S, et al. 2008) . Dicha cepa de E. coli transformada, es capaz de producir concentraciones de FAEE de 1.3 g/L con un rendimiento de producción de 0.018 g/ (L/h) en presencia de glucosa y ácido oleico (Atsumi S, et al. 2008) . En este mismo sentido, Kalscheuer R. et al, demostraron que mediante la expresión heteróloga en E. coli de las enzimas piruvato decarboxilasa (Pdc) y alcohol deshidrogenasa (AdhB) de Zymomonas mobilis y la sintetasa inespecífica (Atf1) de Acinetobacter cepa baylyi ADP1, se consigue la producción de FAEE (Kalscheuer R. et al 2008) . Dicha síntesis del etanol se combinó con la posterior esterificación del mismo con los restos de acilo del Coenzima A de ácidos grasos cuando las bacterias se cultivaban en condiciones aeróbicas, en presencia de glucosa y ácido oleico.
La solicitud de patente internacional WO2011/038134, divulga también la producción de biodiesel a partir de organismos bacterianos modificados genéticamente. Dicha solicitud internacional muestra ejemplos de bacterias E. coli capaces de producir biodiesel gracias a la expresión o sobreexpresión de un gen que codifica para una tioesterasa, de un gen que codifica para una Acil-CoA sintetasa y de un gen que codifica para una éster-sintasa. El biocombustible generado por las bacterias descritas en dicha solicitud internacional está compuesto principalmente por ácidos grasos lineales de número par de carbonos, en promedio, el 50% de los mismos, con una insaturación.
Otros autores también han divulgado la producción de biocombustibles a partir de la conversión de glicerol en etanol por medio de diferentes cepas de E.coli modificadas genéticamente con el propósito de producir dichos biocombustibles (Shams Yazdani S. et al 2008; da Silva GP. et al. 2009; Choi WJ. 2008) . Otras especies bacterianas, también han sido modificadas genéticamente para la producción de dicho biocombustible mediante la transformación de glicerol en etanol. Por ejemplo, cepas mutantes de Klebsiella pneumoniae, obtenidas mediante irradiación y transformadas con los genes pdc y adhII obtenidos de Z. mobilis y que sobre-expresan las enzimas pdc y adh, respectivamente, muestran una producción de etanol a partir de glicerol de 25 g/L (Oh BR. et al, 2011) . Hasta la fecha, las principales cepas bacterianas utilizadas para la síntesis de FAEE a partir de glicerol han sido cepas patógenas (o potencialmente patógenas) , como por ejemplo, Z. mobilis y E. coli, como se ha comentado previamente.
La finalidad de la obtención de dichas cepas bacterianas, capaces de sintetizar FAEE a partir de glicerol, es que dichas cepas sean capaces de secretar al exterior los FAEE sintetizados. En este sentido, las bacterias remanentes que queden en el medio, al ser tóxicas, pueden constituir un problema de manejo de residuos tóxicos, ya que, por ejemplo, E. coli es la responsable de la contaminación entérica de aguas de consumo (coliformes) .
La presente invención describe un método de producción de biodiesel (FAEE) a partir de glicerina como fuente de carbono, mediante la transformación de bacterias no patógenas, del género Bacillus, con diferentes genes mutados sintéticos heterólogos, implicados en la síntesis de dichos FAEE. Las bacterias del género Bacillus están aprobadas para consumo en humanos y son utilizadas a gran escala en la industria como biofactorías para la producción de diferentes enzimas y proteínas (Westers et al. 2004) . Más específicamente, la invención divulga bacterias de la especie Bacillus subtilis transformadas con genes mutados sintéticos heterólogos, diseñados específicamente para aumentar su expresión en las bacterias de dicha especie. En este sentido, se han aislado los genes de las enzimas piruvato descarboxilasa (pdc) (SEQ ID NO: 1) y alcohol deshidrogenasa (adhB) (SEQ ID NO: 3) pertenecientes a Zymomonas mobilis, además de la porción activa de la enzima tioesterasa A (’tesA) (SEQ ID NO: 5) de Escherichia coli y la sintetasa de triacilgliceroles y ceras (atf1) (SEQ ID NO: 7) de Acinetobacter sp. ADP1. A partir de las secuencias de dichos genes, se han obtenido los correspondientes genes mutados sintéticos de las enzimas pdc (SEQ ID NO: 2) , adhB (SEQ ID NO: 4) , ’tesA (SEQ ID NO: 6) y atf1 (SEQ ID NO: 8) , con los que se ha transformado a las bacterias B. subtilis descritas en la presente invención. Además, para incrementar el rendimiento en la producción de biodiesel mediante las bacterias descritas...
Reivindicaciones:
1. Construcción génica que comprende al menos los genes caracterizados por las SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6 y SEQ ID NO: 8, junto con al menos un promotor.
2. Construcción génica según la reivindicación 1 caracterizada por que además comprende al menos una de las secuencias nucleotídicas que codifican para los genes seleccionados entre: lcfA, yhfL, yhfT, accD, accA, accB o accC y/o cualquiera de sus combinaciones.
3. Construcción génica según la reivindicación 2 caracterizada por que las secuencias nucleotídicas que codifican para los genes lcfA, yhfL, yhfT, accD, accA, accB o accC pertenecen a la especie bacteriana B. subtilis.
4. Construcción génica según las reivindicaciones 1 a 3 caracterizada por que el promotor es inducible o constitutivo.
5. Construcción génica según la reivindicación 4 caracterizado por que el promotor inducible es el promotor Pspac o Pspachy.
6. Construcción génica según la reivindicación 4 caracterizada por que el promotor constitutivo es el promotor Pacp.
7. Construcción génica según cualquiera de las reivindicaciones anteriores caracterizada por que se selecciona de entre cualquiera de las siguientes: pNAKA62, pNAKA52, pNAKA53 y/o combinaciones de las mismas.
8. Vector de expresión que comprende las secuencias SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6 o SEQ ID NO: 8.
9. Vector de expresión según la reivindicación 8 caracterizado por que además comprende al menos una de las secuencias nucleotídicas que codifican para los genes seleccionados entre: lcfA, yhfL, yhfT, accD, accA, accB o accC y/o cualquiera de sus combinaciones.
10. Vector de expresión según la reivindicación 9 caracterizado por que las secuencias nucleotídicas que codifican para los genes lcfA, yhfL, yhfT, accD, accA, accB o accC pertenecen a la especie bacteriana B. subtilis.
11. Vector de expresión según cualquiera de las reivindicaciones 8 a 10 caracterizado por comprender al menos uno de las construcciones génicas según las reivindicaciones 1 a 7.
12. Vector de expresión según la reivindicación 11 que consiste en el plásmido pNAKA62.
13. Vector de expresión según la reivindicación 11 que consiste en los plásmidos pNAKA62 y pNAKA52.
14. Vector de expresión según la reivindicación 11 que consiste en los plásmidos pNAKA62 y pNAKA53.
15. Célula transformada con cualquiera de los vectores de expresión de las reivindicaciones 8 a 14.
16. Célula según la reivindicación 15 caracterizada por ser una célula bacteriana.
17. Célula según la reivindicación 16 caracterizada por ser una célula bacteriana del género Bacillus.
18. Célula según la reivindicación 17 caracterizada por ser de de la especie B. subtilis.
19. Célula según cualquiera de las reivindicaciones 15 a 18 caracterizada por que se selecciona de entre cualquiera de las siguientes: CECT 7968 o CECT 7969.
20. Uso de las células de las reivindicaciones 15 a 19 para la producción de biocombustible.
21. Uso según la reivindicación 20 caracterizado por que el biocombustible es biodiesel.
22. Método de producción de biocombustible que comprende cultivar las células de las reivindicaciones 15 a 19 en presencia de una fuente de carbono, en condiciones adecuadas.
23. Método según la reivindicación 22 donde la fuente de carbono es glicerina.
24. Método según la reivindicación 23 caracterizado por que la glicerina utilizada como fuente de carbono es un subproducto.
25. Método según las reivindicaciones 22 a 24 que además comprende el aislamiento del biocombustible producido.
26. Método según la reivindicación 25 donde el aislamiento se realiza mediante procedimientos de extracción orgánica.
27. Método según las reivindicaciones 22 a 26 caracterizado por que el biocombustible obtenido está compuesto por ácidos grasos ramificados y saturados.
28. Método según la reivindicación 27 caracterizado por que los ácidos grasos son preferentemente: isoC15:0, n-c15:0, anteiso-C15:0, iso-C16:0, n-C16:0, iso-C17:0, n-c17:0, anteiso-C17:0 y n-C18:0.
29. Método según las reivindicaciones 22 a 28 caracterizado por que el biocombustible es biodiesel.
30. Método según cualquiera de las reivindicaciones 22 a 29 caracterizado por que además comprende la adición de al menos un aditivo.
31. Composición de biocombustible que comprende etil-ésteres de ácidos grasos ramificados y saturados.
32. Composición según la reivindicación 31 caracterizada por que los etil-ésteres de ácidos grasos son: iso-C15:0, n-c15:0, anteiso-C15:0, iso-C16:0, n-C16:0, iso-C17:0, n-c17:0, anteiso-C17:0 y n-C18:0.
33. Composición según las reivindicaciones 31 a 32 caracterizada por que el biocombustible es biodiesel.
34. Composición según las reivindicaciones 31 a 33 caracterizada por que además comprende al menos un aditivo.
Figura 1
Figura 2
LISTADO DE SECUENCIAS
<110> Iden Biotechnology S.L.
<120> Producción de biodiesel a partir de glicerina <130> P-03963
<160> 16
<170> PatentIn version 3.5
<210> 1
<211> 1707
<212> DNA
<213> Zymomonas mobilis <400> 1 atgagttata ctgtcggtac ctatttagcg gagcggcttg tccagattgg tctcaagcat 60 cacttcgcag tcgcgggcga ctacaacctc gtccttcttg acaacctgct tttgaacaaa 120 aacatggagc aggtttattg ctgtaacgaa ctgaactgcg gtttcagtgc agaaggttat 180 gctcgtgcca aaggcgcagc agcagccgtc gttacctaca gcgtcggtgc gctttccgca 240 tttgatgcta tcggtggcgc ctatgcagaa aaccttccgg ttatcctgat ctccggtgct 300 ccgaacaaca atgaccacgc tgctggtcac gtgttgcatc acgctcttgg caaaaccgac 360 tatcactatc agttggaaat ggccaagaac atcacggccg ccgctgaagc gatttatacc 420 ccggaagaag ctccggctaa aatcgatcac gtgattaaaa ctgctcttcg tgagaagaag 480 ccggtttatc tcgaaatcgc ttgcaacatt gcttccatgc cctgcgccgc tcctggaccg 540 gcaagcgcat tgttcaatga cgaagccagc gacgaagctt ctttgaatgc agcggttgaa 600 gaaaccctga aattcatcgc caaccgcgac aaagttgccg tcctcgtcgg cagcaagctg 660 cgcgcagctg gtgctgaaga agctgctgtc aaatttgctg atgctcttgg tggcgcagtt 720 gctaccatgg ctgctgcaaa aagcttcttc ccagaagaaa acccgcatta catcggtacc 780 tcatggggtg aagtcagcta tccgggcgtt gaaaagacga tgaaagaagc cgatgcggtt 840 atcgctctgg ctcctgtctt taacgactac tccaccactg gttggacgga tattcctgat 900 cctaagaaac tggttctcgc tgaaccgcgt tctgtcgtcg ttaacggcat tcgcttcccc 960 agcgtccatc tgaaagacta tctgacccgt ttggctcaga aagtttccaa gaaaaccggt 1020 gctttggact tcttcaaatc cctcaatgca ggtgaactga agaaagccgc tccggctgat 1080
ccgagtgctc cgttggtcaa cgcagaaatc gcccgtcagg tcgaagctct tctgaccccg 1140 aacacgacgg ttattgctga aaccggtgac tcttggttca atgctcagcg catgaagctc 1200 ccgaacggtg ctcgcgttga atatgaaatg cagtggggtc acattggttg gtccgttcct 1260 gccgccttcg gttatgccgt cggtgctccg gaacgtcgca acatcctcat ggttggtgat 1320 ggttccttcc agctgacggc tcaggaagtc gctcagatgg ttcgcctgaa actgccggtt 1380 atcatcttct tgatcaataa ctatggttac accatcgaag ttatgatcca tgatggtccg 1440 tacaacaaca tcaagaactg ggattatgcc ggtctgatgg aagtgttcaa cggtaacggt 1500 ggttatgaca gcggtgctgg taaaggcctg aaggctaaaa ccggtggcga actggcagaa 1560 gctatcaagg ttgctctggc aaacaccgac ggcccaaccc tgatcgaatg cttcatcggt 1620 cgtgaagact gcactgaaga attggtcaaa tggggtaagc gcgttgctgc cgccaacagc 1680 cgtaagcctg ttaacaagct cctctag 1707
<210> 2
<211> 1707
<212> DNA
<213> Artificial Sequence <220>
<223> secuencia mutada sintética del gen pdc
<400> 2 atgtcttata ctgttggtac ttatttagct gaacgtcttg ttcaaattgg tcttaaacat 60 cacttcgcag ttgctggtga ctacaacctt gttcttcttg acaaccttct tcttaacaaa 120 aacatggaac aagtttattg ctgtaacgaa cttaactgcg gtttctctgc agaaggttat 180 gctcgtgcta aaggtgcagc agcagctgtt gttacttact ctgttggtgc tctttctgca 240 ttcgatgcta tcggtggtgc ttatgcagaa aaccttcctg ttatccttat ctctggtgct 300 cctaacaaca acgaccacgc tgctggtcac gttcttcatc acgctcttgg taaaactgac 360 tatcactatc aacttgaaat ggctaaaaac atcactgctg ctgctgaagc tatttatact 420 cctgaagaag ctcctgctaa aatcgatcac gttattaaaa ctgctcttcg tgaaaaaaaa 480 cctgtttatc ttgaaatcgc ttgcaacatt gcttctatgc cttgcgctgc tcctggacct 540 gcatctgcac ttttcaacga cgaagcatct gacgaagcat ctcttaacgc agctgttgaa 600 gaaactctta aattcatcgc taaccgcgac aaagttgctg ttcttgttgg ttctaaactt 660
cgcgcagctg gtgctgaaga agctgctgtt aaattcgctg atgctcttgg tggtgcagtt 720 gctactatgg ctgctgcaaa atctttcttc ccagaagaaa accctcatta catcggtact 780 tcttggggtg aagtttctta tcctggtgtt gaaaaaacta tgaaagaagc tgatgctgtt 840 atcgctcttg ctcctgtttt caacgactac tctactactg gttggactga tattcctgat 900 cctaaaaaac ttgttcttgc tgaacctcgt tctgttgttg ttaacggtat tcgcttccct 960 tctgttcatc ttaaagacta tcttactcgt cttgctcaaa aagtttctaa aaaaactggt 1020 gctcttgact tcttcaaatc tcttaacgca ggtgaactta aaaaagctgc tcctgctgat 1080 ccttctgctc ctcttgttaa cgcagaaatc gctcgtcaag ttgaagctct tcttactcct 1140 aacactactg ttattgctga aactggtgac tcttggttca acgctcaacg catgaaactt 1200 cctaacggtg ctcgcgttga atatgaaatg caatggggtc acattggttg gtctgttcct 1260 gctgctttcg gttatgctgt tggtgctcct gaacgtcgca acatccttat ggttggtgat 1320 ggttctttcc aacttactgc tcaagaagtt gctcaaatgg ttcgccttaa acttcctgtt 1380 atcatcttcc ttatcaacaa ctatggttac actatcgaag ttatgatcca tgatggtcct 1440 tacaacaaca tcaaaaactg ggattatgct ggtcttatgg aagttttcaa cggtaacggt 1500 ggttatgact ctggtgctgg taaaggtctt aaagctaaaa ctggtggtga acttgcagaa 1560 gctatcaaag ttgctcttgc aaacactgac ggtccaactc ttatcgaatg cttcatcggt 1620 cgtgaagact gcactgaaga acttgttaaa tggggtaaac gcgttgctgc tgctaactct 1680 cgtaaacctg ttaacaaact tctttag 1707
<210> 3
<211> 1152
<212> DNA
<213> Zymomonas mobilis <400> 3 atggcttctt caacttttta tattcctttc gtcaacgaaa tgggcgaagg ttcgcttgaa 60 aaagcaatca aggatcttaa cggcagcggc tttaaaaatg cgctgatcgt ttctgatgct 120 ttcatgaaca aatccggtgt tgtgaagcag gttgctgacc tgttgaaagc acagggtatt 180 aattctgctg tttatgatgg cgttatgccg aacccgactg ttaccgcagt tctggaaggc 240 cttaagatcc tgaaggataa caattcagac ttcgtcatct ccctcggtgg tggttctccc 300
catgactgcg ccaaagccat cgctctggtc gcaaccaatg gtggtgaagt caaagactac 360 gaaggtatcg acaaatctaa gaaacctgcc ctgcctttga tgtcaatcaa cacgacggct 420 ggtacggctt ctgaaatgac gcgtttctgc atcatcactg atgaagtccg tcacgttaag 480 atggccattg ttgaccgtca cgttaccccg atggtttccg tcaacgatcc tctgttgatg 540 gttggtatgc caaaaggcct gaccgccgcc accggtatgg atgctctgac ccacgcattt 600 gaagcttatt cttcaacggc agctactccg atcaccgatg cttgcgcctt gaaggctgcg 660 tccatgatcg ctaagaatct gaagaccgct tgcgacaacg gtaaggatat gccagctcgt 720 gaagctatgg cttatgccca attcctcgct ggtatggcct tcaacaacgc ttcgcttggt 780 tatgtccatg ctatggctca ccagttgggc ggctactaca acctgccgca tggtgtctgc 840 aacgctgttc tgcttccgca tgttctggct tataacgcct ctgtcgttgc tggtcgtctg 900 aaagacgttg gtgttgctat gggtctcgat atcgccaatc tcggtgataa agaaggcgca 960 gaagccacca ttcaggctgt tcgcgatctg gctgcttcca ttggtattcc agcaaatctg 1020 accgagctgg gtgctaagaa agaagatgtg ccgcttcttg ctgaccacgc tctgaaagat 1080 gcttgtgctc tgaccaaccc gcgtcagggt gatcagaaag aagttgaaga actcttcctg 1140 agcgctttct aa 1152
<210> 4
<211> 1152
<212> DNA
<213> Artificial Sequence <220>
<223> Secuencia mutada sintética del gen adhB
<400> 4 atggcttctt ctactttcta tattcctttc gttaacgaaa tgggtgaagg ttctcttgaa 60 aaagcaatca aagaccttaa cggttctggt ttcaaaaacg ctcttatcgt ttctgatgct 120 ttcatgaaca aatctggtgt tgttaaacaa gttgctgacc ttcttaaagc acaaggtatt 180 aactctgctg tttatgatgg tgttatgcct aaccctactg ttactgcagt tcttgaaggt 240 cttaaaatcc ttaaagataa caactctgac ttcgttatct ctcttggtgg tggttctcct 300 catgactgcg ctaaagctat cgctcttgtt gcaactaacg gtggtgaagt taaagactac 360 gaaggtatcg acaaatctaa aaaacctgct cttcctctta tgtctatcaa cactactgct 420
ggtactgctt ctgaaatgac tcgtttctgc atcatcactg atgaagttcg tcacgttaaa 480 atggctattg ttgaccgtca cgttactcct atggtttctg ttaacgatcc tcttcttatg 540 gttggtatgc caaaaggtct tactgctgct actggtatgg atgctcttac tcacgcattc 600 gaagcatatt cttctactgc agctactcct atcactgatg cttgcgctct taaagctgct 660 tctatgatcg ctaaaaacct taaaactgct tgcgacaacg gtaaagatat gccagctcgt 720 gaagctatgg cttatgctca attccttgct ggtatggctt tcaacaacgc ttctcttggt 780 tatgttcatg ctatggctca ccaacttggt ggttactaca accttcctca tggtgtttgc 840 aacgctgttc ttcttcctca tgttcttgct tataacgctt ctgttgttgc tggtcgtctt 900 aaagacgttg gtgttgctat gggtcttgat atcgctaacc ttggtgataa agaaggtgca 960 gaagctacta ttcaagctgt tcgcgatctt gctgcttcta ttggtattcc agcaaacctt 1020 actgaacttg gtgctaaaaa agaagatgtt cctcttcttg ctgaccacgc tcttaaagat 1080 gcttgtgctc ttactaaccc tcgtcaaggt gatcaaaaag aagttgaaga acttttcctt 1140 tctgctttct aa 1152
<210> 5
<211> 555
<212> DNA
<213> Escherichia coli <400> 5 atggcagcgg acacgttatt gattctgggt gatagcctga gcgccgggta tcgaatgtct 60 gccagcgcgg cctggcctgc cttgttgaat gataagtggc agagtaaaac gtcggtagtt 120 aatgccagca tcagcggcga cacctcgcaa caaggactgg cgcgccttcc ggctctgctg 180 aaacagcatc agccgcgttg ggtgctggtt gaactgggcg gcaatgacgg tttgcgtggt 240 tttcagccac agcaaaccga gcaaacgctg cgccagattt tgcaggatgt caaagccgcc 300 aacgctgaac cattgttaat gcaaatacgt ctgcctgcaa actatggtcg ccgttataat 360 gaagccttta gcgccattta ccccaaactc gccaaagagt ttgatgttcc gctgctgccc 420 ttttttatgg aagaggtcta cctcaagcca caatggatgc aggatgacgg tattcatccc 480 aaccgcgacg cccagccgtt tattgccgac tggatggcga agcagttgca gcctttagta 540 aatcatgact cataa 555 <210> 6
<211> 555
<212> DNA
<213> Artificial Sequence <220>
<223> Secuencia mutada sintética del gen ´tesA
<400> 6 atggcagctg acactttact tattcttggt gattctcttt ctgctggtta tcgtatgtct 60 gcttctgctg cttggcctgc tcttcttaac gataaatggc aatctaaaac ttctgtagtt 120 aacgcttcta tctctggtga cacttctcaa caaggacttg ctcgccttcc tgctcttctt 180 aaacaacatc aacctcgttg ggttcttgtt gaacttggtg gtaacgacgg tcttcgtggt 240 ttccaaccac aacaaactga acaaactctt cgccaaattc ttcaagatgt taaagctgct 300 aacgctgaac cacttttaat gcaaatccgt cttcctgcaa actatggtcg ccgttataac 360 gaagcattct ctgctattta ccctaaactt gctaaagaat tcgatgttcc tcttcttcct 420 ttcttcatgg aagaagttta ccttaaacca caatggatgc aagatgacgg tattcatcct 480 aaccgcgacg ctcaaccttt cattgctgac tggatggcta aacaacttca acctttagta 540 aaccatgact cttaa 555
<210> 7
<211> 1377
<212> DNA
<213> Acinetobacter cepa baylyi ADP1
<400> 7 atgcgcccat tacatccgat tgattttata ttcctgtcac tagaaaaaag acaacagcct 60 atgcatgtag gtggtttatt tttgtttcag attcctgata acgccccaga cacctttatt 120 caagatctgg tgaatgatat ccggatatca aaatcaatcc ctgttccacc attcaacaat 180 aaactgaatg ggcttttttg ggatgaagat gaagagtttg atttagatca tcattttcgt 240 catattgcac tgcctcatcc tggtcgtatt cgtgaattgc ttatttatat ttcacaagag 300 cacagtacgc tgctagatcg ggcaaagccc ttgtggacct gcaatattat tgaaggaatt 360 gaaggcaatc gttttgccat gtacttcaaa attcaccatg cgatggtcga tggcgttgct 420 ggtatgcggt taattgaaaa atcactctcc catgatgtaa cagaaaaaag tatcgtgcca 480 ccttggtgtg ttgagggaaa acgtgcaaag cgcttaagag aacctaaaac aggtaaaatt 540
aagaaaatca tgtctggtat taagagtcag cttcaggcga cacccacagt cattcaagag 600 ctttctcaga cagtatttaa agatattgga cgtaatcctg atcatgtttc aagctttcag 660 gcgccttgtt ctattttgaa tcagcgtgtg agctcatcgc gacgttttgc agcacagtct 720 tttgacctag atcgttttcg taatattgcc aaatcgttga atgtgaccat taatgatgtt 780 gtactagcgg tatgttctgg tgcattacgt gcgtatttga tgagtcataa tagtttgcct 840 tcaaaaccat taattgccat ggttccagcc tctattcgca atgacgattc agatgtcagc 900 aaccgtatta cgatgattct ggcaaatttg gcaacccaca aagatgatcc tttacaacgt 960 cttgaaatta tccgccgtag tgttcaaaac tcaaagcaac gcttcaaacg tatgaccagc 1020 gatcagattc taaattatag tgctgtcgta tatggccctg caggactcaa cataatttct 1080 ggcatgatgc caaaacgcca agccttcaat ctggttattt ccaatgtgcc tggcccaaga 1140 gagccacttt actggaatgg tgccaaactt gatgcactct acccagcttc aattgtatta 1200 gacggtcaag cattgaatat tacaatgacc agttatttag ataaacttga agttggtttg 1260 attgcatgcc gtaatgcatt gccaagaatg cagaatttac tgacacattt agaagaagaa 1320 attcaactat ttgaaggcgt aattgcaaag caggaagata ttaaaacagc caattaa 1377
<210> 8
<211> 1377
<212> DNA
<213> Artificial Sequence <220>
<223> Secuencia mutada sintética del gen atf1
<400> 8 atgcgcccat tacatcctat tgatttcatc ttcctttctc ttgaaaaacg tcaacaacct 60 atgcatgtag gtggtttatt ccttttccaa attcctgata acgctccaga cactttcatt 120 caagaccttg ttaacgatat ccgtatctct aaatctatcc ctgttccacc attcaacaac 180 aaacttaacg gtcttttctg ggatgaagat gaagaattcg atttagatca tcatttccgt 240 catattgcac ttcctcatcc tggtcgtatt cgtgaacttc ttatttatat ttctcaagaa 300 cactctactc ttcttgatcg tgcaaaacct ctttggactt gcaacattat tgaaggaatt 360 gaaggtaacc gtttcgctat gtacttcaaa attcaccatg ctatggttga tggtgttgct 420 ggtatgcgtt taattgaaaa atctctttct catgatgtaa cagaaaaatc tatcgttcca 480
ccttggtgtg ttgaaggaaa acgtgcaaaa cgcttacgtg aacctaaaac aggtaaaatt 540 aaaaaaatca tgtctggtat taaatctcaa cttcaagcta cacctacagt tattcaagaa 600 ctttctcaaa cagtattcaa agatattgga cgtaaccctg atcatgtttc ttctttccaa 660 gctccttgtt ctattcttaa ccaacgtgtt tcttcttctc gtcgtttcgc agcacaatct 720 ttcgaccttg atcgtttccg taacattgct aaatctctta acgttactat taacgatgtt 780 gtacttgctg tatgttctgg tgcattacgt gcttatctta tgtctcataa ctctcttcct 840 tctaaaccat taattgctat ggttccagct tctattcgca acgacgattc tgatgtttct 900 aaccgtatta ctatgattct tgcaaacctt gcaactcaca aagatgatcc tttacaacgt 960 cttgaaatta tccgccgttc tgttcaaaac tctaaacaac gcttcaaacg tatgacttct 1020 gatcaaattc ttaactattc tgctgttgta tatggtcctg caggacttaa catcatttct 1080 ggtatgatgc caaaacgcca agcattcaac cttgttattt ctaacgttcc tggtccacgt 1140 gaaccacttt actggaacgg tgctaaactt gatgcacttt acccagcttc tattgtatta 1200 gacggtcaag cacttaacat tacaatgact tcttatttag ataaacttga agttggtctt 1260 attgcatgtc gtaacgcact tccacgtatg caaaacttac ttacacattt agaagaagaa 1320 attcaacttt tcgaaggtgt aattgcaaaa caagaagata ttaaaacagc taactaa 1377
<210> 9
<211> 37
<212> DNA
<213> Artificial Sequence <220>
<223> Cebador directo del gen LcfA
<400> 9 ggagcttcta gatctaaagg ggaggtttta tgcagtc 37
<210> 10
<211> 36
<212> DNA
<213> Artificial Sequence <220>
<223> Cebador inverso del gen LcfA
<400> 10 ttgcttgcat gctcatggat ccttcctata gaaatg 36
<210> 11 <211> 36 <212> DNA <213> Artificial Sequence
<220> <223> Cebador directo del gen yhfL
<400> 11 ggcttcacta atctagatct attcattcac ttaagg 36
<210> 12 <211> 35 <212> DNA <213> Artificial Sequence
<220> <223> Cebador inverso del gen yhfL
<400> 12 aacgaagtcg acggatccgc tttttcattt tattg 35
<210> 13 <211> 33 <212> DNA <213> Artificial Sequence
<220> <223> Cebador directo del operon accDA
<400> 13 agcgagtcta gatgttgcgg aaggcattat gac 33
<210> 14 <211> 38 <212> DNA <213> Artificial Sequence
<220> <223> Cebador inverso del operón accDA
<400> 14 aaaagcatgc cgtcgactta gtttaccccg atatattg 38
<210> 15 <211> 37 <212> DNA <213> Artificial Sequence
<220>
<223> Cebador directo del operón accBC
<400> 15 tataaatcta gaaatgtcga ccataggagt gcgattg
<210> 16
<211> 32
<212> DNA
<213> Artificial Sequence <220>
<223> Cebador inverso del operón accBC
<400> 16 ttcatcgcat gcacctccgt aaaaattatg ag 32
Patentes similares o relacionadas:
Composiciones útiles en el tratamiento de la deficiencia de ornitina transcarbamilasa (OTC), del 8 de Julio de 2020, de THE TRUSTEES OF THE UNIVERSITY OF PENNSYLVANIA: Un vector vírico recombinante que comprende una secuencia de ácido nucleico que codifica la proteína ornitina transcarbamilasa humana (hOTC) y secuencias […]
Microorganismo con productividad de l-lisina aumentada y procedimiento para producir l-lisina utilizando el mismo, del 27 de Mayo de 2020, de CJ CHEILJEDANG CORPORATION: Una subunidad beta prima (subunidad-β') mutante de la ARN polimerasa, en la que la subunidad beta prima (subunidad-β') mutante de la ARN polimerasa tiene […]
Reducción de sacarosa y generación de fibra insoluble en zumos, del 27 de Mayo de 2020, de DANISCO US INC: Un método para producir una bebida baja en calorías y alta en fibras insolubles comprendiendo; tratamiento de una bebida que contiene sacarosa con una glucosiltransferasa para […]
Una célula acetogénica genéticamente modificada, del 13 de Mayo de 2020, de Evonik Operations GmbH: Una célula microbiana acetogénica que es capaz de producir al menos un alcohol superior a partir de una fuente de carbono, en donde la célula microbiana acetogénica está genéticamente […]
Construcción de nuevas variantes de dextransacarasa DSR-S por ingeniería genética, del 6 de Mayo de 2020, de CENTRE NATIONAL DE LA RECHERCHE SCIENTIFIQUE: 1. Una dextransacarasa que consiste en una secuencia que tiene el 90 %, el 95 % o el 98 % de similitud de secuencia con una secuencia de aminoácidos seleccionada del fragmento […]
Cepas bacterianas que expresan genes de metilasa y sus usos, del 22 de Abril de 2020, de LOMA LINDA UNIVERSITY: Una bacteria aislada para usar en la producción de ADN plasmídico metilado, en donde la bacteria comprende un polinucleótido exógeno que codifica una CpG metilasa […]
Construcción de nuevas variantes de dextransacarasa DSR-S por ingeniería genética, del 22 de Abril de 2020, de CENTRE NATIONAL DE LA RECHERCHE SCIENTIFIQUE: Una secuencia de nucleótidos que consiste en una secuencia de nucleótidos como se define en SEQ ID NO: 1 o la secuencia complementaria a la secuencia como […]
Fábrica de células bacterianas modificadas genéticamente para la producción de tiamina, del 22 de Abril de 2020, de Biosyntia ApS: Bacteria modificada genéticamente para la producción de tiamina no fosforilada; en la que dicha bacteria se caracteriza por tener transgenes […]